+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3502 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural reorganization of the chromatin remodeling enzyme Chd1 upon engagement with nucleosomes | |||||||||
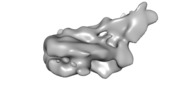
 マップデータ マップデータ | Structure of S.cerevisiae chromatin remodeling enzyme Chd1 bound to Nucleosome. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) / Saccharomyces cerevisiae (パン酵母) /  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 15.0 Å クライオ電子顕微鏡法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Sundaramoorthy R / Owen-Hughes T | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: Structural reorganization of the chromatin remodeling enzyme Chd1 upon engagement with nucleosomes. 著者: Ramasubramanian Sundaramoorthy / Amanda L Hughes / Vijender Singh / Nicola Wiechens / Daniel P Ryan / Hassane El-Mkami / Maxim Petoukhov / Dmitri I Svergun / Barbara Treutlein / Salina Quack ...著者: Ramasubramanian Sundaramoorthy / Amanda L Hughes / Vijender Singh / Nicola Wiechens / Daniel P Ryan / Hassane El-Mkami / Maxim Petoukhov / Dmitri I Svergun / Barbara Treutlein / Salina Quack / Monika Fischer / Jens Michaelis / Bettina Böttcher / David G Norman / Tom Owen-Hughes /   要旨: The yeast Chd1 protein acts to position nucleosomes across genomes. Here, we model the structure of the Chd1 protein in solution and when bound to nucleosomes. In the apo state, the DNA-binding ...The yeast Chd1 protein acts to position nucleosomes across genomes. Here, we model the structure of the Chd1 protein in solution and when bound to nucleosomes. In the apo state, the DNA-binding domain contacts the edge of the nucleosome while in the presence of the non-hydrolyzable ATP analog, ADP-beryllium fluoride, we observe additional interactions between the ATPase domain and the adjacent DNA gyre 1.5 helical turns from the dyad axis of symmetry. Binding in this conformation involves unravelling the outer turn of nucleosomal DNA and requires substantial reorientation of the DNA-binding domain with respect to the ATPase domains. The orientation of the DNA-binding domain is mediated by sequences in the N-terminus and mutations to this part of the protein have positive and negative effects on Chd1 activity. These observations indicate that the unfavorable alignment of C-terminal DNA-binding region in solution contributes to an auto-inhibited state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3502.map.gz emd_3502.map.gz | 55.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3502-v30.xml emd-3502-v30.xml emd-3502.xml emd-3502.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3502_fsc.xml emd_3502_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3502.png emd_3502.png | 54 KB | ||
| その他 |  emd_3502_half_map_1.map.gz emd_3502_half_map_1.map.gz emd_3502_half_map_2.map.gz emd_3502_half_map_2.map.gz | 46.2 MB 46.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3502 http://ftp.pdbj.org/pub/emdb/structures/EMD-3502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3502 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3502.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3502.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of S.cerevisiae chromatin remodeling enzyme Chd1 bound to Nucleosome. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map1 of the reconstruction
| ファイル | emd_3502_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map1 of the reconstruction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
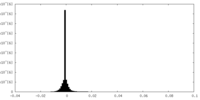
| 密度ヒストグラム |
-ハーフマップ: Half map2 of the reconstruction
| ファイル | emd_3502_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map2 of the reconstruction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Chd1-Nucleosome complex
| 全体 | 名称: Chd1-Nucleosome complex |
|---|---|
| 要素 |
|
-超分子 #1: Chd1-Nucleosome complex
| 超分子 | 名称: Chd1-Nucleosome complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: S.cerevisiae chromatin remodelling enzyme in complex with Nucleosome |
|---|---|
| 分子量 | 理論値: 400 KDa |
-超分子 #2: chd1
| 超分子 | 名称: chd1 / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #3: nucleosome
| 超分子 | 名称: nucleosome / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Chromodomain-Helicase-DNA containing protein
| 分子 | 名称: Chromodomain-Helicase-DNA containing protein / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 配列 | 文字列: MAAKDISTEV LQNPELYGLR RSHRAAAHQQ NYFNDSDDED DEDNIKQSRR KRMTTIEDDE DEFEDEEGE EDSGEDEDEE DFEEDDDYYG SPIKQNRSKP KSRTKSKSKS KPKSQSEKQS T VKIPTRFS NRQNKTVNYN IDYSDDDLLE SEDDYGSEEA LSEENVHEAS ...文字列: MAAKDISTEV LQNPELYGLR RSHRAAAHQQ NYFNDSDDED DEDNIKQSRR KRMTTIEDDE DEFEDEEGE EDSGEDEDEE DFEEDDDYYG SPIKQNRSKP KSRTKSKSKS KPKSQSEKQS T VKIPTRFS NRQNKTVNYN IDYSDDDLLE SEDDYGSEEA LSEENVHEAS ANPQPEDFHG ID IVINHRL KTSLEEGKVL EKTVPDLNNC KENYEFLIKW TDESHLHNTW ETYESIGQVR GLK RLDNYC KQFIIEDQQV RLDPYVTAED IEIMDMERER RLDEFEEFHV PERIIDSQRA SLED GTSQL QYLVKWRRLN YDEATWENAT DIVKLAPEQV KHFQNRENSK ILPQYSSNYT SQRPR FEKL SVQPPFIKGG ELRDFQLTGI NWMAFLWSKG DNGILADEMG LGKTVQTVAF ISWLIF ARR QNGPHIIVVP LSTMPAWLDT FEKWAPDLNC ICYMGNQKSR DTIREYEFYT NPRAKGK KT MKFNVLLTTY EYILKDRAEL GSIKWQFMAV DEAHRLKNAE SSLYESLNSF KVANRMLI T GTPLQNNIKE LAALVNFLMP GRFTIDQEID FENQDEEQEE YIHDLHRRIQ PFILRRLKK DVEKSLPSKT ERILRVELSD VQTEYYKNIL TKNYSALTAG AKGGHFSLLN IMNELKKASN HPYLFDNAE ERVLQKFGDG KMTRENVLRG LIMSSGKMVL LDQLLTRLKK DGHRVLIFSQ M VRMLDILG DYLSIKGINF QRLDGTVPSA QRRISIDHFN SPDSNDFVFL LSTRAGGLGI NL MTADTVV IFDSDWNPQA DLQAMARAHR IGQKNHVMVY RLVSKDTVEE EVLERARKKM ILE YAIISL GVTDGNKYTK KNEPNAGELS AILKFGAGNM FTATDNQKKL EDLNLDDVLN HAED HVTTP DLGESHLGGE EFLKQFEVTD YKADIDWDDI IPEEELKKLQ DEEQKRKDEE YVKEQ LEMM NRRDNALKKI KNSVNGDGTA ANSDSDDDST SRSSRRRARA NDMDSIGESE VRALYK AIL KFGNLKEILD ELIADGTLPV KSFEKYGETY DEMMEAAKDC VHEEEKNRKE ILEKLEK HA TAYRAKLKSG EIKAENQPKD NPLTRLSLKK REKKAVLFNF KGVKSLNAES LLSRVEDL K YLKNLINSNY KDDPLKFSLG NNTPKPVQNW SSNWTKEEDE KLLIGVFKYG YGSWTQIRD DPFLGITDKI FLNEVHNPVA KKSASSSDTT PTPSKKGKGI TGSSKKVPGA IHLGRRVDYL LSFLRGGLN TKSPSADIGS KKLPTGPSKK RQRKPANHSK SMTPEI |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solutions were made fresh | |||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER/RHODIUM / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100.00 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | This sample was monodisperse. One Chd1 enzyme bound to the Nucleosome |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 4.0 µm / 最小 デフォーカス(補正後): 1.8 µm / 倍率(補正後): 59000 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 59000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / デジタル化 - 画像ごとのフレーム数: 4-18 / 撮影したグリッド数: 1 / 実像数: 2560 / 平均露光時間: 1.0 sec. / 平均電子線量: 2.27 e/Å2 詳細: Images are collected in movie mode at 22 images per second |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X