+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30990 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of alpha 7 homo-tetradecamer | ||||||||||||||||||
 マップデータ マップデータ | Symmetric type of alpha 7 homo-tetradecamer | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 regulation of endopeptidase activity / Regulation of ornithine decarboxylase (ODC) / proteasome core complex / Cross-presentation of soluble exogenous antigens (endosomes) / regulation of endopeptidase activity / Regulation of ornithine decarboxylase (ODC) / proteasome core complex / Cross-presentation of soluble exogenous antigens (endosomes) /  Somitogenesis / proteasome core complex, alpha-subunit complex / Somitogenesis / proteasome core complex, alpha-subunit complex /  proteasome complex / Regulation of activated PAK-2p34 by proteasome mediated degradation / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin ... proteasome complex / Regulation of activated PAK-2p34 by proteasome mediated degradation / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin ... regulation of endopeptidase activity / Regulation of ornithine decarboxylase (ODC) / proteasome core complex / Cross-presentation of soluble exogenous antigens (endosomes) / regulation of endopeptidase activity / Regulation of ornithine decarboxylase (ODC) / proteasome core complex / Cross-presentation of soluble exogenous antigens (endosomes) /  Somitogenesis / proteasome core complex, alpha-subunit complex / Somitogenesis / proteasome core complex, alpha-subunit complex /  proteasome complex / Regulation of activated PAK-2p34 by proteasome mediated degradation / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D / AUF1 (hnRNP D0) binds and destabilizes mRNA / TNFR2 non-canonical NF-kB pathway / Assembly of the pre-replicative complex / Vpu mediated degradation of CD4 / Degradation of DVL / proteasomal protein catabolic process / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Dectin-1 mediated noncanonical NF-kB signaling / Hh mutants are degraded by ERAD / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / Degradation of AXIN / Defective CFTR causes cystic fibrosis / Degradation of GLI1 by the proteasome / Activation of NF-kappaB in B cells / Hedgehog ligand biogenesis / Negative regulation of NOTCH4 signaling / G2/M Checkpoints / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Autodegradation of the E3 ubiquitin ligase COP1 / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / Regulation of RUNX3 expression and activity / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / MAPK6/MAPK4 signaling / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / ABC-family proteins mediated transport / Degradation of beta-catenin by the destruction complex / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / CDK-mediated phosphorylation and removal of Cdc6 / CLEC7A (Dectin-1) signaling / SCF(Skp2)-mediated degradation of p27/p21 / Regulation of expression of SLITs and ROBOs / FCERI mediated NF-kB activation / Regulation of PTEN stability and activity / Interleukin-1 signaling / Orc1 removal from chromatin / Regulation of RAS by GAPs / Separation of Sister Chromatids / Regulation of RUNX2 expression and activity / UCH proteinases / The role of GTSE1 in G2/M progression after G2 checkpoint / KEAP1-NFE2L2 pathway / Antigen processing: Ubiquitination & Proteasome degradation / Downstream TCR signaling / proteasome complex / Regulation of activated PAK-2p34 by proteasome mediated degradation / Autodegradation of Cdh1 by Cdh1:APC/C / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D / AUF1 (hnRNP D0) binds and destabilizes mRNA / TNFR2 non-canonical NF-kB pathway / Assembly of the pre-replicative complex / Vpu mediated degradation of CD4 / Degradation of DVL / proteasomal protein catabolic process / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Dectin-1 mediated noncanonical NF-kB signaling / Hh mutants are degraded by ERAD / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / Degradation of AXIN / Defective CFTR causes cystic fibrosis / Degradation of GLI1 by the proteasome / Activation of NF-kappaB in B cells / Hedgehog ligand biogenesis / Negative regulation of NOTCH4 signaling / G2/M Checkpoints / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Autodegradation of the E3 ubiquitin ligase COP1 / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / Regulation of RUNX3 expression and activity / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / MAPK6/MAPK4 signaling / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / ABC-family proteins mediated transport / Degradation of beta-catenin by the destruction complex / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / CDK-mediated phosphorylation and removal of Cdc6 / CLEC7A (Dectin-1) signaling / SCF(Skp2)-mediated degradation of p27/p21 / Regulation of expression of SLITs and ROBOs / FCERI mediated NF-kB activation / Regulation of PTEN stability and activity / Interleukin-1 signaling / Orc1 removal from chromatin / Regulation of RAS by GAPs / Separation of Sister Chromatids / Regulation of RUNX2 expression and activity / UCH proteinases / The role of GTSE1 in G2/M progression after G2 checkpoint / KEAP1-NFE2L2 pathway / Antigen processing: Ubiquitination & Proteasome degradation / Downstream TCR signaling /  Neddylation / RUNX1 regulates transcription of genes involved in differentiation of HSCs / ER-Phagosome pathway / proteasome-mediated ubiquitin-dependent protein catabolic process / Ub-specific processing proteases / Neddylation / RUNX1 regulates transcription of genes involved in differentiation of HSCs / ER-Phagosome pathway / proteasome-mediated ubiquitin-dependent protein catabolic process / Ub-specific processing proteases /  シナプス / シナプス /  ubiquitin protein ligase binding / extracellular exosome / ubiquitin protein ligase binding / extracellular exosome /  核質 / 核質 /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
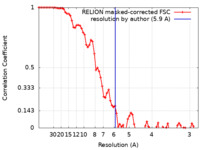
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.9 Å クライオ電子顕微鏡法 / 解像度: 5.9 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Song C / Murata K | ||||||||||||||||||
| 資金援助 |  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2021 ジャーナル: Int J Mol Sci / 年: 2021タイトル: Structural Fluctuations of the Human Proteasome α7 Homo-Tetradecamer Double Ring Imply the Proteasomal α-Ring Assembly Mechanism. 著者: Chihong Song / Tadashi Satoh / Taichiro Sekiguchi / Koichi Kato / Kazuyoshi Murata /  要旨: The 20S proteasome, which is composed of layered α and β heptameric rings, is the core complex of the eukaryotic proteasome involved in proteolysis. The α7 subunit is a component of the α ring, ...The 20S proteasome, which is composed of layered α and β heptameric rings, is the core complex of the eukaryotic proteasome involved in proteolysis. The α7 subunit is a component of the α ring, and it self-assembles into a homo-tetradecamer consisting of two layers of α7 heptameric rings. However, the structure of the α7 double ring in solution has not been fully elucidated. We applied cryo-electron microscopy to delineate the structure of the α7 double ring in solution, revealing a structure different from the previously reported crystallographic model. The D7-symmetrical double ring was stacked with a 15° clockwise twist and a separation of 3 Å between the two rings. Two more conformations, dislocated and fully open, were also identified. Our observations suggest that the α7 double-ring structure fluctuates considerably in solution, allowing for the insertion of homologous α subunits, finally converting to the hetero-heptameric α rings in the 20S proteasome. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30990.map.gz emd_30990.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30990-v30.xml emd-30990-v30.xml emd-30990.xml emd-30990.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30990_fsc.xml emd_30990_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30990.png emd_30990.png | 113.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30990 http://ftp.pdbj.org/pub/emdb/structures/EMD-30990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30990 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30990.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30990.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Symmetric type of alpha 7 homo-tetradecamer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.42 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Proteasome subunit alpha type-7
| 全体 | 名称: Proteasome subunit alpha type-7 プロテアソーム プロテアソーム |
|---|---|
| 要素 |
|
-超分子 #1: Proteasome subunit alpha type-7
| 超分子 | 名称: Proteasome subunit alpha type-7 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Proteasome subunit alpha type-3
| 分子 | 名称: Proteasome subunit alpha type-3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.469252 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSSIGTGYDL SASTFSPDGR VFQVEYAMKA VENSSTAIGI RCKDGVVFGV EKLVLSKLYE EGSNKRLFNV DRHVGMAVAG LLADARSLA DIAREEASNF RSNFGYNIPL KHLADRVAMY VHAYTLYSAV RPFGCSFMLG SYSVNDGAQL YMIDPSGVSY G YWGCAIGK ...文字列: MSSIGTGYDL SASTFSPDGR VFQVEYAMKA VENSSTAIGI RCKDGVVFGV EKLVLSKLYE EGSNKRLFNV DRHVGMAVAG LLADARSLA DIAREEASNF RSNFGYNIPL KHLADRVAMY VHAYTLYSAV RPFGCSFMLG SYSVNDGAQL YMIDPSGVSY G YWGCAIGK ARQAAKTEIE KLQMKEMTCR DIVKEVAKII YIVHDEVKDK AFELELSWVG ELTNGRHEIV PKDIREEAEK YA KESLKEE DESDDDNM |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 糖包埋 | 材質: Ice |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: MOLYBDENUM / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2200FS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 40.0 µm / 倍率(補正後): 45065 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 4.2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 40000 Bright-field microscopy / Cs: 4.2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 40000 |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 76.0 K / 最高: 77.0 K |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-20 (5k x 3k) デジタル化 - サイズ - 横: 5120 pixel / デジタル化 - サイズ - 縦: 3840 pixel / デジタル化 - サンプリング間隔: 6.4 µm / 平均露光時間: 5.0 sec. / 平均電子線量: 40.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー