+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Intermediate resolution structure of barley (1,3;1,4)-beta-glucan synthase CslF6. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | plant-type cell wall organization or biogenesis /  Cellulose synthase / Cellulose synthase /  Cellulose synthase / Cellulose synthase /  cellulose synthase (UDP-forming) activity / cellulose biosynthetic process / Nucleotide-diphospho-sugar transferases / membrane => GO:0016020 / Cellulose synthase-like CslF6 cellulose synthase (UDP-forming) activity / cellulose biosynthetic process / Nucleotide-diphospho-sugar transferases / membrane => GO:0016020 / Cellulose synthase-like CslF6 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Hordeum vulgare (オオムギ) Hordeum vulgare (オオムギ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Ho R / Purushotham P / Zimmer J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Mechanism of mixed-linkage glucan biosynthesis by barley cellulose synthase-like CslF6 (1,3;1,4)-β-glucan synthase. 著者: Pallinti Purushotham / Ruoya Ho / Long Yu / Geoffrey B Fincher / Vincent Bulone / Jochen Zimmer /    要旨: Mixed-linkage (1,3;1,4)-β-glucans, which are widely distributed in cell walls of the grasses, are linear glucose polymers containing predominantly (1,4)-β-linked glucosyl units interspersed with ...Mixed-linkage (1,3;1,4)-β-glucans, which are widely distributed in cell walls of the grasses, are linear glucose polymers containing predominantly (1,4)-β-linked glucosyl units interspersed with single (1,3)-β-linked glucosyl units. Their distribution in cereal grains and unique structures are important determinants of dietary fibers that are beneficial to human health. We demonstrate that the barley cellulose synthase-like CslF6 enzyme is sufficient to synthesize a high-molecular weight (1,3;1,4)-β-glucan in vitro. Biochemical and cryo-electron microscopy analyses suggest that CslF6 functions as a monomer. A conserved "switch motif" at the entrance of the enzyme's transmembrane channel is critical to generate (1,3)-linkages. There, a single-point mutation markedly reduces (1,3)-linkage formation, resulting in the synthesis of cellulosic polysaccharides. Our results suggest that CslF6 monitors the orientation of the nascent polysaccharide's second or third glucosyl unit. Register-dependent interactions with these glucosyl residues reposition the polymer's terminal glucosyl unit to form either a (1,3)- or (1,4)-β-linkage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
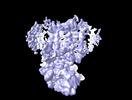
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27655.map.gz emd_27655.map.gz | 92.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27655-v30.xml emd-27655-v30.xml emd-27655.xml emd-27655.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27655.png emd_27655.png | 66 KB | ||
| その他 |  emd_27655_half_map_1.map.gz emd_27655_half_map_1.map.gz emd_27655_half_map_2.map.gz emd_27655_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27655 http://ftp.pdbj.org/pub/emdb/structures/EMD-27655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27655 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8dqkMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27655.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27655.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_27655_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27655_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Barley cellulose synthase-like F6
| 全体 | 名称: Barley cellulose synthase-like F6 |
|---|---|
| 要素 |
|
-超分子 #1: Barley cellulose synthase-like F6
| 超分子 | 名称: Barley cellulose synthase-like F6 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Hordeum vulgare (オオムギ) Hordeum vulgare (オオムギ) |
-分子 #1: Cellulose synthase-like CslF6
| 分子 | 名称: Cellulose synthase-like CslF6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Hordeum vulgare (オオムギ) Hordeum vulgare (オオムギ) |
| 分子量 | 理論値: 105.193797 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAPAVAGGGR VRSNEPVAAA AAAPAASGKP CVCGFQVCAC TGSAAVASAA SSLDMDIVAM GQIGAVNDES WVGVELGEDG ETDESGAAV DDRPVFRTEK IKGVLLHPYR VLIFVRLIAF TLFVIWRISH KNPDAMWLWV TSICGEFWFG FSWLLDQLPK L NPINRVPD ...文字列: MAPAVAGGGR VRSNEPVAAA AAAPAASGKP CVCGFQVCAC TGSAAVASAA SSLDMDIVAM GQIGAVNDES WVGVELGEDG ETDESGAAV DDRPVFRTEK IKGVLLHPYR VLIFVRLIAF TLFVIWRISH KNPDAMWLWV TSICGEFWFG FSWLLDQLPK L NPINRVPD LAVLRQRFDR PDGTSTLPGL DIFVTTADPI KEPILSTANS VLSILAADYP VDRNTCYVSD DSGMLLTYEA LA ESSKFAT LWVPFCRKHG IEPRGPESYF ELKSHPYMGR AQDEFVNDRR RVRKEYDEFK ARINSLEHDI KQRNDGYNAA IAH SQGVPR PTWMADGTQW EGTWVDASEN HRRGDHAGIV LVLLNHPSHR RQTGPPASAD NPLDLSGVDV RLPMLVYVSR EKRP GHDHQ KKAGAMNALT RASALLSNSP FILNLDCDHY INNSQALRAG ICFMVGRDSD TVAFVQFPQR FEGVDPTDLY ANHNR IFFD GTLRALDGMQ GPIYVGTGCL FRRITVYGFD PPRINVGGPC FPRLAGLFAK TKYEKPGLEM TTAKAKAAPV PAKGKH GFL PLPKKTYGKS DAFVDTIPRA SHPSPYAAAA EGIVADEATI VEAVNVTAAA FEKKTGWGKE IGWVYDTVTE DVVTGYR MH IKGWRSRYCS IYPHAFIGTA PINLTERLFQ VLRWSTGSLE IFFSKNNPLF GSTYLHPLQR VAYINITTYP FTAIFLIF Y TTVPALSFVT GHFIVQRPTT MFYVYLGIVL STLLVIAVLE VKWAGVTVFE WFRNGQFWMT ASCSAYLAAV CQVLTKVIF RRDISFKLTS KLPSGDEKKD PYADLYVVRW TPLMITPIII IFVNIIGSAV AFAKVLDGEW THWLKVAGGV FFNFWVLFHL YPFAKGILG KHGKTPVVVL VWWAFTFVIT AVLYINIPHM HTSGGKHTTV HGHHGKKLVD TGLYGWLH |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 53.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: AlphaFold2 predicted structure |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 80347 |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X