+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2718 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Conformational Snapshots of Inducible Nitric Oxide Synthase (iNOS) | |||||||||
 マップデータ マップデータ | Single-particle reconstruction of iNOS: Group I, Conformation i | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  heterogeneity / random conical tilt / heterogeneity / random conical tilt /  nitric oxide synthase (一酸化窒素合成酵素) / nitric oxide synthase (一酸化窒素合成酵素) /  calmodulin (カルモジュリン) / calmodulin (カルモジュリン) /  heme (ヘム) / heme (ヘム) /  electron transfer (電子移動反応) / flavin electron transfer (電子移動反応) / flavin | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Nitric oxide stimulates guanylate cyclase / ROS and RNS production in phagocytes / G protein-coupled receptor signaling pathway coupled to cGMP nucleotide second messenger /  Peroxisomal protein import / Peroxisomal protein import /  cAMP-dependent protein kinase regulator activity / prostaglandin secretion / positive regulation of killing of cells of another organism / cAMP-dependent protein kinase regulator activity / prostaglandin secretion / positive regulation of killing of cells of another organism /  tetrahydrobiopterin binding / tetrahydrobiopterin binding /  arginine binding / cortical cytoskeleton ...Nitric oxide stimulates guanylate cyclase / ROS and RNS production in phagocytes / G protein-coupled receptor signaling pathway coupled to cGMP nucleotide second messenger / arginine binding / cortical cytoskeleton ...Nitric oxide stimulates guanylate cyclase / ROS and RNS production in phagocytes / G protein-coupled receptor signaling pathway coupled to cGMP nucleotide second messenger /  Peroxisomal protein import / Peroxisomal protein import /  cAMP-dependent protein kinase regulator activity / prostaglandin secretion / positive regulation of killing of cells of another organism / cAMP-dependent protein kinase regulator activity / prostaglandin secretion / positive regulation of killing of cells of another organism /  tetrahydrobiopterin binding / tetrahydrobiopterin binding /  arginine binding / cortical cytoskeleton / superoxide metabolic process / cGMP-mediated signaling / arginine binding / cortical cytoskeleton / superoxide metabolic process / cGMP-mediated signaling /  nitric-oxide synthase binding / cellular response to cytokine stimulus / regulation of cytokine production involved in inflammatory response / peptidyl-cysteine S-nitrosylation / regulation of insulin secretion / cellular response to organic cyclic compound / nitric-oxide synthase (NADPH) / blood vessel remodeling / response to tumor necrosis factor / nitric-oxide synthase binding / cellular response to cytokine stimulus / regulation of cytokine production involved in inflammatory response / peptidyl-cysteine S-nitrosylation / regulation of insulin secretion / cellular response to organic cyclic compound / nitric-oxide synthase (NADPH) / blood vessel remodeling / response to tumor necrosis factor /  nitric-oxide synthase activity / nitric oxide mediated signal transduction / arginine catabolic process / nitric oxide biosynthetic process / negative regulation of blood pressure / response to hormone / positive regulation of interleukin-8 production / response to bacterium / nitric-oxide synthase activity / nitric oxide mediated signal transduction / arginine catabolic process / nitric oxide biosynthetic process / negative regulation of blood pressure / response to hormone / positive regulation of interleukin-8 production / response to bacterium /  Hsp90 protein binding / negative regulation of protein catabolic process / Hsp90 protein binding / negative regulation of protein catabolic process /  beta-catenin binding / beta-catenin binding /  血圧 / cellular response to type II interferon / 血圧 / cellular response to type II interferon /  ペルオキシソーム / positive regulation of interleukin-6 production / ペルオキシソーム / positive regulation of interleukin-6 production /  概日リズム / cellular response to xenobiotic stimulus / FMN binding / 概日リズム / cellular response to xenobiotic stimulus / FMN binding /  flavin adenine dinucleotide binding / flavin adenine dinucleotide binding /  NADP binding / NADP binding /  actin binding / regulation of cell population proliferation / cellular response to lipopolysaccharide / response to lipopolysaccharide / response to hypoxia / actin binding / regulation of cell population proliferation / cellular response to lipopolysaccharide / response to lipopolysaccharide / response to hypoxia /  calmodulin binding / intracellular signal transduction / defense response to bacterium / calmodulin binding / intracellular signal transduction / defense response to bacterium /  cadherin binding / cadherin binding /  炎症 / iron ion binding / positive regulation of apoptotic process / negative regulation of gene expression / 炎症 / iron ion binding / positive regulation of apoptotic process / negative regulation of gene expression /  heme binding / heme binding /  protein kinase binding / perinuclear region of cytoplasm / protein homodimerization activity / protein kinase binding / perinuclear region of cytoplasm / protein homodimerization activity /  extracellular space / identical protein binding / extracellular space / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 65.0 Å ネガティブ染色法 / 解像度: 65.0 Å | |||||||||
 データ登録者 データ登録者 | Campbell MG / Smith BC / Potter CS / Carragher B / Marletta MA | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2014 ジャーナル: Proc Natl Acad Sci U S A / 年: 2014タイトル: Molecular architecture of mammalian nitric oxide synthases. 著者: Melody G Campbell / Brian C Smith / Clinton S Potter / Bridget Carragher / Michael A Marletta /  要旨: NOSs are homodimeric multidomain enzymes responsible for producing NO. In mammals, NO acts as an intercellular messenger in a variety of signaling reactions, as well as a cytotoxin in the innate ...NOSs are homodimeric multidomain enzymes responsible for producing NO. In mammals, NO acts as an intercellular messenger in a variety of signaling reactions, as well as a cytotoxin in the innate immune response. Mammals possess three NOS isoforms--inducible, endothelial, and neuronal NOS--that are composed of an N-terminal oxidase domain and a C-terminal reductase domain. Calmodulin (CaM) activates NO synthesis by binding to the helical region connecting these two domains. Although crystal structures of isolated domains have been reported, no structure is available for full-length NOS. We used high-throughput single-particle EM to obtain the structures and higher-order domain organization of all three NOS holoenzymes. The structures of inducible, endothelial, and neuronal NOS with and without CaM bound are similar, consisting of a dimerized oxidase domain flanked by two separated reductase domains. NOS isoforms adopt many conformations enabled by three flexible linkers. These conformations represent snapshots of the continuous electron transfer pathway from the reductase domain to the oxidase domain, which reveal that only a single reductase domain participates in electron transfer at a time, and that CaM activates NOS by constraining rotational motions and by directly binding to the oxidase domain. Direct visualization of these large conformational changes induced during electron transfer provides significant insight into the molecular underpinnings governing NO formation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2718.map.gz emd_2718.map.gz | 56.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2718-v30.xml emd-2718-v30.xml emd-2718.xml emd-2718.xml | 9.8 KB 9.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  image2718.png image2718.png | 80.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2718 http://ftp.pdbj.org/pub/emdb/structures/EMD-2718 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2718 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2718 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  2719C  2720C  2721C  2722C  2723C  2724C  2725C  2726C  2727C  2728C  2729C  2730C 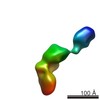 2731C  2732C  2733C  2734C  2735C  2736C  2737C  2738C  2739C  2740C  2741C  2742C  2743C  2744C  2745C  2746C  2747C  2748C  2749C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2718.map.gz / 形式: CCP4 / 大きさ: 162.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2718.map.gz / 形式: CCP4 / 大きさ: 162.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle reconstruction of iNOS: Group I, Conformation i | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Murine Inducible Nitric Oxide Synthase
| 全体 | 名称: Murine Inducible Nitric Oxide Synthase |
|---|---|
| 要素 |
|
-超分子 #1000: Murine Inducible Nitric Oxide Synthase
| 超分子 | 名称: Murine Inducible Nitric Oxide Synthase / タイプ: sample / ID: 1000 / 詳細: Sample is highly flexible / 集合状態: Homodimer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 260 KDa |
-分子 #1: Inducible Nitric Oxide Synthase
| 分子 | 名称: Inducible Nitric Oxide Synthase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: iNOS / コピー数: 1 / 集合状態: Homodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) / 別称: Mouse / 細胞: Macrophages Mus musculus (ハツカネズミ) / 別称: Mouse / 細胞: Macrophages |
| 分子量 | 理論値: 260 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換細胞: JM109 / 組換プラスミド: pCWiNOS Escherichia coli (大腸菌) / 組換細胞: JM109 / 組換プラスミド: pCWiNOS |
| 配列 | UniProtKB:  Nitric oxide synthase, inducible Nitric oxide synthase, inducibleGO: nitric oxide biosynthetic process, FMN binding,  NADP binding, NADP binding,  calmodulin binding, calmodulin binding,  flavin adenine dinucleotide binding, flavin adenine dinucleotide binding,  heme binding, iron ion binding, heme binding, iron ion binding,  nitric-oxide synthase activity nitric-oxide synthase activityInterPro: Ser/Thr protein kinase, TGFB receptor |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 50 mM TEA pH 7.5, 150 mM NaCl, and 5 mM DTT |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: 3 microliters of sample were applied to grid. The specimen was stained twice with 2% uranyl formate, then allowed to air-dry. |
| グリッド | 詳細: Glow discharged C-flat grid with 2-micron-diameter holes overlaid by thin 1.5 nm continuous carbon |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 114705 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 62000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 62000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle max: 55 |
| 日付 | 2013年3月6日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 2226 / 平均電子線量: 37 e/Å2 詳細: Each area is imaged twice: once at 55 degrees and again with no tilt. |
| Tilt angle min | 0 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each Image |
|---|---|
| 最終 2次元分類 | クラス数: 1 |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 65.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Appion, Spider / 詳細: See publication. / 使用した粒子像数: 225 |
| 詳細 | See publication. |
 ムービー
ムービー コントローラー
コントローラー











