+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of D-site Rac1-bound WAVE Regulatory Complex | |||||||||
 マップデータ マップデータ | WRC230VCA-Rac1 complex sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peripheral region of growth cone / negative regulation of synaptic vesicle recycling /  intracellular protein-containing complex / intracellular protein-containing complex /  SCAR complex / positive regulation of neurotrophin TRK receptor signaling pathway / lamellipodium morphogenesis / positive regulation of Arp2/3 complex-mediated actin nucleation / SCAR complex / positive regulation of neurotrophin TRK receptor signaling pathway / lamellipodium morphogenesis / positive regulation of Arp2/3 complex-mediated actin nucleation /  Arp2/3 complex binding / modification of synaptic structure / regulation of actin polymerization or depolymerization ...peripheral region of growth cone / negative regulation of synaptic vesicle recycling / Arp2/3 complex binding / modification of synaptic structure / regulation of actin polymerization or depolymerization ...peripheral region of growth cone / negative regulation of synaptic vesicle recycling /  intracellular protein-containing complex / intracellular protein-containing complex /  SCAR complex / positive regulation of neurotrophin TRK receptor signaling pathway / lamellipodium morphogenesis / positive regulation of Arp2/3 complex-mediated actin nucleation / SCAR complex / positive regulation of neurotrophin TRK receptor signaling pathway / lamellipodium morphogenesis / positive regulation of Arp2/3 complex-mediated actin nucleation /  Arp2/3 complex binding / modification of synaptic structure / regulation of actin polymerization or depolymerization / Arp2/3 complex binding / modification of synaptic structure / regulation of actin polymerization or depolymerization /  regulation of respiratory burst / central region of growth cone / modification of postsynaptic actin cytoskeleton / negative regulation of interleukin-23 production / dendrite extension / regulation of neutrophil migration / localization within membrane / Activated NTRK2 signals through CDK5 / regulation of respiratory burst / central region of growth cone / modification of postsynaptic actin cytoskeleton / negative regulation of interleukin-23 production / dendrite extension / regulation of neutrophil migration / localization within membrane / Activated NTRK2 signals through CDK5 /  regulation of translation at postsynapse, modulating synaptic transmission / regulation of translation at postsynapse, modulating synaptic transmission /  NADPH oxidase complex / negative regulation of receptor-mediated endocytosis / regulation of hydrogen peroxide metabolic process / ruffle assembly / NTRK2 activates RAC1 / engulfment of apoptotic cell / Inactivation of CDC42 and RAC1 / filopodium tip / WNT5:FZD7-mediated leishmania damping / SEMA3A-Plexin repulsion signaling by inhibiting Integrin adhesion / : / regulation of modification of postsynaptic actin cytoskeleton / cortical cytoskeleton organization / NADPH oxidase complex / negative regulation of receptor-mediated endocytosis / regulation of hydrogen peroxide metabolic process / ruffle assembly / NTRK2 activates RAC1 / engulfment of apoptotic cell / Inactivation of CDC42 and RAC1 / filopodium tip / WNT5:FZD7-mediated leishmania damping / SEMA3A-Plexin repulsion signaling by inhibiting Integrin adhesion / : / regulation of modification of postsynaptic actin cytoskeleton / cortical cytoskeleton organization /  respiratory burst / regulation of actin filament polymerization / hepatocyte growth factor receptor signaling pathway / RNA 7-methylguanosine cap binding / ruffle organization / cell projection assembly / respiratory burst / regulation of actin filament polymerization / hepatocyte growth factor receptor signaling pathway / RNA 7-methylguanosine cap binding / ruffle organization / cell projection assembly /  thioesterase binding / negative regulation of fibroblast migration / thioesterase binding / negative regulation of fibroblast migration /  regulation of stress fiber assembly / RHO GTPases activate CIT / Nef and signal transduction / axon extension / sphingosine-1-phosphate receptor signaling pathway / PCP/CE pathway / motor neuron axon guidance / RHO GTPases activate KTN1 / regulation of nitric oxide biosynthetic process / regulation of stress fiber assembly / RHO GTPases activate CIT / Nef and signal transduction / axon extension / sphingosine-1-phosphate receptor signaling pathway / PCP/CE pathway / motor neuron axon guidance / RHO GTPases activate KTN1 / regulation of nitric oxide biosynthetic process /  regulation of lamellipodium assembly / positive regulation of neutrophil chemotaxis / Azathioprine ADME / Activation of RAC1 / positive regulation of ruffle assembly / positive regulation of dendrite development / DCC mediated attractive signaling / positive regulation of cell-substrate adhesion / MET activates RAP1 and RAC1 / regulation of lamellipodium assembly / positive regulation of neutrophil chemotaxis / Azathioprine ADME / Activation of RAC1 / positive regulation of ruffle assembly / positive regulation of dendrite development / DCC mediated attractive signaling / positive regulation of cell-substrate adhesion / MET activates RAP1 and RAC1 /  Wnt signaling pathway, planar cell polarity pathway / Sema4D mediated inhibition of cell attachment and migration / CD28 dependent Vav1 pathway / Ephrin signaling / Wnt signaling pathway, planar cell polarity pathway / Sema4D mediated inhibition of cell attachment and migration / CD28 dependent Vav1 pathway / Ephrin signaling /  protein kinase A binding / protein kinase A binding /  lamellipodium assembly / lamellipodium assembly /  regulation of myelination / cortical actin cytoskeleton organization / positive regulation of Rho protein signal transduction / establishment or maintenance of cell polarity / regulation of myelination / cortical actin cytoskeleton organization / positive regulation of Rho protein signal transduction / establishment or maintenance of cell polarity /  regulation of cell size / DSCAM interactions / Activation of RAC1 downstream of NMDARs / small GTPase-mediated signal transduction / Rho GDP-dissociation inhibitor binding / positive regulation of actin filament polymerization / NRAGE signals death through JNK / Rac protein signal transduction / protein kinase A regulatory subunit binding / filamentous actin / RHO GTPases activate PAKs / dendritic growth cone / positive regulation of focal adhesion assembly / lamellipodium membrane / semaphorin-plexin signaling pathway / regulation of cell size / DSCAM interactions / Activation of RAC1 downstream of NMDARs / small GTPase-mediated signal transduction / Rho GDP-dissociation inhibitor binding / positive regulation of actin filament polymerization / NRAGE signals death through JNK / Rac protein signal transduction / protein kinase A regulatory subunit binding / filamentous actin / RHO GTPases activate PAKs / dendritic growth cone / positive regulation of focal adhesion assembly / lamellipodium membrane / semaphorin-plexin signaling pathway /  excitatory synapse / ficolin-1-rich granule membrane / Sema3A PAK dependent Axon repulsion / RHOG GTPase cycle / EPH-ephrin mediated repulsion of cells / RHO GTPases Activate NADPH Oxidases / RHO GTPases Activate WASPs and WAVEs / anatomical structure morphogenesis / RAC3 GTPase cycle / RAC2 GTPase cycle / RHO GTPases activate IQGAPs / positive regulation of axon extension / localization / PTK6 Regulates RHO GTPases, RAS GTPase and MAP kinases / positive regulation of lamellipodium assembly / axonal growth cone / response to electrical stimulus excitatory synapse / ficolin-1-rich granule membrane / Sema3A PAK dependent Axon repulsion / RHOG GTPase cycle / EPH-ephrin mediated repulsion of cells / RHO GTPases Activate NADPH Oxidases / RHO GTPases Activate WASPs and WAVEs / anatomical structure morphogenesis / RAC3 GTPase cycle / RAC2 GTPase cycle / RHO GTPases activate IQGAPs / positive regulation of axon extension / localization / PTK6 Regulates RHO GTPases, RAS GTPase and MAP kinases / positive regulation of lamellipodium assembly / axonal growth cone / response to electrical stimulus類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
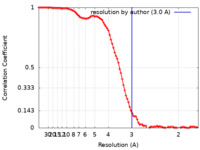
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.0 Å クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Ding B / Yang S / Chen B / Chowdhury S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structures reveal a key mechanism of WAVE regulatory complex activation by Rac1 GTPase. 著者: Bojian Ding / Sheng Yang / Matthias Schaks / Yijun Liu / Abbigale J Brown / Klemens Rottner / Saikat Chowdhury / Baoyu Chen /    要旨: The Rho-family GTPase Rac1 activates the WAVE regulatory complex (WRC) to drive Arp2/3 complex-mediated actin polymerization in many essential processes. Rac1 binds to WRC at two distinct sites-the ...The Rho-family GTPase Rac1 activates the WAVE regulatory complex (WRC) to drive Arp2/3 complex-mediated actin polymerization in many essential processes. Rac1 binds to WRC at two distinct sites-the A and D sites. Precisely how Rac1 binds and how the binding triggers WRC activation remain unknown. Here we report WRC structures by itself, and when bound to single or double Rac1 molecules, at ~3 Å resolutions by cryogenic-electron microscopy. The structures reveal that Rac1 binds to the two sites by distinct mechanisms, and binding to the A site, but not the D site, drives WRC activation. Activation involves a series of unique conformational changes leading to the release of sequestered WCA (WH2-central-acidic) polypeptide, which stimulates the Arp2/3 complex to polymerize actin. Together with biochemical and cellular analyses, the structures provide a novel mechanistic understanding of how the Rac1-WRC-Arp2/3-actin signaling axis is regulated in diverse biological processes and diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
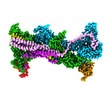
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26733.map.gz emd_26733.map.gz | 12.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26733-v30.xml emd-26733-v30.xml emd-26733.xml emd-26733.xml | 30.6 KB 30.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26733_fsc.xml emd_26733_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26733.png emd_26733.png | 115.7 KB | ||
| マスクデータ |  emd_26733_msk_1.map emd_26733_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_26733_additional_1.map.gz emd_26733_additional_1.map.gz emd_26733_half_map_1.map.gz emd_26733_half_map_1.map.gz emd_26733_half_map_2.map.gz emd_26733_half_map_2.map.gz | 9.2 MB 59 MB 59 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26733 http://ftp.pdbj.org/pub/emdb/structures/EMD-26733 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26733 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26733 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26733.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26733.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WRC230VCA-Rac1 complex sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8757 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26733_msk_1.map emd_26733_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: WRC230VCA-Rac1 complex masked unsharpened map
| ファイル | emd_26733_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WRC230VCA-Rac1 complex masked unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: WRC230VCA-Rac1 complex unmasked unfiltered unsharpened first half map
| ファイル | emd_26733_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WRC230VCA-Rac1 complex unmasked unfiltered unsharpened first half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: WRC230VCA-Rac1 complex unmasked unfiltered unsharpened second half map...
| ファイル | emd_26733_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | WRC230VCA-Rac1 complex unmasked unfiltered unsharpened second half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : WAVE regulatory complex with Rac1 bound to D-site
| 全体 | 名称: WAVE regulatory complex with Rac1 bound to D-site |
|---|---|
| 要素 |
|
-超分子 #1: WAVE regulatory complex with Rac1 bound to D-site
| 超分子 | 名称: WAVE regulatory complex with Rac1 bound to D-site / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 360 KDa |
-分子 #1: Cytoplasmic FMR1-interacting protein 1
| 分子 | 名称: Cytoplasmic FMR1-interacting protein 1 / タイプ: protein_or_peptide / ID: 1 詳細: This construct contains two additional uncleaved residues "GA" in the N terminus from the construct design and purification procedure. Densities for these residues are not observed in the map ...詳細: This construct contains two additional uncleaved residues "GA" in the N terminus from the construct design and purification procedure. Densities for these residues are not observed in the map and were not included in the sample sequence to avoid numbering shifts. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 145.36375 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAAQVTLEDA LSNVDLLEEL PLPDQQPCIE PPPSSLLYQP NFNTNFEDRN AFVTGIARYI EQATVHSSMN EMLEEGQEYA VMLYTWRSC SRAIPQVKCN EQPNRVEIYE KTVEVLEPEV TKLMNFMYFQ RNAIERFCGE VRRLCHAERR KDFVSEAYLI T LGKFINMF ...文字列: MAAQVTLEDA LSNVDLLEEL PLPDQQPCIE PPPSSLLYQP NFNTNFEDRN AFVTGIARYI EQATVHSSMN EMLEEGQEYA VMLYTWRSC SRAIPQVKCN EQPNRVEIYE KTVEVLEPEV TKLMNFMYFQ RNAIERFCGE VRRLCHAERR KDFVSEAYLI T LGKFINMF AVLDELKNMK CSVKNDHSAY KRAAQFLRKM ADPQSIQESQ NLSMFLANHN KITQSLQQQL EVISGYEELL AD IVNLCVD YYENRMYLTP SEKHMLLKVM GFGLYLMDGS VSNIYKLDAK KRINLSKIDK YFKQLQVVPL FGDMQIELAR YIK TSAHYE ENKSRWTCTS SGSSPQYNIC EQMIQIREDH MRFISELARY SNSEVVTGSG RQEAQKTDAE YRKLFDLALQ GLQL LSQWS AHVMEVYSWK LVHPTDKYSN KDCPDSAEEY ERATRYNYTS EEKFALVEVI AMIKGLQVLM GRMESVFNHA IRHTV YAAL QDFSQVTLRE PLRQAIKKKK NVIQSVLQAI RKTVCDWETG HEPFNDPALR GEKDPKSGFD IKVPRRAVGP SSTQLY MVR TMLESLIADK SGSKKTLRSS LEGPTILDIE KFHRESFFYT HLINFSETLQ QCCDLSQLWF REFFLELTMG RRIQFPI EM SMPWILTDHI LETKEASMME YVLYSLDLYN DSAHYALTRF NKQFLYDEIE AEVNLCFDQF VYKLADQIFA YYKVMAGS L LLDKRLRSEC KNQGATIHLP PSNRYETLLK QRHVQLLGRS IDLNRLITQR VSAAMYKSLE LAIGRFESED LTSIVELDG LLEINRMTHK LLSRYLTLDG FDAMFREANH NVSAPYGRIT LHVFWELNYD FLPNYCYNGS TNRFVRTVLP FSQEFQRDKQ PNAQPQYLH GSKALNLAYS SIYGSYRNFV GPPHFQVICR LLGYQGIAVV MEELLKVVKS LLQGTILQYV KTLMEVMPKI C RLPRHEYG SPGILEFFHH QLKDIVEYAE LKTVCFQNLR EVGNAILFCL LIEQSLSLEE VCDLLHAAPF QNILPRVHVK EG ERLDAKM KRLESKYAPL HLVPLIERLG TPQQIAIARE GDLLTKERLC CGLSMFEVIL TRIRSFLDDP IWRGPLPSNG VMH VDECVE FHRLWSAMQF VYCIPVGTHE FTVEQCFGDG LHWAGCMIIV LLGQQRRFAV LDFCYHLLKV QKHDGKDEII KNVP LKKMV ERIRKFQILN DEIITILDKY LKSGDGEGTP VEHVRCFQPP IHQSLASS |
-分子 #2: Nck-associated protein 1
| 分子 | 名称: Nck-associated protein 1 / タイプ: protein_or_peptide / ID: 2 詳細: This construct contains two additional uncleaved residues "GA" in the N terminus from the construct design and purification procedure. Densities for these residues are not observed in the map ...詳細: This construct contains two additional uncleaved residues "GA" in the N terminus from the construct design and purification procedure. Densities for these residues are not observed in the map and were not included in the sample sequence to avoid numbering shifts. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 128.940727 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSRSVLQPSQ QKLAEKLTIL NDRGVGMLTR LYNIKKACGD PKAKPSYLID KNLESAVKFI VRKFPAVETR NNNQQLAQLQ KEKSEILKN LALYYFTFVD VMEFKDHVCE LLNTIDVCQV FFDITVNFDL TKNYLDLIIT YTTLMILLSR IEERKAIIGL Y NYAHEMTH ...文字列: MSRSVLQPSQ QKLAEKLTIL NDRGVGMLTR LYNIKKACGD PKAKPSYLID KNLESAVKFI VRKFPAVETR NNNQQLAQLQ KEKSEILKN LALYYFTFVD VMEFKDHVCE LLNTIDVCQV FFDITVNFDL TKNYLDLIIT YTTLMILLSR IEERKAIIGL Y NYAHEMTH GASDREYPRL GQMIVDYENP LKKMMEEFVP HSKSLSDALI SLQMVYPRRN LSADQWRNAQ LLSLISAPST ML NPAQSDT MPCEYLSLDA MEKWIIFGFI LCHGILNTDA TALNLWKLAL QSSSCLSLFR DEVFHIHKAA EDLFVNIRGY NKR INDIRE CKEAAVSHAG SMHRERRKFL RSALKELATV LSDQPGLLGP KALFVFMALS FARDEIIWLL RHADNMPKKS ADDF IDKHI AELIFYMEEL RAHVRKYGPV MQRYYVQYLS GFDAVVLNEL VQNLSVCPED ESIIMSSFVN TMTSLSVKQV EDGEV FDFR GMRLDWFRLQ AYTSVSKASL GLADHRELGK MMNTIIFHTK MVDSLVEMLV ETSDLSIFCF YSRAFEKMFQ QCLELP SQS RYSIAFPLLC THFMSCTHEL CPEERHHIGD RSLSLCNMFL DEMAKQARNL ITDICTEQCT LSDQLLPKHC AKTISQA VN KKSKKQTGKK GEPEREKPGV ESMRKNRLVV TNLDKLHTAL SELCFSINYV PNMVVWEHTF TPREYLTSHL EIRFTKSI V GMTMYNQATQ EIAKPSELLT SVRAYMTVLQ SIENYVQIDI TRVFNNVLLQ QTQHLDSHGE PTITSLYTNW YLETLLRQV SNGHIAYFPA MKAFVNLPTE NELTFNAEEY SDISEMRSLS ELLGPYGMKF LSESLMWHIS SQVAELKKLV VENVDVLTQM RTSFDKPDQ MAALFKRLSS VDSVLKRMTI IGVILSFRSL AQEALRDVLS YHIPFLVSSI EDFKDHIPRE TDMKVAMNVY E LSSAAGLP CEIDPALVVA LSSQKSENIS PEEEYKIACL LMVFVAVSLP TLASNVMSQY SPAIEGHCNN IHCLAKAINQ IA AALFTIH KGSIEDRLKE FLALASSSLL KIGQETDKTT TRNRESVYLL LDMIVQESPF LTMDLLESCF PYVLLRNAYH AVY KQSVTS SA |
-分子 #3: Wiskott-Aldrich syndrome protein family member 1
| 分子 | 名称: Wiskott-Aldrich syndrome protein family member 1 / タイプ: protein_or_peptide / ID: 3 詳細: Residues 231-248 are inserted as a flexible linker sequence. This construct contains two additional uncleaved residues "GA" in the N terminus from the construct design and purification ...詳細: Residues 231-248 are inserted as a flexible linker sequence. This construct contains two additional uncleaved residues "GA" in the N terminus from the construct design and purification procedure. Densities for these residues are not observed in the map and were not included in the sample sequence to avoid numbering shifts. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.009406 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MPLVKRNIDP RHLCHTALPR GIKNELECVT NISLANIIRQ LSSLSKYAED IFGELFNEAH SFSFRVNSLQ ERVDRLSVSV TQLDPKEEE LSLQDITMRK AFRSSTIQDQ QLFDRKTLPI PLQETYDVCE QPPPLNILTP YRDDGKEGLK FYTNPSYFFD L WKEKMLQD ...文字列: MPLVKRNIDP RHLCHTALPR GIKNELECVT NISLANIIRQ LSSLSKYAED IFGELFNEAH SFSFRVNSLQ ERVDRLSVSV TQLDPKEEE LSLQDITMRK AFRSSTIQDQ QLFDRKTLPI PLQETYDVCE QPPPLNILTP YRDDGKEGLK FYTNPSYFFD L WKEKMLQD TEDKRKEKRK QKQKNLDRPH EPEKVPRAPH DRRREWQKLA QGPELAEDDA NLLHKHIEVA NGGGSGGSGG SG GSGGSGG SKRHPSTLPV ISDARSVLLE AIRKGIQLRK VEEQREQEAK HERIENDVAT ILSRRIAVEY SDSEDDSEFD EVD WLE |
-分子 #4: Protein BRICK1
| 分子 | 名称: Protein BRICK1 / タイプ: protein_or_peptide / ID: 4 詳細: This construct contains uncleaved residues "GHMGAA" in the N terminus from the construct design and purification procedure. Densities for the residues are not observed in the map and were not ...詳細: This construct contains uncleaved residues "GHMGAA" in the N terminus from the construct design and purification procedure. Densities for the residues are not observed in the map and were not included in the sample sequence to avoid numbering shifts. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.756915 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAGQEDPVQR EIHQDWANRE YIEIITSSIK KIADFLNSFD MSCRSRLATL NEKLTALERR IEYIEARVTK GETLT |
-分子 #5: Abl interactor 2
| 分子 | 名称: Abl interactor 2 / タイプ: protein_or_peptide / ID: 5 詳細: The sequence only contains residues 1-158. Also, there are two additional uncleaved residues "GH" in the N terminus from the construct design and purification procedure. Densities for these ...詳細: The sequence only contains residues 1-158. Also, there are two additional uncleaved residues "GH" in the N terminus from the construct design and purification procedure. Densities for these residues are not observed in the map and were not included in the sample sequence to avoid numbering shifts. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.041482 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAELQMLLEE EIPGGRRALF DSYTNLERVA DYCENNYIQS ADKQRALEET KAYTTQSLAS VAYLINTLAN NVLQMLDIQA SQLRRMESS INHISQTVDI HKEKVARREI GILTTNKNTS RTHKIIAPAN LERPVRYIRK PIDYTILDDI GHGVKVSTQ |
-分子 #6: Ras-related C3 botulinum toxin substrate 1
| 分子 | 名称: Ras-related C3 botulinum toxin substrate 1 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO / EC番号:  低分子量GTPアーゼ 低分子量GTPアーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 21.010486 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MQAIKCVVVG DGAVGKTCLL ISYTTNAFSG EYIPTVFDNY SANVMVDGKP VNLGLWDTAG LEDYDRLRPL SYPQTDVFLI CFSLVSPAS FENVRAKWYP EVRHHCPNTP IILVGTKLDL RDDKDTIEKL KEKKLTPITY PQGLAMAKEI GAVKYLECSA L TQRGLKTV FDEAIRAVLC PPPVKKRKRK |
-分子 #7: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-分子 #8: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 8 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.41 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.019 kPa / 詳細: 30 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 120000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | Data were collected by shifting the stage to target exposure positions. |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 実像数: 2512 / 平均露光時間: 40.0 sec. / 平均電子線量: 45.27 e/Å2 詳細: Each micrograph was acquired as dose-fractionated movies consisting of 62 frames per movie. |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X