+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2663 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of SMG1C complex, comprising SMG1 kinase, SMG8 and SMG9 | |||||||||
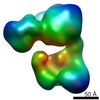
 マップデータ マップデータ | Recosntruction of the SMG1C complex, comprising SMG1, SMG8 and SMG9 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NMD /  SMG1 / SMG8 / SMG9 / PIKK / RNA degradation SMG1 / SMG8 / SMG9 / PIKK / RNA degradation | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA metabolic process / diacylglycerol-dependent serine/threonine kinase activity /  chromatoid body / chromatoid body /  eye development / eye development /  regulation of telomere maintenance / regulation of telomere maintenance /  regulation of protein kinase activity / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / telomeric DNA binding / phosphatidylinositol phosphate biosynthetic process / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) ...RNA metabolic process / diacylglycerol-dependent serine/threonine kinase activity / regulation of protein kinase activity / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / telomeric DNA binding / phosphatidylinositol phosphate biosynthetic process / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) ...RNA metabolic process / diacylglycerol-dependent serine/threonine kinase activity /  chromatoid body / chromatoid body /  eye development / eye development /  regulation of telomere maintenance / regulation of telomere maintenance /  regulation of protein kinase activity / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / telomeric DNA binding / phosphatidylinositol phosphate biosynthetic process / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / mRNA export from nucleus / regulation of protein kinase activity / nuclear-transcribed mRNA catabolic process, nonsense-mediated decay / telomeric DNA binding / phosphatidylinositol phosphate biosynthetic process / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / mRNA export from nucleus /  brain development / brain development /  heart development / peptidyl-serine phosphorylation / in utero embryonic development / protein autophosphorylation / heart development / peptidyl-serine phosphorylation / in utero embryonic development / protein autophosphorylation /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / protein kinase activity /  DNA修復 / protein serine kinase activity / protein serine/threonine kinase activity / DNA damage response / negative regulation of apoptotic process / DNA修復 / protein serine kinase activity / protein serine/threonine kinase activity / DNA damage response / negative regulation of apoptotic process /  RNA binding / RNA binding /  核質 / 核質 /  ATP binding / identical protein binding / ATP binding / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 20.8 Å ネガティブ染色法 / 解像度: 20.8 Å | |||||||||
 データ登録者 データ登録者 | Melero R / Uchiyama A / Castano R / Kataoka N / Kurosawa H / Ohno S / Yamashita A / Llorca O | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2014 ジャーナル: Structure / 年: 2014タイトル: Structures of SMG1-UPFs complexes: SMG1 contributes to regulate UPF2-dependent activation of UPF1 in NMD. 著者: Roberto Melero / Akiko Uchiyama / Raquel Castaño / Naoyuki Kataoka / Hitomi Kurosawa / Shigeo Ohno / Akio Yamashita / Oscar Llorca /   要旨: SMG1, a PI3K-related kinase, plays a critical role in nonsense-mediated mRNA decay (NMD) in mammals. SMG1-mediated phosphorylation of the UPF1 helicase is an essential step during NMD initiation. ...SMG1, a PI3K-related kinase, plays a critical role in nonsense-mediated mRNA decay (NMD) in mammals. SMG1-mediated phosphorylation of the UPF1 helicase is an essential step during NMD initiation. Both SMG1 and UPF1 are presumably activated by UPF2, but this regulation is incompletely understood. Here we reveal that SMG1C (a complex containing SMG1, SMG8, and SMG9) contributes to regulate NMD by recruiting UPF1 and UPF2 to distinct sites in the vicinity of the kinase domain. UPF2 binds SMG1 in an UPF1-independent manner in vivo, and the SMG1C-UPF2 structure shows UPF2 recognizes the FRB domain, a region that regulates the related mTOR kinase. The molecular architectures of several SMG1C-UPFs complexes, obtained by combining electron microscopy with in vivo and in vitro interaction analyses, competition experiments, and mutations, suggest that UPF2 can be transferred to UPF1 within SMG1C, inducing UPF2-dependent conformational changes required to activate UPF1 within an SMG1C-UPF1-UPF2 complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2663.map.gz emd_2663.map.gz | 10.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2663-v30.xml emd-2663-v30.xml emd-2663.xml emd-2663.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2663.png EMD-2663.png | 68 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2663 http://ftp.pdbj.org/pub/emdb/structures/EMD-2663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2663 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2663.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2663.map.gz / 形式: CCP4 / 大きさ: 11.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Recosntruction of the SMG1C complex, comprising SMG1, SMG8 and SMG9 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SMG1C complex, comprising SMG1 kinase, SMG8 and SMG9
| 全体 | 名称: SMG1C complex, comprising SMG1 kinase, SMG8 and SMG9 |
|---|---|
| 要素 |
|
-超分子 #1000: SMG1C complex, comprising SMG1 kinase, SMG8 and SMG9
| 超分子 | 名称: SMG1C complex, comprising SMG1 kinase, SMG8 and SMG9 タイプ: sample / ID: 1000 / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 580 KDa |
-分子 #1: Serine/threonine-protein kinase SMG1
| 分子 | 名称: Serine/threonine-protein kinase SMG1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: SMG-1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 410 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: 293T cells Homo sapiens (ヒト) / 組換細胞: 293T cells |
| 配列 | UniProtKB: Serine/threonine-protein kinase SMG1 GO:  DNA修復, RNA metabolic process, nuclear-transcribed mRNA catabolic process, nonsense-mediated decay, DNA修復, RNA metabolic process, nuclear-transcribed mRNA catabolic process, nonsense-mediated decay,  ATP binding ATP binding |
-分子 #2: SMG8
| 分子 | 名称: SMG8 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: SMG-8 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 109 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: 293T cells Homo sapiens (ヒト) / 組換細胞: 293T cells |
| 配列 | UniProtKB: Nonsense-mediated mRNA decay factor SMG8 GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: Nonsense-mediated mRNA decay factor SMG8 |
-分子 #3: SMG9
| 分子 | 名称: SMG9 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: SMG-9 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 60 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: 293T cells Homo sapiens (ヒト) / 組換細胞: 293T cells |
| 配列 | UniProtKB: Nonsense-mediated mRNA decay factor SMG9 GO: nuclear-transcribed mRNA catabolic process, nonsense-mediated decay InterPro: P-loop containing nucleoside triphosphate hydrolase, Nonsense-mediated mRNA decay factor SMG8/SMG9 |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 10 mM HEPES-KOH, 150 mM NaCl, 20% glycerol, 10 mM MgCl2 |
| 染色 | タイプ: NEGATIVE / 詳細: 1% uranyl formate |
| グリッド | 詳細: 400 mesh grid with thin carbon support, glow discharged |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1230 |
|---|---|
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 倍率(補正後): 54926 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.9 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 40000 Bright-field microscopy / Cs: 2.9 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 40000 |
| 試料ステージ | 試料ホルダーモデル: JEOL |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected using a TVIPS F416 CMOS and the EM-MENU software (TVIPS) |
| 日付 | 2012年4月5日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) デジタル化 - サンプリング間隔: 15.6 µm / 実像数: 542 / 平均電子線量: 15 e/Å2 詳細: Using a TVIPS F416 CMOS and the EM-TOOLS software (TVIPS) ビット/ピクセル: 16 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph using BSOFT |
|---|---|
| 最終 2次元分類 | クラス数: 490 |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.8 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN, EMAN2, Xmipp / 使用した粒子像数: 21020 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The structure was separately fitted using Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | HEAT repeat regions from DNA-PKcs were separately fitted into SMG1 using Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー