+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24952 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | tRNA-like Structure from Brome Mosaic Virus | ||||||||||||
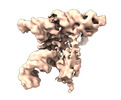
 マップデータ マップデータ | Cryo-EM map of brome mosaic virus (BMV) tRNA-like structure (TLS) generated using cryoSPARC. | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |    Brome mosaic virus (ウイルス) Brome mosaic virus (ウイルス) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.3 Å クライオ電子顕微鏡法 / 解像度: 4.3 Å | ||||||||||||
 データ登録者 データ登録者 | Kieft JS / Bonilla SL | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: A viral RNA hijacks host machinery using dynamic conformational changes of a tRNA-like structure. 著者: Steve L Bonilla / Madeline E Sherlock / Andrea MacFadden / Jeffrey S Kieft /  要旨: Viruses require multifunctional structured RNAs to hijack their host’s biochemistry, but their mechanisms can be obscured by the difficulty of solving conformationally dynamic RNA structures. Using ...Viruses require multifunctional structured RNAs to hijack their host’s biochemistry, but their mechanisms can be obscured by the difficulty of solving conformationally dynamic RNA structures. Using cryo–electron microscopy (cryo-EM), we visualized the structure of the mysterious viral transfer RNA (tRNA)–like structure (TLS) from the brome mosaic virus, which affects replication, translation, and genome encapsidation. Structures in isolation and those bound to tyrosyl-tRNA synthetase (TyrRS) show that this ~55-kilodalton purported tRNA mimic undergoes large conformational rearrangements to bind TyrRS in a form that differs substantially from that of tRNA. Our study reveals how viral RNAs can use a combination of static and dynamic RNA structures to bind host machinery through highly noncanonical interactions, and we highlight the utility of cryo-EM for visualizing small, conformationally dynamic structured RNAs. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24952.map.gz emd_24952.map.gz | 326.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24952-v30.xml emd-24952-v30.xml emd-24952.xml emd-24952.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24952_fsc.xml emd_24952_fsc.xml | 19.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24952.png emd_24952.png | 85.4 KB | ||
| その他 |  emd_24952_additional_1.map.gz emd_24952_additional_1.map.gz emd_24952_half_map_1.map.gz emd_24952_half_map_1.map.gz emd_24952_half_map_2.map.gz emd_24952_half_map_2.map.gz | 630.4 MB 621 MB 621 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24952 http://ftp.pdbj.org/pub/emdb/structures/EMD-24952 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24952 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24952 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7samMC  7sc6C  7scqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10993 (タイトル: Cryo-EM micrographs of BMV TLS RNA / Data size: 6.7 TB EMPIAR-10993 (タイトル: Cryo-EM micrographs of BMV TLS RNA / Data size: 6.7 TBData #1: Aligned micrographs (with and without dose-weighting), dataset 1 [micrographs - single frame] Data #2: Aligned micrographs (with and without dose-weighting), dataset 2 [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24952.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24952.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of brome mosaic virus (BMV) tRNA-like structure (TLS) generated using cryoSPARC. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.413 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Sharpened (B factor: 257.7) cryo-EM map of brome...
| ファイル | emd_24952_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened (B factor: 257.7) cryo-EM map of brome mosaic virus (BMV) tRNA-like structure (TLS) generated using cryoSPARC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map A of brome mosaic virus (BMV) tRNA-like...
| ファイル | emd_24952_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map A of brome mosaic virus (BMV) tRNA-like structure (TLS) generated using cryoSPARC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map B of brome mosaic virus (BMV) tRNA-like...
| ファイル | emd_24952_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map B of brome mosaic virus (BMV) tRNA-like structure (TLS) generated using cryoSPARC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : tRNA-like structure at the 3' UTR of the brome mosaic virus (BMV)...
| 全体 | 名称: tRNA-like structure at the 3' UTR of the brome mosaic virus (BMV) genome |
|---|---|
| 要素 |
|
-超分子 #1: tRNA-like structure at the 3' UTR of the brome mosaic virus (BMV)...
| 超分子 | 名称: tRNA-like structure at the 3' UTR of the brome mosaic virus (BMV) genome タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: RNA was generated by in vitro transcription of sequence-verified linear DNA template |
|---|---|
| 由来(天然) | 生物種:    Brome mosaic virus (ウイルス) Brome mosaic virus (ウイルス) |
| 分子量 | 理論値: 54.56 KDa |
-分子 #1: Viral RNA
| 分子 | 名称: Viral RNA / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:    Brome mosaic virus (ウイルス) Brome mosaic virus (ウイルス) |
| 分子量 | 理論値: 55.044559 KDa |
| 配列 | 文字列: GGCGUGGUUG ACACGCAGAC CUCUUACAAG AGUGUCUAGG UGCCUUUGAG AGUUACUCUU UGCUCUCUUC GGAAGAACCC UUAGGGGUU CGUGCAUGGG CUUGCAUAGC AAGUCUUAGA AUGCGGGUAC CGUACAGUGU UGAAAAACAC UGUAAAUCUC U AAAAGAGA CCA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 構成要素 - 濃度: 50.0 mM / 構成要素 - 名称: NaMOPS / 詳細: Solution contained 10 mM magnesium chloride. |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | This sample was monodisperse as determined by native gel electrophoresis. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -0.8 µm / 最小 デフォーカス(公称値): -2.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -0.8 µm / 最小 デフォーカス(公称値): -2.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: OTHER |
|---|---|
| 最終 角度割当 | タイプ: OTHER |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 4.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 128226 |
FSC曲線 (解像度の算出) | 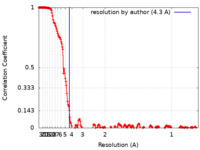 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7sam: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X