+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24107 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
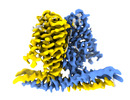
| タイトル | Cryo-EM structure of TACAN in the apo form (TMEM120A) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein heterooligomerization / nuclear inner membrane / fat cell differentiation / detection of mechanical stimulus involved in sensory perception of pain / monoatomic ion transmembrane transport / protein homooligomerization / monoatomic ion channel activity /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Niu Y / Tao X / MacKinnon R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Analysis of the mechanosensor channel functionality of TACAN. 著者: Yiming Niu / Xiao Tao / George Vaisey / Paul Dominic B Olinares / Hanan Alwaseem / Brian T Chait / Roderick MacKinnon /  要旨: Mechanosensitive ion channels mediate transmembrane ion currents activated by mechanical forces. A mechanosensitive ion channel called TACAN was recently reported. We began to study TACAN with the ...Mechanosensitive ion channels mediate transmembrane ion currents activated by mechanical forces. A mechanosensitive ion channel called TACAN was recently reported. We began to study TACAN with the intent to understand how it senses mechanical forces and functions as an ion channel. Using cellular patch-recording methods, we failed to identify mechanosensitive ion channel activity. Using membrane reconstitution methods, we found that TACAN, at high protein concentrations, produces heterogeneous conduction levels that are not mechanosensitive and are most consistent with disruptions of the lipid bilayer. We determined the structure of TACAN using single-particle cryo-electron microscopy and observed that it is a symmetrical dimeric transmembrane protein. Each protomer contains an intracellular-facing cleft with a coenzyme A cofactor, confirmed by mass spectrometry. The TACAN protomer is related in three-dimensional structure to a fatty acid elongase, ELOVL7. Whilst its physiological function remains unclear, we anticipate that TACAN is not a mechanosensitive ion channel. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24107.map.gz emd_24107.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24107-v30.xml emd-24107-v30.xml emd-24107.xml emd-24107.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24107.png emd_24107.png | 101.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24107 http://ftp.pdbj.org/pub/emdb/structures/EMD-24107 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24107 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24107 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24107.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24107.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homo-dimeric assembly of wild type TACAN(TMEM120A)
| 全体 | 名称: Homo-dimeric assembly of wild type TACAN(TMEM120A) |
|---|---|
| 要素 |
|
-超分子 #1: Homo-dimeric assembly of wild type TACAN(TMEM120A)
| 超分子 | 名称: Homo-dimeric assembly of wild type TACAN(TMEM120A) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Ion channel TACAN
| 分子 | 名称: Ion channel TACAN / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 40.797297 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MQSPPPDPLG DCLRNWEDLQ QDFQGIQETH RLYRLKLEEL TKLQANCTNS ITRQKKRLQE LALVLKKCRP SLPSESMEAA QELENQMKE RQGLFFDMEA YLPKKNGLYL SLVLGNVNVT LLSKQAKFAY KDEYEKFKLY LTIILIVISF TCRFLLNSRV T DAAFNFLL ...文字列: MQSPPPDPLG DCLRNWEDLQ QDFQGIQETH RLYRLKLEEL TKLQANCTNS ITRQKKRLQE LALVLKKCRP SLPSESMEAA QELENQMKE RQGLFFDMEA YLPKKNGLYL SLVLGNVNVT LLSKQAKFAY KDEYEKFKLY LTIILIVISF TCRFLLNSRV T DAAFNFLL VWYYCTLTIR ESILINNGSR IKGWWVFHHY VSTFLSGVML TWPDGLMYQK FRNQFLSFSM YQSFVQFLQY YY QSGCLYR LRALGERHTM DLTVEGFQSW MWRGLTFLLP FLFFGHFWQL FNALTLFNLA RDPECKEWQV LMCGFPFLLL FLG NFFTTL RVVHQKFHSQ QHGNKKD |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 20 mM HEPES pH 7.4, 250 mM NaCl, and 0.06% Digitonin (w/v) |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 75.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: Gctf (ver. 1.0.6) |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD ソフトウェア: (名称: RELION (ver. 3.0), RELION (ver. 3.1), cryoSPARC (ver. 2.9.0)) |
| 最終 3次元分類 | クラス数: 4 / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2.9.0) / 使用した粒子像数: 110090 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2.9.0) / 使用した粒子像数: 110090 |
 ムービー
ムービー コントローラー
コントローラー