+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23859 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mouse CTPS1 bound to inhibitor R80 | |||||||||
 マップデータ マップデータ | Mouse CTPS1 bound to inhibitor R80 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Interconversion of nucleotide di- and triphosphates /  cytoophidium / CTP synthase (glutamine hydrolysing) / cytoophidium / CTP synthase (glutamine hydrolysing) /  CTP synthase activity / 'de novo' CTP biosynthetic process / pyrimidine nucleobase biosynthetic process / CTP biosynthetic process / glutamine metabolic process / B cell proliferation / T cell proliferation ...Interconversion of nucleotide di- and triphosphates / CTP synthase activity / 'de novo' CTP biosynthetic process / pyrimidine nucleobase biosynthetic process / CTP biosynthetic process / glutamine metabolic process / B cell proliferation / T cell proliferation ...Interconversion of nucleotide di- and triphosphates /  cytoophidium / CTP synthase (glutamine hydrolysing) / cytoophidium / CTP synthase (glutamine hydrolysing) /  CTP synthase activity / 'de novo' CTP biosynthetic process / pyrimidine nucleobase biosynthetic process / CTP biosynthetic process / glutamine metabolic process / B cell proliferation / T cell proliferation / CTP synthase activity / 'de novo' CTP biosynthetic process / pyrimidine nucleobase biosynthetic process / CTP biosynthetic process / glutamine metabolic process / B cell proliferation / T cell proliferation /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.4 Å クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Lynch EM / Dimattia MA / Kollman JM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Structural basis for isoform-specific inhibition of human CTPS1. 著者: Eric M Lynch / Michael A DiMattia / Steven Albanese / Gydo C P van Zundert / Jesse M Hansen / Joel D Quispe / Madison A Kennedy / Andreas Verras / Kenneth Borrelli / Angela V Toms / Neelu ...著者: Eric M Lynch / Michael A DiMattia / Steven Albanese / Gydo C P van Zundert / Jesse M Hansen / Joel D Quispe / Madison A Kennedy / Andreas Verras / Kenneth Borrelli / Angela V Toms / Neelu Kaila / Kevin D Kreutter / Joshua J McElwee / Justin M Kollman /  要旨: Cytidine triphosphate synthase 1 (CTPS1) is necessary for an effective immune response, as revealed by severe immunodeficiency in CTPS1-deficient individuals [E. Martin ], [] [510], [288-292] ([2014]) ...Cytidine triphosphate synthase 1 (CTPS1) is necessary for an effective immune response, as revealed by severe immunodeficiency in CTPS1-deficient individuals [E. Martin ], [] [510], [288-292] ([2014]). CTPS1 expression is up-regulated in activated lymphocytes to expand CTP pools [E. Martin ], [] [510], [288-292] ([2014]), satisfying increased demand for nucleic acid and lipid synthesis [L. D. Fairbanks, M. Bofill, K. Ruckemann, H. A. Simmonds], [ ] [270], [29682-29689] ([1995]). Demand for CTP in other tissues is met by the CTPS2 isoform and nucleoside salvage pathways [E. Martin ], [] [510], [288-292] ([2014]). Selective inhibition of the proliferative CTPS1 isoform is therefore desirable in the treatment of immune disorders and lymphocyte cancers, but little is known about differences in regulation of the isoforms or mechanisms of known inhibitors. We show that CTP regulates both isoforms by binding in two sites that clash with substrates. CTPS1 is less sensitive to CTP feedback inhibition, consistent with its role in increasing CTP levels in proliferation. We also characterize recently reported small-molecule inhibitors, both CTPS1 selective and nonselective. Cryo-electron microscopy (cryo-EM) structures reveal these inhibitors mimic CTP binding in one inhibitory site, where a single amino acid substitution explains selectivity for CTPS1. The inhibitors bind to CTPS assembled into large-scale filaments, which for CTPS1 normally represents a hyperactive form of the enzyme [E. M. Lynch ], [] [24], [507-514] ([2017]). This highlights the utility of cryo-EM in drug discovery, particularly for cases in which targets form large multimeric assemblies not amenable to structure determination by other techniques. Both inhibitors also inhibit the proliferation of human primary T cells. The mechanisms of selective inhibition of CTPS1 lay the foundation for the design of immunosuppressive therapies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23859.map.gz emd_23859.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23859-v30.xml emd-23859-v30.xml emd-23859.xml emd-23859.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23859.png emd_23859.png | 198.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23859 http://ftp.pdbj.org/pub/emdb/structures/EMD-23859 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23859 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23859 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7mipMC  7mgzC  7mh0C  7mh1C  7mifC  7migC  7mihC  7miiC  7miuC  7mivC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23859.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23859.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Mouse CTPS1 bound to inhibitor R80 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : mCTPS1 tetramer
| 全体 | 名称: mCTPS1 tetramer |
|---|---|
| 要素 |
|
-超分子 #1: mCTPS1 tetramer
| 超分子 | 名称: mCTPS1 tetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: CTP synthase 1
| 分子 | 名称: CTP synthase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: CTP synthase (glutamine hydrolysing) |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 66.770398 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKYILVTGGV ISGIGKGVIA SSVGTILKSC GLHVTSIKID PYINIDAGTF SPYEHGEVFV LDDGGEVDLD LGNYERFLDI RLTKDNNLT TGKIYQYVIN KERKGDYLGK TVQVVPHITD AIQEWVMRQA LIPVDEDGLE PQVCVIELGG TVGDIESMPF I EAFRQFQF ...文字列: MKYILVTGGV ISGIGKGVIA SSVGTILKSC GLHVTSIKID PYINIDAGTF SPYEHGEVFV LDDGGEVDLD LGNYERFLDI RLTKDNNLT TGKIYQYVIN KERKGDYLGK TVQVVPHITD AIQEWVMRQA LIPVDEDGLE PQVCVIELGG TVGDIESMPF I EAFRQFQF KVKRENFCNI HVSLVPQPSS TGEQKTKPTQ NSVRELRGLG LSPDLVVCRC SNPLDTSVKE KISMFCHVEP EQ VICVHDV SSIYRVPLLL EEQGVVDYFL RRLDLPIERQ SRKMLMKWKE MADRYDRLLE TCSIALVGKY TKFSDSYASV IKA LEHSAL AINHKLEIKY IDSTDLEPST LQEEPVRYHE AWQKLCSAHG VLVPGGFGVR GTEGKIQAIA WARKQKKPFL GVCL GMQLA VVEFSRNVLG WQDANSTEFD PKTSHPVVID MPEHNPGQMG GTMRLGKRRT LFQTKNSVMR KLYGDTDYLE ERHRH RFEV NPVLKKCLEE QGLKFVGQDV EGERMEIVEL EDHPFFVGVQ YHPEFLSRPI KPSPPYFGLL LASVGRLPHY LQKGCR LSP RDTYSDRSGS SSPDSEITEL KFPSISQD |
-分子 #2: GLUTAMINE
| 分子 | 名称: GLUTAMINE / タイプ: ligand / ID: 2 / コピー数: 4 / 式: GLN |
|---|---|
| 分子量 | 理論値: 146.144 Da |
| Chemical component information | 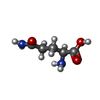 ChemComp-GLN: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: URIDINE 5'-TRIPHOSPHATE
| 分子 | 名称: URIDINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 4 / 式: UTP |
|---|---|
| 分子量 | 理論値: 484.141 Da |
| Chemical component information |  ChemComp-UTP: |
-分子 #5: N-(1-{2-[(cyclopropanesulfonyl)amino]-1,3-thiazol-4-yl}cyclopropy...
| 分子 | 名称: N-(1-{2-[(cyclopropanesulfonyl)amino]-1,3-thiazol-4-yl}cyclopropyl)-5-(6-ethoxypyrazin-2-yl)pyridine-2-carboxamide タイプ: ligand / ID: 5 / コピー数: 4 / 式: ZG4 |
|---|---|
| 分子量 | 理論値: 486.567 Da |
| Chemical component information | 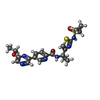 ChemComp-ZG4: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 90.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 再構成 | 想定した対称性 - 点群: D2 (2回x2回 2面回転対称 ) )解像度のタイプ: BY AUTHOR / 解像度: 2.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 238267 |
 ムービー
ムービー コントローラー
コントローラー
















