+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23630 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
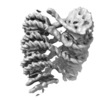
| タイトル | H2A.Z Dodecanucleosome 30 nm Fiber | |||||||||
 マップデータ マップデータ | Chromatin fiber containing mouse histone variant H2A.Z | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Inhibition of DNA recombination at telomere / DNA Damage/Telomere Stress Induced Senescence / Recognition and association of DNA glycosylase with site containing an affected purine / Deposition of new CENPA-containing nucleosomes at the centromere / Cleavage of the damaged purine / heterochromatin formation => GO:0031507 / Condensation of Prophase Chromosomes / PRC2 methylates histones and DNA / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / multicellular organism development ...Inhibition of DNA recombination at telomere / DNA Damage/Telomere Stress Induced Senescence / Recognition and association of DNA glycosylase with site containing an affected purine / Deposition of new CENPA-containing nucleosomes at the centromere / Cleavage of the damaged purine / heterochromatin formation => GO:0031507 / Condensation of Prophase Chromosomes / PRC2 methylates histones and DNA / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / multicellular organism development / nucleosomal DNA binding / RNA polymerase II core promoter sequence-specific DNA binding /  ヘテロクロマチン / cellular response to estradiol stimulus / ヘテロクロマチン / cellular response to estradiol stimulus /  ユークロマチン / chromatin DNA binding / structural constituent of chromatin / cellular response to insulin stimulus / ユークロマチン / chromatin DNA binding / structural constituent of chromatin / cellular response to insulin stimulus /  ヌクレオソーム / RNA polymerase II cis-regulatory region sequence-specific DNA binding / protein heterodimerization activity / positive regulation of transcription by RNA polymerase II / ヌクレオソーム / RNA polymerase II cis-regulatory region sequence-specific DNA binding / protein heterodimerization activity / positive regulation of transcription by RNA polymerase II /  DNA binding / DNA binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |  Xenopus laevis (アフリカツメガエル) / Xenopus laevis (アフリカツメガエル) /   Mus musculus (ハツカネズミ) / unidentified (未定義) Mus musculus (ハツカネズミ) / unidentified (未定義) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.5 Å クライオ電子顕微鏡法 / 解像度: 7.5 Å | |||||||||
 データ登録者 データ登録者 | Lewis T / Sokolova V / Ng H / Tan D | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2021 ジャーナル: Nucleic Acids Res / 年: 2021タイトル: Structural basis of chromatin regulation by histone variant H2A.Z. 著者: Tyler S Lewis / Vladyslava Sokolova / Harry Jung / Honkit Ng / Dongyan Tan /  要旨: The importance of histone variant H2A.Z in transcription regulation has been well established, yet its mechanism-of-action remains enigmatic. Conflicting evidence exists in support of both an ...The importance of histone variant H2A.Z in transcription regulation has been well established, yet its mechanism-of-action remains enigmatic. Conflicting evidence exists in support of both an activating and a repressive role of H2A.Z in transcription. Here we report cryo-electron microscopy (cryo-EM) structures of nucleosomes and chromatin fibers containing H2A.Z and those containing canonical H2A. The structures show that H2A.Z incorporation results in substantial structural changes in both nucleosome and chromatin fiber. While H2A.Z increases the mobility of DNA terminus in nucleosomes, it simultaneously enables nucleosome arrays to form a more regular and condensed chromatin fiber. We also demonstrated that H2A.Z's ability to enhance nucleosomal DNA mobility is largely attributed to its characteristic shorter C-terminus. Our study provides the structural basis for H2A.Z-mediated chromatin regulation, showing that the increase flexibility of the DNA termini in H2A.Z nucleosomes is central to its dual-functions in chromatin regulation and in transcription. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23630.map.gz emd_23630.map.gz | 16.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23630-v30.xml emd-23630-v30.xml emd-23630.xml emd-23630.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23630_fsc.xml emd_23630_fsc.xml | 12.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23630.png emd_23630.png | 73.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23630 http://ftp.pdbj.org/pub/emdb/structures/EMD-23630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23630 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23630.map.gz / 形式: CCP4 / 大きさ: 58 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23630.map.gz / 形式: CCP4 / 大きさ: 58 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Chromatin fiber containing mouse histone variant H2A.Z | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.079 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : In vitro reconstituted 12x167 bp chromatin
+超分子 #1: In vitro reconstituted 12x167 bp chromatin
+超分子 #2: Histone H2A.Z
+超分子 #3: Histone H2B
+超分子 #4: 12 tandem repeats of 167 bp of 601 Windom nucleosome-positioning ...
+超分子 #5: Histone H3
+超分子 #6: Histone H4
+分子 #1: Histone H2A.Z
+分子 #2: Histone H2B
+分子 #3: Histone H3
+分子 #4: Histone H4
+分子 #5: 12 tandem repeats of 167 bp of 601 Windom nucleosome-positioning ...
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Sample was absorbed on the grid for thirty seconds, then blotted for five seconds with a blot force zero before plunge freezing.. | ||||||||||||
| 詳細 | The sample is crosslinked with 0.1% glutaraldehyde |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 5 / 実像数: 14425 / 平均電子線量: 31.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー