+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22844 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
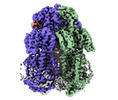
| タイトル | Cryo-electron microscopy structure of the heavy metal efflux pump CusA in a homogeneous binding copper(1) state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | efflux /  pump (ポンプ) / heavy metal. copper / pump (ポンプ) / heavy metal. copper /  silver (銀) / closed / open / transport / silver (銀) / closed / open / transport /  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報silver ion transport / silver ion transmembrane transporter activity / plasma membrane copper ion transport / copper ion transmembrane transport / response to silver ion / silver ion transmembrane transport / copper ion export / copper ion transmembrane transporter activity / detoxification of copper ion / response to copper ion ...silver ion transport / silver ion transmembrane transporter activity / plasma membrane copper ion transport / copper ion transmembrane transport / response to silver ion / silver ion transmembrane transport / copper ion export / copper ion transmembrane transporter activity / detoxification of copper ion / response to copper ion / xenobiotic transmembrane transporter activity / intracellular copper ion homeostasis / response to toxic substance / copper ion binding /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Moseng MA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2021 ジャーナル: mBio / 年: 2021タイトル: Cryo-EM Structures of CusA Reveal a Mechanism of Metal-Ion Export. 著者: Mitchell A Moseng / Meinan Lyu / Tanadet Pipatpolkai / Przemyslaw Glaza / Corey C Emerson / Phoebe L Stewart / Phillip J Stansfeld / Edward W Yu /   要旨: Gram-negative bacteria utilize the resistance-nodulation-cell division (RND) superfamily of efflux pumps to expel a variety of toxic compounds from the cell. The CusA membrane protein, which ...Gram-negative bacteria utilize the resistance-nodulation-cell division (RND) superfamily of efflux pumps to expel a variety of toxic compounds from the cell. The CusA membrane protein, which recognizes and extrudes biocidal Cu(I) and Ag(I) ions, belongs to the heavy-metal efflux (HME) subfamily of RND efflux pumps. We here report four structures of the trimeric CusA heavy-metal efflux pump in the presence of Cu(I) using single-particle cryo-electron microscopy (cryo-EM). We discover that different CusA protomers within the trimer are able to bind Cu(I) ions simultaneously. Our structural data combined with molecular dynamics (MD) simulations allow us to propose a mechanism for ion transport where each CusA protomer functions independently within the trimer. The bacterial RND superfamily of efflux pumps mediate resistance to a variety of biocides, including Cu(I) and Ag(I) ions. Here we report four cryo-EM structures of the trimeric CusA pump in the presence of Cu(I). Combined with MD simulations, our data indicate that each CusA protomer within the trimer recognizes and extrudes Cu(I) independently. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22844.map.gz emd_22844.map.gz | 12.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22844-v30.xml emd-22844-v30.xml emd-22844.xml emd-22844.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22844.png emd_22844.png | 185.7 KB | ||
| Filedesc metadata |  emd-22844.cif.gz emd-22844.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22844 http://ftp.pdbj.org/pub/emdb/structures/EMD-22844 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22844 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22844 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22844.map.gz / 形式: CCP4 / 大きさ: 13.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22844.map.gz / 形式: CCP4 / 大きさ: 13.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CusA in symmetric copper binding form
| 全体 | 名称: CusA in symmetric copper binding form |
|---|---|
| 要素 |
|
-超分子 #1: CusA in symmetric copper binding form
| 超分子 | 名称: CusA in symmetric copper binding form / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Cation efflux system protein CusA
| 分子 | 名称: Cation efflux system protein CusA / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 115.833945 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGHHHHHHMI EWIIRRSVAN RFLVLMGALF LSIWGTWTII NTPVDALPDL SDVQVIIKTS YPGQAPQIVE NQVTYPLTTT MLSVPGAKT VRGFSQFGDS YVYVIFEDGT DPYWARSRVL EYLNQVQGKL PAGVSAELGP DATGVGWIYE YALVDRSGKH D LADLRSLQ ...文字列: MGHHHHHHMI EWIIRRSVAN RFLVLMGALF LSIWGTWTII NTPVDALPDL SDVQVIIKTS YPGQAPQIVE NQVTYPLTTT MLSVPGAKT VRGFSQFGDS YVYVIFEDGT DPYWARSRVL EYLNQVQGKL PAGVSAELGP DATGVGWIYE YALVDRSGKH D LADLRSLQ DWFLKYELKT IPDVAEVASV GGVVKEYQVV IDPQRLAQYG ISLAEVKSAL DASNQEAGGS SIELAEAEYM VR ASGYLQT LDDFNHIVLK ASENGVPVYL RDVAKVQIGP EMRRGIAELN GEGEVAGGVV ILRSGKNARE VIAAVKDKLE TLK SSLPEG VEIVTTYDRS QLIDRAIDNL SGKLLEEFIV VAVVCALFLW HVRSALVAII SLPLGLCIAF IVMHFQGLNA NIMS LGGIA IAVGAMVDAA IVMIENAHKR LEEWQHQHPD ATLDNKTRWQ VITDASVEVG PALFISLLII TLSFIPIFTL EGQEG RLFG PLAFTKTYAM AGAALLAIVV IPILMGYWIR GKIPPESSNP LNRFLIRVYH PLLLKVLHWP KTTLLVAALS VLTVLW PLN KVGGEFLPQI NEGDLLYMPS TLPGISAAEA ASMLQKTDKL IMSVPEVARV FGKTGKAETA TDSAPLEMVE TTIQLKP QE QWRPGMTMDK IIEELDNTVR LPGLANLWVP PIRNRIDMLS TGIKSPIGIK VSGTVLADID AMAEQIEEVA RTVPGVAS A LAERLEGGRY INVEINREKA ARYGMTVADV QLFVTSAVGG AMVGETVEGI ARYPINLRYP QSWRDSPQAL RQLPILTPM KQQITLADVA DIKVSTGPSM LKTENARPTS WIYIDARDRD MVSVVHDLQK AIAEKVQLKP GTSVAFSGQF ELLERANHKL KLMVPMTLM IIFVLLYLAF RRVGEALLII SSVPFALVGG IWLLWWMGFH LSVATGTGFI ALAGVAAEFG VVMLMYLRHA I EAVPSLNN PQTFSEQKLD EALYHGAVLR VRPKAMTVAV IIAGLLPILW GTGAGSEVMS RIAAPMIGGM ITAPLLSLFI IP AAYKLMW LHRHRVRK UniProtKB: Cation efflux system protein CusA |
-分子 #2: COPPER (I) ION
| 分子 | 名称: COPPER (I) ION / タイプ: ligand / ID: 2 / コピー数: 3 / 式: CU1 |
|---|---|
| 分子量 | 理論値: 63.546 Da |
| Chemical component information |  ChemComp-CU1: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.5 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: OTHER |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 3452 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7kf6: |
 ムービー
ムービー コントローラー
コントローラー