+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22757 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Aldolase, rabbit muscle (beam-tilt refinement x3) Fructose-bisphosphate aldolase Fructose-bisphosphate aldolase | |||||||||
 マップデータ マップデータ | Aldolase, rabbit muscle (beam-tilt refinement x3) Fructose-bisphosphate aldolase Fructose-bisphosphate aldolase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  glycolysis (解糖系) / glycolysis (解糖系) /  gluconeogenesis (糖新生) / carbohydrate degradation / gluconeogenesis (糖新生) / carbohydrate degradation /  homotetramer / homotetramer /  LYASE (リアーゼ) LYASE (リアーゼ) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of Arp2/3 complex-mediated actin nucleation /  fructose-bisphosphate aldolase / fructose-bisphosphate aldolase /  fructose-bisphosphate aldolase activity / fructose-bisphosphate aldolase activity /  M band / I band / glycolytic process / protein homotetramerization / positive regulation of cell migration M band / I band / glycolytic process / protein homotetramerization / positive regulation of cell migration類似検索 - 分子機能 | |||||||||
| 生物種 |   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) | |||||||||
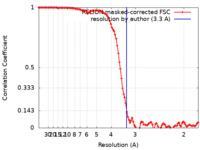
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Kearns SK / Cash JN | |||||||||
| 資金援助 | 2件
| |||||||||
 引用 引用 |  ジャーナル: IUCrJ / 年: 2020 ジャーナル: IUCrJ / 年: 2020タイトル: High-resolution cryo-EM using beam-image shift at 200 keV. 著者: Jennifer N Cash / Sarah Kearns / Yilai Li / Michael A Cianfrocco /  要旨: Recent advances in single-particle cryo-electron microscopy (cryo-EM) data collection utilize beam-image shift to improve throughput. Despite implementation on 300 keV cryo-EM instruments, it ...Recent advances in single-particle cryo-electron microscopy (cryo-EM) data collection utilize beam-image shift to improve throughput. Despite implementation on 300 keV cryo-EM instruments, it remains unknown how well beam-image-shift data collection affects data quality on 200 keV instruments and the extent to which aberrations can be computationally corrected. To test this, a cryo-EM data set for aldolase was collected at 200 keV using beam-image shift and analyzed. This analysis shows that the instrument beam tilt and particle motion initially limited the resolution to 4.9 Å. After particle polishing and iterative rounds of aberration correction in , a 2.8 Å resolution structure could be obtained. This analysis demonstrates that software correction of microscope aberrations can provide a significant improvement in resolution at 200 keV. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22757.map.gz emd_22757.map.gz | 96.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22757-v30.xml emd-22757-v30.xml emd-22757.xml emd-22757.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22757_fsc.xml emd_22757_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22757.png emd_22757.png | 78.4 KB | ||
| Filedesc metadata |  emd-22757.cif.gz emd-22757.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22757 http://ftp.pdbj.org/pub/emdb/structures/EMD-22757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22757 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ka3MC  7k9lC  7k9xC  7ka2C  7ka4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10519 (タイトル: Single particle cryo electron microscopy of aldolase (rabbit, muscle) using beam-tilt on Talos Arctica EMPIAR-10519 (タイトル: Single particle cryo electron microscopy of aldolase (rabbit, muscle) using beam-tilt on Talos ArcticaData size: 87.6 Data #1: Unaligned micrographs of aldolase collected with beam-tilt at 200 kV [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22757.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22757.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Aldolase, rabbit muscle (beam-tilt refinement x3) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homotetramer of aldolase
| 全体 | 名称: Homotetramer of aldolase |
|---|---|
| 要素 |
|
-超分子 #1: Homotetramer of aldolase
| 超分子 | 名称: Homotetramer of aldolase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
| 分子量 | 理論値: 157 kDa/nm |
-分子 #1: Fructose-bisphosphate aldolase A
| 分子 | 名称: Fructose-bisphosphate aldolase A / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  fructose-bisphosphate aldolase fructose-bisphosphate aldolase |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
| 分子量 | 理論値: 39.263672 KDa |
| 組換発現 | 生物種:   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
| 配列 | 文字列: PHSHPALTPE QKKELSDIAH RIVAPGKGIL AADESTGSIA KRLQSIGTEN TEENRRFYRQ LLLTADDRVN PCIGGVILFH ETLYQKADD GRPFPQVIKS KGGVVGIKVD KGVVPLAGTN GETTTQGLDG LSERCAQYKK DGADFAKWRC VLKIGEHTPS A LAIMENAN ...文字列: PHSHPALTPE QKKELSDIAH RIVAPGKGIL AADESTGSIA KRLQSIGTEN TEENRRFYRQ LLLTADDRVN PCIGGVILFH ETLYQKADD GRPFPQVIKS KGGVVGIKVD KGVVPLAGTN GETTTQGLDG LSERCAQYKK DGADFAKWRC VLKIGEHTPS A LAIMENAN VLARYASICQ QNGIVPIVEP EILPDGDHDL KRCQYVTEKV LAAVYKALSD HHIYLEGTLL KPNMVTPGHA CT QKYSHEE IAMATVTALR RTVPPAVTGV TFLSGGQSEE EASINLNAIN KCPLLKPWAL TFSYGRALQA SALKAWGGKK ENL KAAQEE YVKRALANSL ACQGKYTPSG QAGAAASESL FISNHAY UniProtKB: Fructose-bisphosphate aldolase A |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.6 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||
| 詳細 | Pure aldolase isolated from rabbit muscle was purchased as a lyophilized powder (Sigma Aldrich) and solubilized in 20 mM HEPES (pH 7.5), 50 mM NaCl at 1.6 mg/ml. Sample was blotted for 4 seconds with Whatman No. #1 filter paper immediately prior to plunge freezing in liquid ethane cooled by liquid nitrogen. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 4500 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.002 µm / 最小 デフォーカス(公称値): 0.0008 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.002 µm / 最小 デフォーカス(公称値): 0.0008 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 42.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー