+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22513 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Satellite phage P4 procapsid including size determination (Sid) protein | |||||||||
 マップデータ マップデータ | Final masked and sharpened map reconstructed without applying symmetry from symmetry expanded particle. Normalized to mean=0 and 1 stddev. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  Bacteriophage P2, capsid / Phage major capsid protein, P2 family / Bacteriophage P2, capsid / Phage major capsid protein, P2 family /  カプシド / カプシド /  Glycoprotein 3 / Glycoprotein 3 /  カプシド カプシド 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /  Escherichia phage P2 (ファージ) / Escherichia phage P2 (ファージ) /   Enterobacteria phage P4 (ファージ) Enterobacteria phage P4 (ファージ) | |||||||||
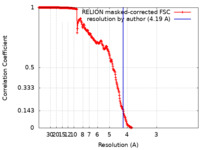
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.19 Å クライオ電子顕微鏡法 / 解像度: 4.19 Å | |||||||||
 データ登録者 データ登録者 | Kizziah JL / Dokland T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2020 ジャーナル: Viruses / 年: 2020タイトル: Structure of the Capsid Size-Determining Scaffold of "Satellite" Bacteriophage P4. 著者: James L Kizziah / Cynthia M Rodenburg / Terje Dokland /  要旨: P4 is a mobile genetic element (MGE) that can exist as a plasmid or integrated into its host genome, but becomes packaged into phage particles by a helper bacteriophage, such as P2. P4 is the ...P4 is a mobile genetic element (MGE) that can exist as a plasmid or integrated into its host genome, but becomes packaged into phage particles by a helper bacteriophage, such as P2. P4 is the original example of what we have termed "molecular piracy", the process by which one MGE usurps the life cycle of another for its own propagation. The P2 helper provides most of the structural gene products for assembly of the P4 virion. However, when P4 is mobilized by P2, the resulting capsids are smaller than those normally formed by P2 alone. The P4-encoded protein responsible for this size change is called Sid, which forms an external scaffolding cage around the P4 procapsids. We have determined the high-resolution structure of P4 procapsids, allowing us to build an atomic model for Sid as well as the gpN capsid protein. Sixty copies of Sid form an intertwined dodecahedral cage around the = 4 procapsid, making contact with only one out of the four symmetrically non-equivalent copies of gpN. Our structure provides a basis for understanding the mutants in gpN that prevent small capsid formation, as well as the "super-sid" mutations that counteract the effect of the mutations, and suggests a model for capsid size redirection by Sid. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22513.map.gz emd_22513.map.gz | 2.3 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22513-v30.xml emd-22513-v30.xml emd-22513.xml emd-22513.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22513_fsc.xml emd_22513_fsc.xml | 30.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22513.png emd_22513.png | 310.7 KB | ||
| マスクデータ |  emd_22513_msk_1.map emd_22513_msk_1.map | 2.4 GB |  マスクマップ マスクマップ | |
| その他 |  emd_22513_half_map_1.map.gz emd_22513_half_map_1.map.gz emd_22513_half_map_2.map.gz emd_22513_half_map_2.map.gz | 2 GB 2 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22513 http://ftp.pdbj.org/pub/emdb/structures/EMD-22513 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22513 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22513 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22513.map.gz / 形式: CCP4 / 大きさ: 2.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22513.map.gz / 形式: CCP4 / 大きさ: 2.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final masked and sharpened map reconstructed without applying symmetry from symmetry expanded particle. Normalized to mean=0 and 1 stddev. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22513_msk_1.map emd_22513_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 from auto-refinement of final map.
| ファイル | emd_22513_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 from auto-refinement of final map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from auto-refinement of final map.
| ファイル | emd_22513_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from auto-refinement of final map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : P4 procapsid
| 全体 | 名称: P4 procapsid |
|---|---|
| 要素 |
|
-超分子 #1: P4 procapsid
| 超分子 | 名称: P4 procapsid / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: BL21(DE3) Escherichia coli (大腸菌) / 組換株: BL21(DE3) |
-分子 #1: Major capsid protein gpN
| 分子 | 名称: Major capsid protein gpN / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage P2 (ファージ) Escherichia phage P2 (ファージ) |
| 分子量 | 理論値: 40.291484 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MRQETRFKFN AYLSRVAELN GIDAGDVSKK FTVEPSVTQT LMNTMQESSD FLTRINIVPV SEMKGEKIGI GVTGSIASTT DTAGGTERQ PKDFSKLASN KYECDQINFD FYIRYKTLDL WARYQDFQLR IRNAIIKRQS LDFIMAGFNG VKRAETSDRS S NPMLQDVA ...文字列: MRQETRFKFN AYLSRVAELN GIDAGDVSKK FTVEPSVTQT LMNTMQESSD FLTRINIVPV SEMKGEKIGI GVTGSIASTT DTAGGTERQ PKDFSKLASN KYECDQINFD FYIRYKTLDL WARYQDFQLR IRNAIIKRQS LDFIMAGFNG VKRAETSDRS S NPMLQDVA VGWLQKYRNE APARVMSKVT DEEGRTTSEV IRVGKGGDYA SLDALVMDAT NNLIEPWYQE DPDLVVIVGR QL LADKYFP IVNKEQDNSE MLAADVIISQ KRIGNLPAVR VPYFPADAML ITKLENLSIY YMDDSHRRVI EENPKLDRVE NYE SMNIDY VVEDYAAGCL VEKIKVGDFS TPAKATAEPG A |
-分子 #2: Size determination protein Sid
| 分子 | 名称: Size determination protein Sid / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage P4 (ファージ) Enterobacteria phage P4 (ファージ) |
| 分子量 | 理論値: 27.296814 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSDHTIPEYL QPALAQLEKA RAAHLENARL MDETVTAIER AEQEKNALAQ ADGNDADDWR TAFRAAGGVL SDELKQRHIE RVARRELVQ EYDNLAVVLN FERERLKGAC DSTATAYRKA HHHLLSLYAE HELEHALNET CEALVRAMHL SILVQENPLA N TTGHQGYV ...文字列: MSDHTIPEYL QPALAQLEKA RAAHLENARL MDETVTAIER AEQEKNALAQ ADGNDADDWR TAFRAAGGVL SDELKQRHIE RVARRELVQ EYDNLAVVLN FERERLKGAC DSTATAYRKA HHHLLSLYAE HELEHALNET CEALVRAMHL SILVQENPLA N TTGHQGYV APEKAVMQQV KSSLEQKIKQ MQISLTGEPV LRLTGLSAAT LPHMDYEVAG TPAQRKVWQD KIDQQGAELK AR GLLS |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 30.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X