+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22168 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of EBV BFLF1 | |||||||||
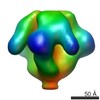
 マップデータ マップデータ | BFLF1 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | viral packaging / viral cleavage /  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||
| 機能・相同性 | Herpesvirus major envelope glycoprotein / Herpesvirus putative major envelope glycoprotein / host cell cytoplasm /  エンベロープ (ウイルス) / host cell nucleus / エンベロープ (ウイルス) / host cell nucleus /  細胞質 / 細胞質 /  Packaging protein UL32 / Packaging protein UL32 /  Packaging protein UL32 homolog Packaging protein UL32 homolog 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Epstein-Barr virus (strain GD1) (ヘルペスウイルス) Epstein-Barr virus (strain GD1) (ヘルペスウイルス) | |||||||||
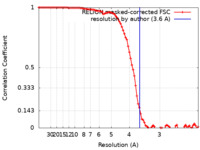
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Didychuk AL / Gates SN / Martin A / Glaunsinger B | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: A pentameric protein ring with novel architecture is required for herpesviral packaging. 著者: Allison L Didychuk / Stephanie N Gates / Matthew R Gardner / Lisa M Strong / Andreas Martin / Britt A Glaunsinger /  要旨: Genome packaging in large double-stranded DNA viruses requires a powerful molecular motor to force the viral genome into nascent capsids, which involves essential accessory factors that are poorly ...Genome packaging in large double-stranded DNA viruses requires a powerful molecular motor to force the viral genome into nascent capsids, which involves essential accessory factors that are poorly understood. Here, we present structures of two such accessory factors from the oncogenic herpesviruses Kaposi's sarcoma-associated herpesvirus (KSHV; ORF68) and Epstein-Barr virus (EBV; BFLF1). These homologous proteins form highly similar homopentameric rings with a positively charged central channel that binds double-stranded DNA. Mutation of individual positively charged residues within but not outside the channel ablates DNA binding, and in the context of KSHV infection, these mutants fail to package the viral genome or produce progeny virions. Thus, we propose a model in which ORF68 facilitates the transfer of newly replicated viral genomes to the packaging motor. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22168.map.gz emd_22168.map.gz | 6.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22168-v30.xml emd-22168-v30.xml emd-22168.xml emd-22168.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22168_fsc.xml emd_22168_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22168.png emd_22168.png | 328.8 KB | ||
| Filedesc metadata |  emd-22168.cif.gz emd-22168.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22168 http://ftp.pdbj.org/pub/emdb/structures/EMD-22168 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22168 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22168 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22168.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22168.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | BFLF1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.14 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Decamer structure of EBV BFLF1
| 全体 | 名称: Decamer structure of EBV BFLF1 |
|---|---|
| 要素 |
|
-超分子 #1: Decamer structure of EBV BFLF1
| 超分子 | 名称: Decamer structure of EBV BFLF1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Epstein-Barr virus (strain GD1) (ヘルペスウイルス) Epstein-Barr virus (strain GD1) (ヘルペスウイルス) |
| 分子量 | 理論値: 536 KDa |
-分子 #1: Packaging protein UL32
| 分子 | 名称: Packaging protein UL32 / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Epstein-Barr virus (strain GD1) (ヘルペスウイルス) Epstein-Barr virus (strain GD1) (ヘルペスウイルス)株: GD1 |
| 分子量 | 理論値: 53.678797 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEDFVPWTVD NLKSQFEAVG LLMAHSYLPA NAEEGIAYPP LVHTYESLSP ASTCRVCDLL DTLVNHSDAP VAFFEDYALL CYYCLNAPR AWISSLITGM DFLHILIKYF PMAGGLDSLF MPSRILAIDI QLHFYICRCF LPVSSSDMIR NANLGYYKLE F LKSILTGQ ...文字列: MEDFVPWTVD NLKSQFEAVG LLMAHSYLPA NAEEGIAYPP LVHTYESLSP ASTCRVCDLL DTLVNHSDAP VAFFEDYALL CYYCLNAPR AWISSLITGM DFLHILIKYF PMAGGLDSLF MPSRILAIDI QLHFYICRCF LPVSSSDMIR NANLGYYKLE F LKSILTGQ SPANFCFKSM WPRTTPTFLT LPGPRTCKDS QDVPGDVGRG LYTALCCHLP TRNRVQHPFL RAEKGGLSPE IT TKADYCG LLLGTWQGTD LLGGPGHHAI GLNAEYSGDE LAELALAITR PEAGDHSQGP CLLAPMFGLR HKNASRTICP LCE SLGAHP DAKDTLDRFK SLILDSFGNN IKILDRIVFL IKTQNTLLDV PCPRLRAWLQ MCTPQDFHKH LFCDPLCAIN HSIT NPSVL FGQIYPPSFQ AFKAALAAGQ NLEQGVCDSL ITLVYIFKST QVARVGKTIL VDVTKELDVV LRIHGLDLVQ SYQTS QVYV UniProtKB:  Packaging protein UL32 Packaging protein UL32 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 20 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.6 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20 | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: 2 second blot, 3 second wait. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 839 / 平均露光時間: 2.4 sec. / 平均電子線量: 48.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー