+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22009 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human TRPA1 in complex with agonist GNE551 | |||||||||
 マップデータ マップデータ | Density-modified map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  TRPA1 / channel / TRPA1 / channel /  agonist (アゴニスト) / agonist (アゴニスト) /  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  MEMBRANE PROTEIN-Agonist complex (生体膜) MEMBRANE PROTEIN-Agonist complex (生体膜) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報temperature-gated cation channel activity / stereocilium bundle / detection of chemical stimulus involved in sensory perception of pain /  thermoception / thermoception /  TRPチャネル / channel activity / response to pain / cellular response to organic substance / intracellularly gated calcium channel activity / detection of mechanical stimulus involved in sensory perception of pain ...temperature-gated cation channel activity / stereocilium bundle / detection of chemical stimulus involved in sensory perception of pain / TRPチャネル / channel activity / response to pain / cellular response to organic substance / intracellularly gated calcium channel activity / detection of mechanical stimulus involved in sensory perception of pain ...temperature-gated cation channel activity / stereocilium bundle / detection of chemical stimulus involved in sensory perception of pain /  thermoception / thermoception /  TRPチャネル / channel activity / response to pain / cellular response to organic substance / intracellularly gated calcium channel activity / detection of mechanical stimulus involved in sensory perception of pain / monoatomic ion transport / sensory perception of pain / response to cold / response to organic substance / calcium ion transmembrane transport / TRPチャネル / channel activity / response to pain / cellular response to organic substance / intracellularly gated calcium channel activity / detection of mechanical stimulus involved in sensory perception of pain / monoatomic ion transport / sensory perception of pain / response to cold / response to organic substance / calcium ion transmembrane transport /  calcium channel activity / intracellular calcium ion homeostasis / response to organic cyclic compound / cellular response to hydrogen peroxide / protein homotetramerization / cell surface receptor signaling pathway / response to xenobiotic stimulus / identical protein binding / calcium channel activity / intracellular calcium ion homeostasis / response to organic cyclic compound / cellular response to hydrogen peroxide / protein homotetramerization / cell surface receptor signaling pathway / response to xenobiotic stimulus / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
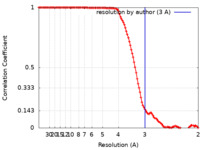
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.0 Å クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Rohou A / Rouge L / Chen H | |||||||||
 引用 引用 |  ジャーナル: Neuron / 年: 2021 ジャーナル: Neuron / 年: 2021タイトル: A Non-covalent Ligand Reveals Biased Agonism of the TRPA1 Ion Channel. 著者: Chang Liu / Rebecca Reese / Simon Vu / Lionel Rougé / Shannon D Shields / Satoko Kakiuchi-Kiyota / Huifen Chen / Kevin Johnson / Yu Patrick Shi / Tania Chernov-Rogan / Demi Maria Zabala ...著者: Chang Liu / Rebecca Reese / Simon Vu / Lionel Rougé / Shannon D Shields / Satoko Kakiuchi-Kiyota / Huifen Chen / Kevin Johnson / Yu Patrick Shi / Tania Chernov-Rogan / Demi Maria Zabala Greiner / Pawan Bir Kohli / David Hackos / Bobby Brillantes / Christine Tam / Tianbo Li / Jianyong Wang / Brian Safina / Steve Magnuson / Matthew Volgraf / Jian Payandeh / Jie Zheng / Alexis Rohou / Jun Chen /  要旨: The TRPA1 ion channel is activated by electrophilic compounds through the covalent modification of intracellular cysteine residues. How non-covalent agonists activate the channel and whether covalent ...The TRPA1 ion channel is activated by electrophilic compounds through the covalent modification of intracellular cysteine residues. How non-covalent agonists activate the channel and whether covalent and non-covalent agonists elicit the same physiological responses are not understood. Here, we report the discovery of a non-covalent agonist, GNE551, and determine a cryo-EM structure of the TRPA1-GNE551 complex, revealing a distinct binding pocket and ligand-interaction mechanism. Unlike the covalent agonist allyl isothiocyanate, which elicits channel desensitization, tachyphylaxis, and transient pain, GNE551 activates TRPA1 into a distinct conducting state without desensitization and induces persistent pain. Furthermore, GNE551-evoked pain is relatively insensitive to antagonist treatment. Thus, we demonstrate the biased agonism of TRPA1, a finding that has important implications for the discovery of effective drugs tailored to different disease etiologies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22009.map.gz emd_22009.map.gz | 5.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22009-v30.xml emd-22009-v30.xml emd-22009.xml emd-22009.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22009_fsc.xml emd_22009_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22009.png emd_22009.png | 155.3 KB | ||
| Filedesc metadata |  emd-22009.cif.gz emd-22009.cif.gz | 6.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22009 http://ftp.pdbj.org/pub/emdb/structures/EMD-22009 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22009 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22009 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22009.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22009.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density-modified map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TRPA1 bound by agonist GNE551
| 全体 | 名称: TRPA1 bound by agonist GNE551 |
|---|---|
| 要素 |
|
-超分子 #1: TRPA1 bound by agonist GNE551
| 超分子 | 名称: TRPA1 bound by agonist GNE551 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Transient receptor potential cation channel subfamily A member 1
| 分子 | 名称: Transient receptor potential cation channel subfamily A member 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 72.622125 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: SPLHFAASYG RINTCQRLLQ DISDTRLLNE GDLHGMTPLH LAAKNGHDKV VQLLLKKGAL FLSDHNGWTA LHHASMGGYT QTMKVILDT NLKCTDRLDE DGNTALHFAA REGHAKAVAL LLSHNADIVL NKQQASFLHL ALHNKRKEVV LTIIRSKRWD E CLKIFSHN ...文字列: SPLHFAASYG RINTCQRLLQ DISDTRLLNE GDLHGMTPLH LAAKNGHDKV VQLLLKKGAL FLSDHNGWTA LHHASMGGYT QTMKVILDT NLKCTDRLDE DGNTALHFAA REGHAKAVAL LLSHNADIVL NKQQASFLHL ALHNKRKEVV LTIIRSKRWD E CLKIFSHN SPGNKCPITE MIEYLPECMK VLLDFCMLHS TEDKSCRDYY IEYNFKYLQC PLEFTKKTPT QDVIYEPLTA LN AMVQNNR IELLNHPVCK EYLLMKWLAY GFRAHMMNLG SYCLGLIPMT ILVVNIKPGM AFNSTGIINE TSDHSEILDT TNS YLIKTC MILVFLSSIF GYCKEAGQIF QQKRNYFMDI SNVLEWIIYT TGIIFVLPLF VEIPAHLQWQ CGAIAVYFYW MNFL LYLQR FENCGIFIVM LEVILKTLLR STVVFIFLLL AFGLSFYILL NLQDPFSSPL LSIIQTFSMM LGDINYRESF LEPYL RNEL AHPVLSFAQL VSFTIFVPIV LMNLLIGLAV GDIADVQKHA SLKRIAMQVE LHTSLEKKLP LWFLRKVDQK STIVYP NKP RSGGMLFHIF CFLFCTGEIR QEIPNADKSL EMEILKQKYR LKDLTFLLEK QHELIKLIIQ KMEIISET UniProtKB: Transient receptor potential cation channel subfamily A member 1 |
-分子 #2: 5-amino-1-[(4-bromo-2-fluorophenyl)methyl]-N-(2,5-dimethoxyphenyl...
| 分子 | 名称: 5-amino-1-[(4-bromo-2-fluorophenyl)methyl]-N-(2,5-dimethoxyphenyl)-1H-1,2,3-triazole-4-carboxamide タイプ: ligand / ID: 2 / コピー数: 4 / 式: ULJ |
|---|---|
| 分子量 | 理論値: 450.262 Da |
| Chemical component information |  ChemComp-ULJ: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.33 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.2 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: HOLEY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: GOLD / 支持フィルム - #1 - トポロジー: HOLEY / 支持フィルム - #1 - Film thickness: 25 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.1 kPa / 詳細: Solarus plasma cleaner | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Triple blot. Put blot in vitroblot Apply 3.5ul to grid, wait 30sec, blot manually Apply 3.5ul to grid, wait 30sec, blot manually, apply final 3.5ul, final blot by vitrobot (3.5s). |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 165000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 11063 / 平均露光時間: 1.6 sec. / 平均電子線量: 41.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー