+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21989 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Inward-facing state of the glutamate transporter homologue GltPh in complex with L-aspartate and sodium ions | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報amino acid:sodium symporter activity / L-aspartate transmembrane transport / L-aspartate transmembrane transporter activity / L-aspartate import across plasma membrane / chloride transmembrane transporter activity / protein homotrimerization / chloride transmembrane transport / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.05 Å クライオ電子顕微鏡法 / 解像度: 3.05 Å | |||||||||
 データ登録者 データ登録者 | Wang X / Boudker O | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Large domain movements through the lipid bilayer mediate substrate release and inhibition of glutamate transporters. 著者: Xiaoyu Wang / Olga Boudker /  要旨: Glutamate transporters are essential players in glutamatergic neurotransmission in the brain, where they maintain extracellular glutamate below cytotoxic levels and allow for rounds of transmission. ...Glutamate transporters are essential players in glutamatergic neurotransmission in the brain, where they maintain extracellular glutamate below cytotoxic levels and allow for rounds of transmission. The structural bases of their function are well established, particularly within a model archaeal homolog, sodium, and aspartate symporter Glt. However, the mechanism of gating on the cytoplasmic side of the membrane remains ambiguous. We report Cryo-EM structures of Glt reconstituted into nanodiscs, including those structurally constrained in the cytoplasm-facing state and either apo, bound to sodium ions only, substrate, or blockers. The structures show that both substrate translocation and release involve movements of the bulky transport domain through the lipid bilayer. They further reveal a novel mode of inhibitor binding and show how solutes release is coupled to protein conformational changes. Finally, we describe how domain movements are associated with the displacement of bound lipids and significant membrane deformations, highlighting the potential regulatory role of the bilayer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21989.map.gz emd_21989.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21989-v30.xml emd-21989-v30.xml emd-21989.xml emd-21989.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21989.png emd_21989.png | 84.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21989 http://ftp.pdbj.org/pub/emdb/structures/EMD-21989 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21989 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21989 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21989.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21989.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.0605 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of inward-facing state of GltPh with L-aspartate and sodi...
| 全体 | 名称: Complex of inward-facing state of GltPh with L-aspartate and sodium ions in MSP1E3 nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: Complex of inward-facing state of GltPh with L-aspartate and sodi...
| 超分子 | 名称: Complex of inward-facing state of GltPh with L-aspartate and sodium ions in MSP1E3 nanodisc タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 134 KDa |
-分子 #1: Glutamate transporter homologue GltPh
| 分子 | 名称: Glutamate transporter homologue GltPh / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) |
| 分子量 | 理論値: 44.643918 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: (FME)GLYRKYIEY PVLQKILIGL ILGAIVGLIL GHYGYAHAVH TYVKPFGDLF VRLLCMLVMP IVFASLVVGA ASISPA RLG RVGVKIVVYY LLTSAFAVTL GIIMARLFNP GAGIHLAVGG QQFQPHQAPP LVHILLDIVP TNPFGALANG QVLPTIF FA ...文字列: (FME)GLYRKYIEY PVLQKILIGL ILGAIVGLIL GHYGYAHAVH TYVKPFGDLF VRLLCMLVMP IVFASLVVGA ASISPA RLG RVGVKIVVYY LLTSAFAVTL GIIMARLFNP GAGIHLAVGG QQFQPHQAPP LVHILLDIVP TNPFGALANG QVLPTIF FA IILGIAITYL MNSENEKVRK SAETLLDAIN GLAEAMYKIV NGVMQYAPIG VFALIAYVMA EQGVHVVGEL AKVTAAVY V GLTLQILLVY FVLLKIYGID PISFIKHAKD AMLTAFVTRS SSGTLPVTMR VAKEMGISEG IYSFTLPLGA TINMDGTAL YQGVATFFIA NALGSHLTVG QQLTIVLTAV LASIGTAGVP GAGAIMLCMV LHSVGLPLTD PNVAAAYAMI LGIDAILDMG RTMVNVTGD LTGTAIVAKT EGTLVPR |
-分子 #2: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 2 / コピー数: 9 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #3: ASPARTIC ACID
| 分子 | 名称: ASPARTIC ACID / タイプ: ligand / ID: 3 / コピー数: 3 / 式: ASP |
|---|---|
| 分子量 | 理論値: 133.103 Da |
| Chemical component information |  ChemComp-ASP: |
-分子 #4: MERCURY (II) ION
| 分子 | 名称: MERCURY (II) ION / タイプ: ligand / ID: 4 / コピー数: 3 / 式: HG |
|---|---|
| 分子量 | 理論値: 200.59 Da |
-分子 #5: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 5 / コピー数: 39 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 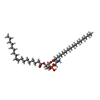 ChemComp-6OU: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 9 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 糖包埋 | 材質: ice |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 69.3 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
|---|---|
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.05 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 74233 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.05 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 74233 |
 ムービー
ムービー コントローラー
コントローラー


























