+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21442 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the full-length A39R/PlexinC1 complex | ||||||||||||||||||
 マップデータ マップデータ | Dimeric map | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of synapse pruning / semaphorin receptor binding / cerebellar climbing fiber to Purkinje cell synapse / Other semaphorin interactions / semaphorin-plexin signaling pathway involved in axon guidance / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / positive regulation of axonogenesis /  regulation of cell migration ...regulation of synapse pruning / semaphorin receptor binding / cerebellar climbing fiber to Purkinje cell synapse / Other semaphorin interactions / semaphorin-plexin signaling pathway involved in axon guidance / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / positive regulation of axonogenesis / regulation of cell migration ...regulation of synapse pruning / semaphorin receptor binding / cerebellar climbing fiber to Purkinje cell synapse / Other semaphorin interactions / semaphorin-plexin signaling pathway involved in axon guidance / semaphorin receptor complex / semaphorin receptor activity / negative regulation of cell adhesion / positive regulation of axonogenesis /  regulation of cell migration / regulation of cell shape / regulation of cell migration / regulation of cell shape /  細胞接着 / 細胞接着 /  signaling receptor binding / extracellular region / signaling receptor binding / extracellular region /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Ectromelia virus (エクトロメリアウイルス) / Ectromelia virus (エクトロメリアウイルス) /   Ectromelia virus (strain Moscow) (エクトロメリアウイルス) Ectromelia virus (strain Moscow) (エクトロメリアウイルス) | ||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Kuo Y-C / Chen H / Shang G / Uchikawa E / Tian H / Bai X / Zhang X | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Cryo-EM structure of the PlexinC1/A39R complex reveals inter-domain interactions critical for ligand-induced activation. 著者: Yi-Chun Kuo / Hua Chen / Guijun Shang / Emiko Uchikawa / Hui Tian / Xiao-Chen Bai / Xuewu Zhang /  要旨: Plexins are receptors for semaphorins that transduce signals for regulating neuronal development and other processes. Plexins are single-pass transmembrane proteins with multiple domains in both the ...Plexins are receptors for semaphorins that transduce signals for regulating neuronal development and other processes. Plexins are single-pass transmembrane proteins with multiple domains in both the extracellular and intracellular regions. Semaphorin activates plexin by binding to its extracellular N-terminal Sema domain, inducing the active dimer of the plexin intracellular region. The mechanism underlying this activation process of plexin is incompletely understood. We present cryo-electron microscopic structure of full-length human PlexinC1 in complex with the viral semaphorin mimic A39R. The structure shows that A39R induces a specific dimer of PlexinC1 where the membrane-proximal domains from the two PlexinC1 protomers are placed close to each other, poised to promote the active dimer of the intracellular region. This configuration is imposed by a distinct conformation of the PlexinC1 extracellular region, stabilized by inter-domain interactions among the Sema and membrane-proximal domains. Our mutational analyses support the critical role of this conformation in PlexinC1 activation. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21442.map.gz emd_21442.map.gz | 163.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21442-v30.xml emd-21442-v30.xml emd-21442.xml emd-21442.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21442.png emd_21442.png | 67.5 KB | ||
| その他 |  emd_21442_additional.map.gz emd_21442_additional.map.gz | 8.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21442 http://ftp.pdbj.org/pub/emdb/structures/EMD-21442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21442 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21442.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21442.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Dimeric map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
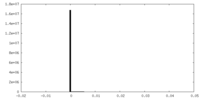
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Focused refined half map
| ファイル | emd_21442_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused refined half map | ||||||||||||
| 投影像・断面図 |
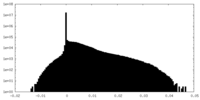
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex between A39R and PlexinC1
| 全体 | 名称: Complex between A39R and PlexinC1 |
|---|---|
| 要素 |
|
-超分子 #1: Complex between A39R and PlexinC1
| 超分子 | 名称: Complex between A39R and PlexinC1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 / 詳細: Full-length PlexinC1 in complex with A39R |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 400 KDa |
-超分子 #2: A39R dimer
| 超分子 | 名称: A39R dimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Ectromelia virus (エクトロメリアウイルス) Ectromelia virus (エクトロメリアウイルス) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: PlexinC1
| 超分子 | 名称: PlexinC1 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Semaphorin-like protein 139
| 分子 | 名称: Semaphorin-like protein 139 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Ectromelia virus (strain Moscow) (エクトロメリアウイルス) Ectromelia virus (strain Moscow) (エクトロメリアウイルス)株: Moscow |
| 分子量 | 理論値: 45.330195 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ELEIEWHKFE TSEEIISTYL IDDVLYTGVN GAVYTFSNNE LNKTGLTNNN NYITTSIKVE DTLVCGTNNG NPKCWKIDGS EDPKYRGRG YAPYQNSKVT IISHNECVLS DINISKEGIK RWRRFDGPCG YDLYTADNVI PKDGVRGAFV DKDGTYDKVY I LFTDTIDT ...文字列: ELEIEWHKFE TSEEIISTYL IDDVLYTGVN GAVYTFSNNE LNKTGLTNNN NYITTSIKVE DTLVCGTNNG NPKCWKIDGS EDPKYRGRG YAPYQNSKVT IISHNECVLS DINISKEGIK RWRRFDGPCG YDLYTADNVI PKDGVRGAFV DKDGTYDKVY I LFTDTIDT KRIVKIPYIA QMCLNDEGGP SSLSSHRWST FLKVELECDI DGRSYRQIIH SKAIKTDNDT ILYVFFDSPY SK SALCTYS MNAIKHSFST SKLGGYTKQL PSPAPGICLP AGKVVPHTTF DIIEQYNELD DIIKPLSQPI FEGPSGVKWF DIK EKENEH REYRIYFIKE NTIYSFDTKS KQTRSAQVDA RLFSVMVTSK PLFIADIGIG VGIPRMKKIL KMGTHHHHHH HH |
-分子 #2: Plexin-C1
| 分子 | 名称: Plexin-C1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 173.487 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ADEPVWRSEQ AIGAIAASQE DGVFVASGSC LDQLDYSLEH SLSRLYRDQA GNCTEPVSLA PPARPRPGSS FSKLLLPYRE GAAGLGGLL LTGWTFDRGA CEVRPLGNLS RNSLRNGTEV VSCHPQGSTA GVVYRAGRNN RWYLAVAATY VLPEPETASR C NPAASDHD ...文字列: ADEPVWRSEQ AIGAIAASQE DGVFVASGSC LDQLDYSLEH SLSRLYRDQA GNCTEPVSLA PPARPRPGSS FSKLLLPYRE GAAGLGGLL LTGWTFDRGA CEVRPLGNLS RNSLRNGTEV VSCHPQGSTA GVVYRAGRNN RWYLAVAATY VLPEPETASR C NPAASDHD TAIALKDTEG RSLATQELGR LKLCEGAGSL HFVDAFLWNG SIYFPYYPYN YTSGAATGWP SMARIAQSTE VL FQGQASL DCGHGHPDGR RLLLSSSLVE ALDVWAGVFS AAAGEGQERR SPTTTALCLF RMSEIQARAK RVSWDFKTAE SHC KEGDQP ERVQPIASST LIHSDLTSVY GTVVMNRTVL FLGTGDGQLL KVILGENLTS NCPEVIYEIK EETPVFYKLV PDPV KNIYI YLTAGKEVRR IRVANCNKHK SCSECLTATD PHCGWCHSLQ RCTFQGDCVH SENLENWLDI SSGAKKCPKI QIIRS SKEK TTVTMVGSFS PRHSKCMVKN VDSSRELCQN KSQPNRTCTC SIPTRATYKD VSVVNVMFSF GSWNLSDRFN FTNCSS LKE CPACVETGCA WCKSARRCIH PFTACDPSDY ERNQEQCPVA VEKTSGGGRP KENKGNRTNQ ALQVFYIKSI EPQKVST LG KSNVIVTGAN FTRASNITMI LKGTSTCDKD VIQVSHVLND THMKFSLPSS RKEMKDVCIQ FDGGNCSSVG SLSYIALP H CSLIFPATTW ISGGQNITMM GRNFDVIDNL IISHELKGNI NVSEYCVATY CGFLAPSLKS SKVRTNVTVK LRVQDTYLD CGTLQYREDP RFTGYRVESE VDTELEVKIQ KENDNFNISK KDIEITLFHG ENGQLNCSFE NITRNQDLTT ILCKIKGIKT ASTIANSSK KVRVKLGNLE LYVEQESVPS TWYFLIVLPV LLVIVIFAAV GVTRHKSKEL SRKQSQQLEL LESELRKEIR D GFAELQMD KLDVVDSFGT VPFLDYKHFA LRTFFPESGG FTHIFTEDMH NRDANDKNES LTALDALICN KSFLVTVIHT LE KQKNFSV KDRCLFASFL TIALQTKLVY LTSILEVLTR DLMEQCSNMQ PKLMLRRTES VVEKLLTNWM SVCLSGFLRE TVG EPFYLL VTTLNQKINK GPVDVITCKA LYTLNEDWLL WQVPEFSTVA LNVVFEKIPE NESADVCRNI SVNVLDCDTI GQAK EKIFQ AFLSKNGSPY GLQLNEIGLE LQMGTRQKEL LDIDSSSVIL EDGITKLNTI GHYEISNGST IKVFKKIANF TSDVE YSDD HCHLILPDSE AFQDVQGKRH RGKHKFKVKE MYLTKLLSTK VAIHSVLEKL FRSIWSLPNS RAPFAIKYFF DFLDAQ AEN KKITDPDVVH IWKTNSLPLR FWVNILKNPQ FVFDIKKTPH IDGCLSVIAQ AFMDAFSLTE QQLGKEAPTN KLLYAKD IP TYKEEVKSYY KAIRDLPPLS SSEMEEFLTQ ESKKHENEFN EEVALTEIYK YIVKYFDEIL NKLERERGLE EAQKQLLH V KVLFDEKKKC KWMGTSSGLE VLFQ |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 18 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 2.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 143750 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 143750 |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X