+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20763 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Katanin hexamer in the ring conformation with resolved protomer one in complex with substrate | |||||||||
 マップデータ マップデータ | Katanin hexamer in the ring conformation with resolved P1 in complex with substrate | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  katanin (カタニン) / microtubule-severing / MEI-1 / microtubule cytoskeleton / katanin (カタニン) / microtubule-severing / MEI-1 / microtubule cytoskeleton /  MOTOR PROTEIN (モータータンパク質) MOTOR PROTEIN (モータータンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic spindle elongation /  MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly / MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly /  katanin complex / meiotic spindle pole / katanin complex / meiotic spindle pole /  microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division ...negative regulation of meiotic spindle elongation / microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division ...negative regulation of meiotic spindle elongation /  MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly / MATH domain binding / striated muscle myosin thick filament assembly / meiotic spindle disassembly /  katanin complex / meiotic spindle pole / katanin complex / meiotic spindle pole /  microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division / meiotic spindle organization / microtubule-severing ATPase / microtubule severing ATPase activity / microtubule severing / female meiotic nuclear division / meiotic spindle organization /  紡錘体 / embryo development ending in birth or egg hatching / microtubule depolymerization / 紡錘体 / embryo development ending in birth or egg hatching / microtubule depolymerization /  isomerase activity / spindle / isomerase activity / spindle /  紡錘体 / microtubule cytoskeleton / 紡錘体 / microtubule cytoskeleton /  microtubule binding / microtubule binding /  protein phosphatase binding / protein phosphatase binding /  微小管 / molecular adaptor activity / 微小管 / molecular adaptor activity /  細胞分裂 / 細胞分裂 /  中心体 / 中心体 /  クロマチン / クロマチン /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Caenorhabditis elegans (センチュウ) / synthetic construct (人工物) Caenorhabditis elegans (センチュウ) / synthetic construct (人工物) | |||||||||
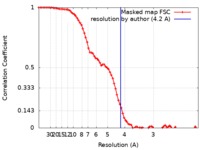
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.2 Å クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Zehr EA / Roll-Mecak A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Dev Cell / 年: 2020 ジャーナル: Dev Cell / 年: 2020タイトル: Katanin Grips the β-Tubulin Tail through an Electropositive Double Spiral to Sever Microtubules. 著者: Elena A Zehr / Agnieszka Szyk / Ewa Szczesna / Antonina Roll-Mecak /  要旨: The AAA ATPase katanin severs microtubules. It is critical in cell division, centriole biogenesis, and neuronal morphogenesis. Its mutation causes microcephaly. The microtubule templates katanin ...The AAA ATPase katanin severs microtubules. It is critical in cell division, centriole biogenesis, and neuronal morphogenesis. Its mutation causes microcephaly. The microtubule templates katanin hexamerization and activates its ATPase. The structural basis for these activities and how they lead to severing is unknown. Here, we show that β-tubulin tails are necessary and sufficient for severing. Cryoelectron microscopy (cryo-EM) structures reveal the essential tubulin tail glutamates gripped by a double spiral of electropositive loops lining the katanin central pore. Each spiral couples allosterically to the ATPase and binds alternating, successive substrate residues, with consecutive residues coordinated by adjacent protomers. This tightly couples tail binding, hexamerization, and ATPase activation. Hexamer structures in different states suggest an ATPase-driven, ratchet-like translocation of the tubulin tail through the pore. A disordered region outside the AAA core anchors katanin to the microtubule while the AAA motor exerts the forces that extract tubulin dimers and sever the microtubule. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20763.map.gz emd_20763.map.gz | 38.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20763-v30.xml emd-20763-v30.xml emd-20763.xml emd-20763.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20763_fsc.xml emd_20763_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20763.png emd_20763.png | 192.9 KB | ||
| Filedesc metadata |  emd-20763.cif.gz emd-20763.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20763 http://ftp.pdbj.org/pub/emdb/structures/EMD-20763 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20763 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20763 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20763.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20763.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Katanin hexamer in the ring conformation with resolved P1 in complex with substrate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Hexameric complex of C.elegans katanin bound to polyglutamate peptide
| 全体 | 名称: Hexameric complex of C.elegans katanin bound to polyglutamate peptide |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric complex of C.elegans katanin bound to polyglutamate peptide
| 超分子 | 名称: Hexameric complex of C.elegans katanin bound to polyglutamate peptide タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
| 分子量 | 理論値: 312 KDa |
-分子 #1: Meiotic spindle formation protein mei-1
| 分子 | 名称: Meiotic spindle formation protein mei-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号:  microtubule-severing ATPase microtubule-severing ATPase |
|---|---|
| 由来(天然) | 生物種:   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
| 分子量 | 理論値: 53.576336 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: GSFTMASMTG GQQMGRGSMN GDVQSVIRGY LERAQVAKTM SDAGRWNEAG DLLRQLMTDV KSCKISASNR DEHDARNTFL RALEANLKL VQQNVRDEDD LHEAMTRQSG SPEPPADPDV WSKPSPPLPS SSKFGATKKG VGAAGPRPRE ISKSTSSMST N PADVKPAN ...文字列: GSFTMASMTG GQQMGRGSMN GDVQSVIRGY LERAQVAKTM SDAGRWNEAG DLLRQLMTDV KSCKISASNR DEHDARNTFL RALEANLKL VQQNVRDEDD LHEAMTRQSG SPEPPADPDV WSKPSPPLPS SSKFGATKKG VGAAGPRPRE ISKSTSSMST N PADVKPAN PTQGILPQNS AGDSFDASAY DAYIVQAVRG TMATNTENTM SLDDIIGMHD VKQVLHEAVT LPLLVPEFFQ GL RSPWKAM VLAGPPGTGK TLIARAIASE SSSTFFTVSS TDLSSKWRGD SEKIVRLLFE LARFYAPSII FIDQIDTLGG QRG NSGEHE ASRRVKSEFL VQMDGSQNKF DSRRVFVLAA TNIPWELDEA LRRRFEKRIF IPLPDIDARK KLIEKSMEGT PKSD EINYD DLAARTEGFS GADVVSLCRT AAINVLRRYD TKSLRGGELT AAMESLKAEL VRNIDFEAAL QAVSPSAGPD TMLKC KEWC DSFGAM UniProtKB: Meiotic spindle formation protein mei-1 |
-分子 #2: Polyglutamate peptide
| 分子 | 名称: Polyglutamate peptide / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 1.567381 KDa |
| 配列 | 文字列: EEEEEEEEEE EE |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 5 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 279.15 K / 装置: LEICA EM GP / 詳細: Load 5 ul sample, wait 10 sec, blot 4.5 sec. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm 最大 デフォーカス(補正後): 2.8000000000000003 µm 最小 デフォーカス(補正後): 1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD  Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 93.0 K / 最高: 103.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7420 pixel / デジタル化 - サイズ - 縦: 7676 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 4 / 実像数: 5911 / 平均露光時間: 10.0 sec. / 平均電子線量: 73.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー