+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20540 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM map of NA-73 Fab in complex with N9 Shanghai2 | |||||||||
 マップデータ マップデータ | CryoEM map of NA-73 Fab in complex with N9 Shanghai | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報exo-alpha-(2->3)-sialidase activity / exo-alpha-(2->6)-sialidase activity / exo-alpha-(2->8)-sialidase activity /  ノイラミニダーゼ / ノイラミニダーゼ /  viral budding from plasma membrane / carbohydrate metabolic process / host cell plasma membrane / virion membrane / viral budding from plasma membrane / carbohydrate metabolic process / host cell plasma membrane / virion membrane /  生体膜 / 生体膜 /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Influenza A virus (A/environment/Shanghai/S1439/2013(H7N9)) (A型インフルエンザウイルス) / Influenza A virus (A/environment/Shanghai/S1439/2013(H7N9)) (A型インフルエンザウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
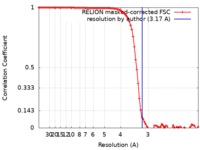
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.17 Å クライオ電子顕微鏡法 / 解像度: 3.17 Å | |||||||||
 データ登録者 データ登録者 | Ward AB / Turner HL / Zhu X | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2019 ジャーナル: Cell Host Microbe / 年: 2019タイトル: Structural Basis of Protection against H7N9 Influenza Virus by Human Anti-N9 Neuraminidase Antibodies. 著者: Xueyong Zhu / Hannah L Turner / Shanshan Lang / Ryan McBride / Sandhya Bangaru / Iuliia M Gilchuk / Wenli Yu / James C Paulson / James E Crowe / Andrew B Ward / Ian A Wilson /  要旨: Influenza virus neuraminidase (NA) is a major target for small-molecule antiviral drugs. Antibodies targeting the NA surface antigen could also inhibit virus entry and egress to provide host ...Influenza virus neuraminidase (NA) is a major target for small-molecule antiviral drugs. Antibodies targeting the NA surface antigen could also inhibit virus entry and egress to provide host protection. However, our understanding of the nature and range of target epitopes is limited because of a lack of human antibody structures with influenza neuraminidase. Here, we describe crystal and cryogenic electron microscopy (cryo-EM) structures of NAs from human-infecting avian H7N9 viruses in complex with five human anti-N9 antibodies, systematically defining several antigenic sites and antibody epitope footprints. These antibodies either fully or partially block the NA active site or bind to epitopes distant from the active site while still showing neuraminidase inhibition. The inhibition of antibodies to NAs was further analyzed by glycan array and solution-based NA activity assays. Together, these structural studies provide insights into protection by anti-NA antibodies and templates for the development of NA-based influenza virus vaccines and therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20540.map.gz emd_20540.map.gz | 31.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20540-v30.xml emd-20540-v30.xml emd-20540.xml emd-20540.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20540_fsc.xml emd_20540_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20540.png emd_20540.png | 137.2 KB | ||
| マスクデータ |  emd_20540_msk_1.map emd_20540_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_20540_half_map_1.map.gz emd_20540_half_map_1.map.gz emd_20540_half_map_2.map.gz emd_20540_half_map_2.map.gz | 58.8 MB 58.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20540 http://ftp.pdbj.org/pub/emdb/structures/EMD-20540 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20540 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20540 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20540.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20540.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of NA-73 Fab in complex with N9 Shanghai | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
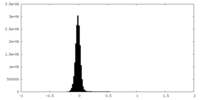
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20540_msk_1.map emd_20540_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
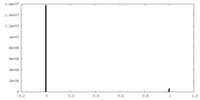
| 密度ヒストグラム |
-ハーフマップ: Halfmap A of CryoEM map of NA-73 Fab in complex with N9 Shanghai
| ファイル | emd_20540_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap A of CryoEM map of NA-73 Fab in complex with N9 Shanghai | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
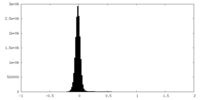
| 密度ヒストグラム |
-ハーフマップ: Halfmap B of CryoEM map of NA-73 Fab in complex with N9 Shanghai
| ファイル | emd_20540_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap B of CryoEM map of NA-73 Fab in complex with N9 Shanghai | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
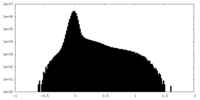
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NA-73 Fab in complex with N9 Shanghai2
| 全体 | 名称: NA-73 Fab in complex with N9 Shanghai2 |
|---|---|
| 要素 |
|
-超分子 #1: NA-73 Fab in complex with N9 Shanghai2
| 超分子 | 名称: NA-73 Fab in complex with N9 Shanghai2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: N9 Shanghai2
| 超分子 | 名称: N9 Shanghai2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A/environment/Shanghai/S1439/2013(H7N9)) (A型インフルエンザウイルス) Influenza A virus (A/environment/Shanghai/S1439/2013(H7N9)) (A型インフルエンザウイルス) |
-超分子 #3: NA-73 Fab
| 超分子 | 名称: NA-73 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Neuraminidase
| 分子 | 名称: Neuraminidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  ノイラミニダーゼ ノイラミニダーゼ |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A/environment/Shanghai/S1439/2013(H7N9)) (A型インフルエンザウイルス) Influenza A virus (A/environment/Shanghai/S1439/2013(H7N9)) (A型インフルエンザウイルス)株: A/environment/Shanghai/S1439/2013(H7N9) |
| 分子量 | 理論値: 48.214508 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: LKPGCNCSHS QPETTNTSQT IINNYYNETN ITNIQMEERT SRNFNNLTKG LCTINSWHIY GKDNAVRIGE SSDVLVTREP YVSCDPDEC RFYALSQGTT IRGKHSNGTI HDRSQYRALI SWPLSSPPTV YNSRVECIGW SSTSCHDGKS RMSICISGPN N NASAVVWY ...文字列: LKPGCNCSHS QPETTNTSQT IINNYYNETN ITNIQMEERT SRNFNNLTKG LCTINSWHIY GKDNAVRIGE SSDVLVTREP YVSCDPDEC RFYALSQGTT IRGKHSNGTI HDRSQYRALI SWPLSSPPTV YNSRVECIGW SSTSCHDGKS RMSICISGPN N NASAVVWY NRRPVAEINT WARNILRTQE SECVCHNGVC PVVFTDGSAT GPADTRIYYF KEGKILKWES LTGTAKHIEE CS CYGERTG ITCTCRDNWQ GSNRPVIQID PVAMTHTSQY ICSPVLTDNP RPNDPNIGKC NDPYPGNNNN GVKGFSYLDG ANT WLGRTI STASRSGYEM LKVPNALTDD RSKPIQGQTI VLNADWSGYS GSFMDYWAEG DCYRACFYVE LIRGRPKEDK VWWT SNSIV SMCSSTEFLG QWNWPDGAKI EYFL |
-分子 #2: NA-73 fragment antibody heavy chain
| 分子 | 名称: NA-73 fragment antibody heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.822797 KDa |
| 組換発現 | 生物種:  Cricetinae gen. sp. (ネズミ) Cricetinae gen. sp. (ネズミ) |
| 配列 | 文字列: QVQLEESGPG LVKPSETLSL TCTVSGYTIS SGYYWGWIRQ PPGKGLEWIG CNNHRGSSYY NPSLKSRVII SVDTTKNKFS LKLSSVTAA DTAVYYCARD PSFWSSTSRT SPYYYGMDVW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK D YFPEPVTV ...文字列: QVQLEESGPG LVKPSETLSL TCTVSGYTIS SGYYWGWIRQ PPGKGLEWIG CNNHRGSSYY NPSLKSRVII SVDTTKNKFS LKLSSVTAA DTAVYYCARD PSFWSSTSRT SPYYYGMDVW GQGTLVTVSS ASTKGPSVFP LAPSSKSTSG GTAALGCLVK D YFPEPVTV SWNSGALTSG VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSC |
-分子 #3: NA-73 fragment antibody light chain
| 分子 | 名称: NA-73 fragment antibody light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.792084 KDa |
| 組換発現 | 生物種:  Cricetinae gen. sp. (ネズミ) Cricetinae gen. sp. (ネズミ) |
| 配列 | 文字列: QSVLTQPPSA SGTPGQRVTI SCSGSSSNIG INTVNWYQQL PGTAPKLLIY SNNQRPSGVP DRFSGSKSGT SASLAISGLQ SEDEADYYC AAWDDNLNGW VFGGGTKLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP ...文字列: QSVLTQPPSA SGTPGQRVTI SCSGSSSNIG INTVNWYQQL PGTAPKLLIY SNNQRPSGVP DRFSGSKSGT SASLAISGLQ SEDEADYYC AAWDDNLNGW VFGGGTKLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.14 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 29000 Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X