+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20143 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the C4-symmetric TRPV2/RTx complex in amphipol resolved to 2.9 A | |||||||||
 マップデータ マップデータ | C4-symmetric TRPV2/RTx complex in amphipol resolved to 2.9 A | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ion channel (イオンチャネル) / ion channel (イオンチャネル) /  calcium channel (カルシウムチャネル) / calcium channel (カルシウムチャネル) /  TRP channel (TRPチャネル) / metal transport TRP channel (TRPチャネル) / metal transport | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報growth cone membrane / response to temperature stimulus / positive regulation of calcium ion import / positive regulation of axon extension / axonal growth cone /  calcium channel activity / calcium channel activity /  cell body / positive regulation of cold-induced thermogenesis / cell body / positive regulation of cold-induced thermogenesis /  細胞膜 / identical protein binding 細胞膜 / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Zubcevic L / Hsu AL | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Symmetry transitions during gating of the TRPV2 ion channel in lipid membranes. 著者: Lejla Zubcevic / Allen L Hsu / Mario J Borgnia / Seok-Yong Lee /  要旨: The Transient Receptor Potential Vanilloid 2 (TRPV2) channel is a member of the temperature-sensing thermoTRPV family. Recent advances in cryo-electronmicroscopy (cryo-EM) and X-ray crystallography ...The Transient Receptor Potential Vanilloid 2 (TRPV2) channel is a member of the temperature-sensing thermoTRPV family. Recent advances in cryo-electronmicroscopy (cryo-EM) and X-ray crystallography have provided many important insights into the gating mechanisms of thermoTRPV channels. Interestingly, crystallographic studies of ligand-dependent TRPV2 gating have shown that the TRPV2 channel adopts two-fold symmetric arrangements during the gating cycle. However, it was unclear if crystal packing forces played a role in stabilizing the two-fold symmetric arrangement of the channel. Here, we employ cryo-EM to elucidate the structure of full-length rabbit TRPV2 in complex with the agonist resiniferatoxin (RTx) in nanodiscs and amphipol. We show that RTx induces two-fold symmetric conformations of TRPV2 in both environments. However, the two-fold symmetry is more pronounced in the native-like lipid environment of the nanodiscs. Our data offers insights into a gating pathway in TRPV2 involving symmetry transitions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20143.map.gz emd_20143.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20143-v30.xml emd-20143-v30.xml emd-20143.xml emd-20143.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20143_fsc.xml emd_20143_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20143.png emd_20143.png | 80.6 KB | ||
| Filedesc metadata |  emd-20143.cif.gz emd-20143.cif.gz | 6.5 KB | ||
| その他 |  emd_20143_half_map_1.map.gz emd_20143_half_map_1.map.gz emd_20143_half_map_2.map.gz emd_20143_half_map_2.map.gz | 45.7 MB 45.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20143 http://ftp.pdbj.org/pub/emdb/structures/EMD-20143 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20143 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20143 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20143.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20143.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C4-symmetric TRPV2/RTx complex in amphipol resolved to 2.9 A | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 1
| ファイル | emd_20143_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_20143_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TRPV2
| 全体 | 名称: TRPV2 |
|---|---|
| 要素 |
|
-超分子 #1: TRPV2
| 超分子 | 名称: TRPV2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: TRPV2 in complex with RTx reconstituted into amphipol A8-35 |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
| 分子量 | 理論値: 300 KDa |
-分子 #1: TRPV2
| 分子 | 名称: TRPV2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) |
| 分子量 | 理論値: 88.72268 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MTSPSSPPAF RLETSDGGQD GAEVDKAQLG YGAGPPPMES RFQDEDRNFP PQIKVNLNYR KGAGASQPDL NRFDRDRLFN VVARGNPED LAGLLEYLRR TSKYLTDSEY TEGSTGKTCL MKAVLNLQDG VNACIQPLLE IDRDSGNPQP LVNAQCTDEY Y RGHSALHI ...文字列: MTSPSSPPAF RLETSDGGQD GAEVDKAQLG YGAGPPPMES RFQDEDRNFP PQIKVNLNYR KGAGASQPDL NRFDRDRLFN VVARGNPED LAGLLEYLRR TSKYLTDSEY TEGSTGKTCL MKAVLNLQDG VNACIQPLLE IDRDSGNPQP LVNAQCTDEY Y RGHSALHI AIEKRSLQCV KLLVENGANV HAKACGHFFQ KNQDTCFYFG ELPLSLAACT KQWDVVNYLL ENPHQPASLQ AQ DSLGNTV LHALVMIADD SAENSALVVR MYDGLLQAGA RLCPNVQLEG IPNLEGLTPL KLAAKEGKIE IFKHILQREF SAP CQSLSR KFTEWCYGPV RVSLYDLASV DSWEENSVLE IIAFHSRSPH RHRMVVLEPL NKLLQAKWDR LIPRFCFNFL CYLV YMLIF TAVAYHQPAL EKQGFPPLKA TAGNSMLLLG HILILLGGVY LLLGQLWYFW RRRLFIWISF MDSYSEILFL LQALL TVLS QVLCFLAIEW YLPLLVSSLA MGWTNLLYYT RGFQHTGIYS VMIEKVILRD LLRFLLVYLV FLFGFAVALV SLSREA QNS RTPAGPNATE VGQPGAGQED EAPPYRSILD ASLELFKFTI GMGELAFQEQ LRFRGVVLLL LLAYVLLTYV LLLNMLI AL MSETVNSVAT DSWSIWKLQK AISVLEMENG YWWCRRKKQR AGVMLTVGTR PDGSPDERWC FRVGEMNWAT WEQTLPRT L CEEPSGAAAP GVMKNPTPAS QRGEDSASEE DHLPLQLLQS RSNLEVLFQG PHHHHHHDYK DDDDK UniProtKB: Transient receptor potential cation channel subfamily V member 2 |
-分子 #2: resiniferatoxin
| 分子 | 名称: resiniferatoxin / タイプ: ligand / ID: 2 / コピー数: 4 / 式: 6EU |
|---|---|
| 分子量 | 理論値: 628.708 Da |
| Chemical component information | 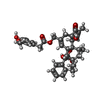 ChemComp-6EU: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 296 K / 装置: LEICA EM GP / 詳細: Blotted 3 seconds before plunging. | ||||||||||||
| 詳細 | TRPV2 in complex with RTx reconstituted into amphipol A8-35, monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源: OTHER |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 実像数: 1293 / 平均電子線量: 42.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X