+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20100 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | B41 SOSIP.664 in complex with the silent-face antibody SF12 and V3-targeting antibody 10-1074 | |||||||||
 マップデータ マップデータ | Final refined sharpened map used for model building and coordinate refinement | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報IgD immunoglobulin complex / IgM immunoglobulin complex / IgA immunoglobulin complex / IgE immunoglobulin complex / CD22 mediated BCR regulation / Fc epsilon receptor (FCERI) signaling / Classical antibody-mediated complement activation / Initial triggering of complement / IgG immunoglobulin complex /  immunoglobulin complex ...IgD immunoglobulin complex / IgM immunoglobulin complex / IgA immunoglobulin complex / IgE immunoglobulin complex / CD22 mediated BCR regulation / Fc epsilon receptor (FCERI) signaling / Classical antibody-mediated complement activation / Initial triggering of complement / IgG immunoglobulin complex / immunoglobulin complex ...IgD immunoglobulin complex / IgM immunoglobulin complex / IgA immunoglobulin complex / IgE immunoglobulin complex / CD22 mediated BCR regulation / Fc epsilon receptor (FCERI) signaling / Classical antibody-mediated complement activation / Initial triggering of complement / IgG immunoglobulin complex /  immunoglobulin complex / immunoglobulin mediated immune response / FCGR activation / Role of phospholipids in phagocytosis / Role of LAT2/NTAL/LAB on calcium mobilization / Scavenging of heme from plasma / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / immunoglobulin complex / immunoglobulin mediated immune response / FCGR activation / Role of phospholipids in phagocytosis / Role of LAT2/NTAL/LAB on calcium mobilization / Scavenging of heme from plasma / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering /  antigen binding / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / FCERI mediated Ca+2 mobilization / FCGR3A-mediated IL10 synthesis / host cell endosome membrane / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / antigen binding / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / FCERI mediated Ca+2 mobilization / FCGR3A-mediated IL10 synthesis / host cell endosome membrane / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers /  Regulation of Complement cascade / Cell surface interactions at the vascular wall / FCGR3A-mediated phagocytosis / FCERI mediated MAPK activation / B cell receptor signaling pathway / Regulation of actin dynamics for phagocytic cup formation / FCERI mediated NF-kB activation / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / clathrin-dependent endocytosis of virus by host cell / blood microparticle / Potential therapeutics for SARS / Regulation of Complement cascade / Cell surface interactions at the vascular wall / FCGR3A-mediated phagocytosis / FCERI mediated MAPK activation / B cell receptor signaling pathway / Regulation of actin dynamics for phagocytic cup formation / FCERI mediated NF-kB activation / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / clathrin-dependent endocytosis of virus by host cell / blood microparticle / Potential therapeutics for SARS /  獲得免疫系 / 獲得免疫系 /  viral protein processing / viral protein processing /  免疫応答 / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / 免疫応答 / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / structural molecule activity / virion attachment to host cell / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / structural molecule activity / virion attachment to host cell / host cell plasma membrane / virion membrane /  extracellular space / extracellular exosome / extracellular region / extracellular space / extracellular exosome / extracellular region /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
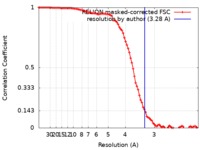
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.28 Å クライオ電子顕微鏡法 / 解像度: 3.28 Å | |||||||||
 データ登録者 データ登録者 | Barnes CO / Bjorkman PJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2019 ジャーナル: Immunity / 年: 2019タイトル: Broad and Potent Neutralizing Antibodies Recognize the Silent Face of the HIV Envelope. 著者: Till Schoofs / Christopher O Barnes / Nina Suh-Toma / Jovana Golijanin / Philipp Schommers / Henning Gruell / Anthony P West / Franziska Bach / Yu Erica Lee / Lilian Nogueira / Ivelin S ...著者: Till Schoofs / Christopher O Barnes / Nina Suh-Toma / Jovana Golijanin / Philipp Schommers / Henning Gruell / Anthony P West / Franziska Bach / Yu Erica Lee / Lilian Nogueira / Ivelin S Georgiev / Robert T Bailer / Julie Czartoski / John R Mascola / Michael S Seaman / M Juliana McElrath / Nicole A Doria-Rose / Florian Klein / Michel C Nussenzweig / Pamela J Bjorkman /   要旨: Broadly neutralizing antibodies (bNAbs) against HIV-1 envelope (Env) inform vaccine design and are potential therapeutic agents. We identified SF12 and related bNAbs with up to 62% neutralization ...Broadly neutralizing antibodies (bNAbs) against HIV-1 envelope (Env) inform vaccine design and are potential therapeutic agents. We identified SF12 and related bNAbs with up to 62% neutralization breadth from an HIV-infected donor. SF12 recognized a glycan-dominated epitope on Env's silent face and was potent against clade AE viruses, which are poorly covered by V3-glycan bNAbs. A 3.3Å cryo-EM structure of a SF12-Env trimer complex showed additional contacts to Env protein residues by SF12 compared with VRC-PG05, the only other known donor-derived silentface antibody, explaining SF12's increased neutralization breadth, potency, and resistance to Env mutation routes. Asymmetric binding of SF12 was associated with distinct N-glycan conformations across Env protomers, demonstrating intra-Env glycan heterogeneity. Administrating SF12 to HIV-1-infected humanized mice suppressed viremia and selected for viruses lacking the N448 glycan. Effective bNAbs can therefore be raised against HIV-1 Env's silent face, suggesting their potential for HIV-1 prevention, therapy, and vaccine development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20100.map.gz emd_20100.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20100-v30.xml emd-20100-v30.xml emd-20100.xml emd-20100.xml | 31.3 KB 31.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20100_fsc.xml emd_20100_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20100.png emd_20100.png | 171.2 KB | ||
| マスクデータ |  emd_20100_msk_1.map emd_20100_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_20100_additional.map.gz emd_20100_additional.map.gz emd_20100_half_map_1.map.gz emd_20100_half_map_1.map.gz emd_20100_half_map_2.map.gz emd_20100_half_map_2.map.gz | 48.9 MB 49.5 MB 49.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20100 http://ftp.pdbj.org/pub/emdb/structures/EMD-20100 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20100 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20100 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20100.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20100.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final refined sharpened map used for model building and coordinate refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20100_msk_1.map emd_20100_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
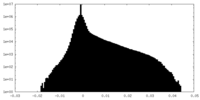
| 投影像・断面図 |
| ||||||||||||
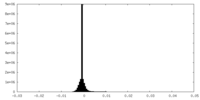
| 密度ヒストグラム |
-追加マップ: Final refined unsharpened map.
| ファイル | emd_20100_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final refined unsharpened map. | ||||||||||||
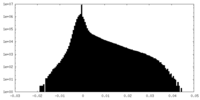
| 投影像・断面図 |
| ||||||||||||
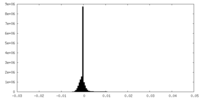
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_20100_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_20100_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex of B41 SOSIP.664 trimer with three SF12 Fabs and one 10-1...
+超分子 #1: Complex of B41 SOSIP.664 trimer with three SF12 Fabs and one 10-1...
+超分子 #2: B41 SOSIP.664 trimer
+超分子 #3: 10-1074 Fab
+超分子 #4: SF12 Fab
+分子 #1: Envelope glycoprotein gp41
+分子 #2: Envelope glycoprotein gp120
+分子 #3: 10-1074 Heavy Chain,10-1074 Heavy Chain
+分子 #4: 10-1074 Light Chain,10-1074 Light Chain
+分子 #5: SF12 Heavy Chain,SF12 Heavy Chain
+分子 #6: SF12 Light Chain,SF12 Light Chain
+分子 #14: 2-acetamido-2-deoxy-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: 0 blot force, 3 second blot time, 3 uL sample added to freshly glow-discharged grids. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 2732 / 平均露光時間: 8.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





































 Z
Z Y
Y X
X