+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20074 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Chimpanzee SIV Env trimeric ectodomain. | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane fusion involved in viral entry into host cell / host cell endosome membrane / membrane => GO:0016020 /  エンベロープ (ウイルス) / structural molecule activity / virion attachment to host cell / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / structural molecule activity / virion attachment to host cell / host cell plasma membrane / virion membrane /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.1 Å クライオ電子顕微鏡法 / 解像度: 4.1 Å | ||||||||||||
 データ登録者 データ登録者 | Pallesen J / Andrabi R / de Val N / Burton DR / Ward AB | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2019 ジャーナル: Cell Rep / 年: 2019タイトル: The Chimpanzee SIV Envelope Trimer: Structure and Deployment as an HIV Vaccine Template. 著者: Raiees Andrabi / Jesper Pallesen / Joel D Allen / Ge Song / Jinsong Zhang / Natalia de Val / Gavin Gegg / Katelyn Porter / Ching-Yao Su / Matthias Pauthner / Amanda Newman / Hilary Bouton- ...著者: Raiees Andrabi / Jesper Pallesen / Joel D Allen / Ge Song / Jinsong Zhang / Natalia de Val / Gavin Gegg / Katelyn Porter / Ching-Yao Su / Matthias Pauthner / Amanda Newman / Hilary Bouton-Verville / Fernando Garces / Ian A Wilson / Max Crispin / Beatrice H Hahn / Barton F Haynes / Laurent Verkoczy / Andrew B Ward / Dennis R Burton /   要旨: Epitope-targeted HIV vaccine design seeks to focus antibody responses to broadly neutralizing antibody (bnAb) sites by sequential immunization. A chimpanzee simian immunodeficiency virus (SIV) ...Epitope-targeted HIV vaccine design seeks to focus antibody responses to broadly neutralizing antibody (bnAb) sites by sequential immunization. A chimpanzee simian immunodeficiency virus (SIV) envelope (Env) shares a single bnAb site, the variable loop 2 (V2)-apex, with HIV, suggesting its possible utility in an HIV immunization strategy. Here, we generate a chimpanzee SIV Env trimer, MT145K, which displays selective binding to HIV V2-apex bnAbs and precursor versions, but no binding to other HIV specificities. We determine the structure of the MT145K trimer by cryo-EM and show that its architecture is remarkably similar to HIV Env. Immunization of an HIV V2-apex bnAb precursor Ab-expressing knockin mouse with the chimpanzee MT145K trimer induces HIV V2-specific neutralizing responses. Subsequent boosting with an HIV trimer cocktail induces responses that exhibit some virus cross-neutralization. Overall, the chimpanzee MT145K trimer behaves as expected from design both in vitro and in vivo and is an attractive potential component of a sequential immunization regimen to induce V2-apex bnAbs. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20074.map.gz emd_20074.map.gz | 78.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20074-v30.xml emd-20074-v30.xml emd-20074.xml emd-20074.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20074_fsc.xml emd_20074_fsc.xml | 9.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20074.png emd_20074.png | 162.7 KB | ||
| その他 |  emd_20074_half_map_1.map.gz emd_20074_half_map_1.map.gz emd_20074_half_map_2.map.gz emd_20074_half_map_2.map.gz | 65.4 MB 65.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20074 http://ftp.pdbj.org/pub/emdb/structures/EMD-20074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20074 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20074 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20074.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20074.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.02 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_20074_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
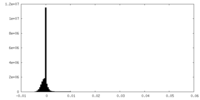
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_20074_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
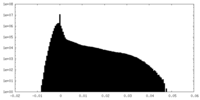
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Chimpanzee SIV Env trimeric ectodomain.
| 全体 | 名称: Chimpanzee SIV Env trimeric ectodomain. |
|---|---|
| 要素 |
|
-超分子 #1: Chimpanzee SIV Env trimeric ectodomain.
| 超分子 | 名称: Chimpanzee SIV Env trimeric ectodomain. / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 450 KDa |
-分子 #1: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 分子量 | 理論値: 51.708664 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ENWWVTVYYG VPVWREAKTT LFCASDAKSY STEAHNIWAT QACVPTDPTP QEVLLPNVTE EFNMWENYMV DQMQEDIISL WEQSLKPCV KLTPLCVTLT CNNPTNTSCT NSTDDRLGDM RNCSFNVTTE LRDKKRKVYS LFYVEDITAI GNNSTYRLIN C NTTAITQA ...文字列: ENWWVTVYYG VPVWREAKTT LFCASDAKSY STEAHNIWAT QACVPTDPTP QEVLLPNVTE EFNMWENYMV DQMQEDIISL WEQSLKPCV KLTPLCVTLT CNNPTNTSCT NSTDDRLGDM RNCSFNVTTE LRDKKRKVYS LFYVEDITAI GNNSTYRLIN C NTTAITQA CPKTSFEPIP IHYCAPAGFA LLKCNDIDYK GNETCKNVST VHCTHGIKPV ATTQLILNGS TADNQTVARI DP SENLAII QLKDPVKITC RRPGNNTRGQ IQIGPAMTFY NIENVVGDTR KAYCEINGTQ WAKALNETKE VLRNILRKNI SFM VPSGGD PEVTNHHFNC GGEFFYCNTS EIINITKINK TENMTIIPCR IRQIVNSWMR VGKGIFAPPI RGNITCTSNI TGML LEIHK NREDQGEDQD QNNTYVCLTG GNMKDIWRSE LYKYKIVEIQ PLGVAPTKCR RY |
-分子 #2: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 分子量 | 理論値: 17.17949 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LGALFLGFLG AAGSTMGAAS VVLTVQARQL LTGIVQQQNN LLRAPEAQQH LLQLSVWGIK QLQARVLAVE RYLRDQQLLG LWGCTGKTI CCTAVRWNKT WGNISDYQVI WNNYTWQQWD REVNNYTGLI YTLLEEANTQ QEKNEKELLE LD |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 24 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 10.0 sec. / 平均電子線量: 94.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X