+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1510 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of full-length Epac2 in complex with cyclic-AMP and Rap. | |||||||||
 マップデータ マップデータ | This is a map of the Epac2-cAMP-Rap1B complex. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GEF / GTPases / Epac / Rap /  cAMP (CAMP) cAMP (CAMP) | |||||||||
| 機能・相同性 |  : / intracellular anatomical structure / Ras guanine-nucleotide exchange factors catalytic domain / cAMP-mediated signaling : / intracellular anatomical structure / Ras guanine-nucleotide exchange factors catalytic domain / cAMP-mediated signaling 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Mus musculus (ハツカネズミ) / synthetic construct (人工物) Mus musculus (ハツカネズミ) / synthetic construct (人工物) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 23.0 Å ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Arias-Palomo E / Rehmann H / Hadders M / Schwede F / Bos JL / Llorca O | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2008 ジャーナル: Nature / 年: 2008タイトル: Structure of Epac2 in complex with a cyclic AMP analogue and RAP1B. 著者: Holger Rehmann / Ernesto Arias-Palomo / Michael A Hadders / Frank Schwede / Oscar Llorca / Johannes L Bos /  要旨: Epac proteins are activated by binding of the second messenger cAMP and then act as guanine nucleotide exchange factors for Rap proteins. The Epac proteins are involved in the regulation of cell ...Epac proteins are activated by binding of the second messenger cAMP and then act as guanine nucleotide exchange factors for Rap proteins. The Epac proteins are involved in the regulation of cell adhesion and insulin secretion. Here we have determined the structure of Epac2 in complex with a cAMP analogue (Sp-cAMPS) and RAP1B by X-ray crystallography and single particle electron microscopy. The structure represents the cAMP activated state of the Epac2 protein with the RAP1B protein trapped in the course of the exchange reaction. Comparison with the inactive conformation reveals that cAMP binding causes conformational changes that allow the cyclic nucleotide binding domain to swing from a position blocking the Rap binding site towards a docking site at the Ras exchange motif domain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1510.map.gz emd_1510.map.gz | 404.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1510-v30.xml emd-1510-v30.xml emd-1510.xml emd-1510.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1510.gif 1510.gif | 51 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1510 http://ftp.pdbj.org/pub/emdb/structures/EMD-1510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1510 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1510.map.gz / 形式: CCP4 / 大きさ: 422.9 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1510.map.gz / 形式: CCP4 / 大きさ: 422.9 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a map of the Epac2-cAMP-Rap1B complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
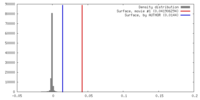
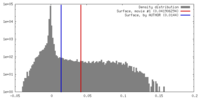
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Epac2-cAMP-Rap1B complex
| 全体 | 名称: Epac2-cAMP-Rap1B complex |
|---|---|
| 要素 |
|
-超分子 #1000: Epac2-cAMP-Rap1B complex
| 超分子 | 名称: Epac2-cAMP-Rap1B complex / タイプ: sample / ID: 1000 集合状態: One monomer of Epac2 binds one monomer of Rap1B in presence of cAMP Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 135 KDa |
-分子 #1: Rap1B
| 分子 | 名称: Rap1B / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Rap1B / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 18 KDa |
| 配列 | GO: intracellular anatomical structure / InterPro: INTERPRO: IPR003577 |
-分子 #2: Epac2
| 分子 | 名称: Epac2 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Epac2 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) / 別称: House Mouse Mus musculus (ハツカネズミ) / 別称: House Mouse |
| 分子量 | 理論値: 115 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換プラスミド: pGEX4T Escherichia coli (大腸菌) / 組換プラスミド: pGEX4T |
| 配列 | GO: cAMP-mediated signaling InterPro: Ras guanine-nucleotide exchange factors catalytic domain |
-分子 #3: Adenosine cyclic monophosphate
| 分子 | 名称: Adenosine cyclic monophosphate / タイプ: ligand / ID: 3 / Name.synonym: cAMP / 詳細: cAMP is the Epac2 activator. / 組換発現: No |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 329.21 Da |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 詳細: 50mM Tris-HCL, 100mM NaCl, 10mM CaCl2, 500 uM cAMP, 5% glycerol. |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Grids with adsorbed protein floated on 2% w/v uranyl formate for 60 seconds |
| グリッド | 詳細: 400 mesh Copper/Palladium grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1230 |
|---|---|
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.9 mm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2.9 mm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: OTHER |
| 詳細 | Microscope JEOL 1230 |
| 日付 | 2007年7月19日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 10.5 µm / ビット/ピクセル: 8 |
- 画像解析
画像解析
| CTF補正 | 詳細: Phase correction at the micrograph level |
|---|---|
| 最終 2次元分類 | クラス数: 233 |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN,Xmipp / 使用した粒子像数: 3442 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)