+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13522 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the actomyosin-V complex in the strong-ADP state (central 3er/2er) | ||||||||||||||||||
 マップデータ マップデータ | Sharpened map of the central three F-actin and two myosin-V-LC molecules filtered to local resolution | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報minus-end directed microfilament motor activity / unconventional myosin complex / insulin-responsive compartment / muscle myosin complex / muscle filament sliding /  myosin II complex / vesicle transport along actin filament / myosin II complex / vesicle transport along actin filament /  myosin complex / structural constituent of muscle / cytoskeletal motor activator activity ...minus-end directed microfilament motor activity / unconventional myosin complex / insulin-responsive compartment / muscle myosin complex / muscle filament sliding / myosin complex / structural constituent of muscle / cytoskeletal motor activator activity ...minus-end directed microfilament motor activity / unconventional myosin complex / insulin-responsive compartment / muscle myosin complex / muscle filament sliding /  myosin II complex / vesicle transport along actin filament / myosin II complex / vesicle transport along actin filament /  myosin complex / structural constituent of muscle / cytoskeletal motor activator activity / microfilament motor activity / myosin complex / structural constituent of muscle / cytoskeletal motor activator activity / microfilament motor activity /  tropomyosin binding / tropomyosin binding /  myosin heavy chain binding / mesenchyme migration / myosin heavy chain binding / mesenchyme migration /  troponin I binding / actin filament bundle / filamentous actin / actin filament bundle assembly / skeletal muscle thin filament assembly / cytoskeletal motor activity / striated muscle thin filament / skeletal muscle myofibril / actin monomer binding / Smooth Muscle Contraction / skeletal muscle fiber development / skeletal muscle tissue development / troponin I binding / actin filament bundle / filamentous actin / actin filament bundle assembly / skeletal muscle thin filament assembly / cytoskeletal motor activity / striated muscle thin filament / skeletal muscle myofibril / actin monomer binding / Smooth Muscle Contraction / skeletal muscle fiber development / skeletal muscle tissue development /  stress fiber / stress fiber /  titin binding / vesicle-mediated transport / actin filament polymerization / titin binding / vesicle-mediated transport / actin filament polymerization /  filopodium / filopodium /  muscle contraction / actin filament organization / muscle contraction / actin filament organization /  マイクロフィラメント / protein localization to plasma membrane / マイクロフィラメント / protein localization to plasma membrane /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / cellular response to insulin stimulus / calcium-dependent protein binding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / cellular response to insulin stimulus / calcium-dependent protein binding /  actin filament binding / actin filament binding /  マイクロフィラメント / マイクロフィラメント /  lamellipodium / lamellipodium /  cell body / vesicle / cell body / vesicle /  calmodulin binding / calmodulin binding /  hydrolase activity / protein domain specific binding / hydrolase activity / protein domain specific binding /  ゴルジ体 / ゴルジ体 /  calcium ion binding / positive regulation of gene expression / magnesium ion binding / extracellular exosome / calcium ion binding / positive regulation of gene expression / magnesium ion binding / extracellular exosome /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Gallus gallus (ニワトリ) / Gallus gallus (ニワトリ) /   Rabbit (ウサギ) / Rabbit (ウサギ) /   Amanita phalloides (タマゴテングタケ) Amanita phalloides (タマゴテングタケ) | ||||||||||||||||||
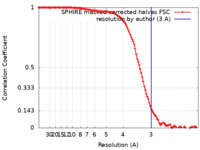
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.0 Å クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Pospich S / Sweeney HL / Houdusse A / Raunser S | ||||||||||||||||||
| 資金援助 |  ドイツ, European Union, ドイツ, European Union,  フランス, フランス,  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: High-resolution structures of the actomyosin-V complex in three nucleotide states provide insights into the force generation mechanism. 著者: Sabrina Pospich / H Lee Sweeney / Anne Houdusse / Stefan Raunser /    要旨: The molecular motor myosin undergoes a series of major structural transitions during its force-producing motor cycle. The underlying mechanism and its coupling to ATP hydrolysis and actin binding are ...The molecular motor myosin undergoes a series of major structural transitions during its force-producing motor cycle. The underlying mechanism and its coupling to ATP hydrolysis and actin binding are only partially understood, mostly due to sparse structural data on actin-bound states of myosin. Here, we report 26 high-resolution cryo-EM structures of the actomyosin-V complex in the strong-ADP, rigor, and a previously unseen post-rigor transition state that binds the ATP analog AppNHp. The structures reveal a high flexibility of myosin in each state and provide valuable insights into the structural transitions of myosin-V upon ADP release and binding of AppNHp, as well as the actomyosin interface. In addition, they show how myosin is able to specifically alter the structure of F-actin. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13522.map.gz emd_13522.map.gz | 2.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13522-v30.xml emd-13522-v30.xml emd-13522.xml emd-13522.xml | 25.8 KB 25.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13522_fsc.xml emd_13522_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13522.png emd_13522.png | 108.1 KB | ||
| マスクデータ |  emd_13522_msk_1.map emd_13522_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13522_additional_1.map.gz emd_13522_additional_1.map.gz emd_13522_additional_2.map.gz emd_13522_additional_2.map.gz emd_13522_half_map_1.map.gz emd_13522_half_map_1.map.gz emd_13522_half_map_2.map.gz emd_13522_half_map_2.map.gz | 3.3 MB 25.6 MB 59.8 MB 59.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13522 http://ftp.pdbj.org/pub/emdb/structures/EMD-13522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13522 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13522 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7pm6MC  7pltC  7pluC  7plvC  7plwC  7plxC  7plyC  7plzC  7pm0C  7pm1C  7pm2C  7pm3C  7pm5C  7pm7C  7pm8C  7pm9C  7pmaC  7pmbC  7pmcC  7pmdC  7pmeC  7pmfC  7pmgC  7pmhC  7pmiC  7pmjC  7pmlC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13522.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13522.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of the central three F-actin and two myosin-V-LC molecules filtered to local resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13522_msk_1.map emd_13522_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
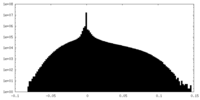
| 密度ヒストグラム |
-追加マップ: Sharpened map of the central three F-actin and...
| ファイル | emd_13522_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of the central three F-actin and two myosin-V-LC molecules filtered to nominal resolution | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
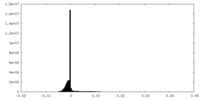
| 密度ヒストグラム |
-追加マップ: Denoised map of the central three F-actin and...
| ファイル | emd_13522_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Denoised map of the central three F-actin and two myosin-V-LC molecules (LAFTER) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
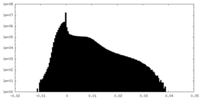
| 密度ヒストグラム |
-ハーフマップ: Half map of full filament
| ファイル | emd_13522_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of full filament | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
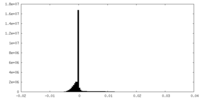
| 密度ヒストグラム |
-ハーフマップ: Half map of full filament
| ファイル | emd_13522_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of full filament | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Actomyosin-V complex in the strong-ADP state
| 全体 | 名称: Actomyosin-V complex in the strong-ADP state ミオフィラメント ミオフィラメント |
|---|---|
| 要素 |
|
-超分子 #1: Actomyosin-V complex in the strong-ADP state
| 超分子 | 名称: Actomyosin-V complex in the strong-ADP state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Aged ADP-bound F-actin stabilized with phalloidin and decorated with myosin-Va-LC in the strong-ADP state (Mg2+-ADP) |
|---|
-分子 #1: Myosin light chain 6B
| 分子 | 名称: Myosin light chain 6B / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 17.090277 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MIEFNKDQLE EFKEAFELFD RVGDGKILYS QCGDVMRALG QNPTNAEVLK VLGNPKSDEL KSRRVDFETF LPMLQAVAKN RGQGTYEDY LEGFRVFDKE GNGKVMGAEL RHVLTTLGEK MTEEEVETVL AGHEDSNGCI NYEAFLKHIL SV |
-分子 #2: Unconventional myosin-Va
| 分子 | 名称: Unconventional myosin-Va / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) |
| 分子量 | 理論値: 91.363953 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAASELYTKY ARVWIPDPEE VWKSAELLKD YKPGDKVLQL RLEEGKDLEY CLDPKTKELP PLRNPDILVG ENDLTALSYL HEPAVLHNL KVRFIDSKLI YTYCGIVLVA INPYEQLPIY GEDIINAYSG QNMGDMDPHI FAVAEEAYKQ MARDERNQSI I VSGESGAG ...文字列: MAASELYTKY ARVWIPDPEE VWKSAELLKD YKPGDKVLQL RLEEGKDLEY CLDPKTKELP PLRNPDILVG ENDLTALSYL HEPAVLHNL KVRFIDSKLI YTYCGIVLVA INPYEQLPIY GEDIINAYSG QNMGDMDPHI FAVAEEAYKQ MARDERNQSI I VSGESGAG KTVSAKYAMR YFATVSGSAS EANVEEKVLA SNPIMESIGN AKTTRNDNSS RFGKYIEIGF DKRYRIIGAN MR TYLLEKS RVVFQAEEER NYHIFYQLCA SAALPEFKTL RLGNANYFHY TKQGGSPVID GIDDAKEMVN TRQACTLLGI SDS YQMGIF RILAGILHLG NVEFASRDSD SCAIPPKHDP LTIFCDLMGV DYEEMAHWLC HRKLATATET YIKPISKLHA INAR DALAK HIYANLFNWI VDHVNKALHS TVKQHSFIGV LDIYGFETFE INSFEQFCIN YANEKLQQQF NMHVFKLEQE EYMKE QIPW TLIDFYDNQP CINLIEAKMG VLDLLDEECK MPKGSDDTWA QKLYNTHLNK CALFEKPRLS NKAFIIKHFA DKVEYQ CEG FLEKNKDTVY EEQIKVLKSS KKFKLLPELF QDEEKAISPT SATPSGRVPL SRTPVKPAKA RPGQTSKEHK KTVGHQF RN SLHLLMETLN ATTPHYVRCI KPNDFKFPFT FDEKRAVQQL RACGVLETIR ISAAGFPSRW TYQEFFSRYR VLMKQKDV L SDRKQTCKNV LEKLILDKDK YQFGKTKIFF RAGQVAYLEK IRADKLRAAC IRIQKTIRGW LMRKKYMRMR R |
-分子 #3: Actin, alpha skeletal muscle
| 分子 | 名称: Actin, alpha skeletal muscle / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rabbit (ウサギ) Rabbit (ウサギ) |
| 分子量 | 理論値: 42.109973 KDa |
| 配列 | 文字列: MCDEDETTAL VCDNGSGLVK AGFAGDDAPR AVFPSIVGRP RHQGVMVGMG QKDSYVGDEA QSKRGILTLK YPIE(HIC)G IIT NWDDMEKIWH HTFYNELRVA PEEHPTLLTE APLNPKANRE KMTQIMFETF NVPAMYVAIQ AVLSLYASGR TTGIVLD SG ...文字列: MCDEDETTAL VCDNGSGLVK AGFAGDDAPR AVFPSIVGRP RHQGVMVGMG QKDSYVGDEA QSKRGILTLK YPIE(HIC)G IIT NWDDMEKIWH HTFYNELRVA PEEHPTLLTE APLNPKANRE KMTQIMFETF NVPAMYVAIQ AVLSLYASGR TTGIVLD SG DGVTHNVPIY EGYALPHAIM RLDLAGRDLT DYLMKILTER GYSFVTTAER EIVRDIKEKL CYVALDFENE MATAASSS S LEKSYELPDG QVITIGNERF RCPETLFQPS FIGMESAGIH ETTYNSIMKC DIDIRKDLYA NNVMSGGTTM YPGIADRMQ KEITALAPST MKIKIIAPPE RKYSVWIGGS ILASLSTFQQ MWITKQEYDE AGPSIVHRKC F |
-分子 #4: Phalloidin
| 分子 | 名称: Phalloidin / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Amanita phalloides (タマゴテングタケ) Amanita phalloides (タマゴテングタケ) |
| 分子量 | 理論値: 808.899 Da |
| 配列 | 文字列: W(EEP)A(DTH)C(HYP)A |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 5 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 286 K / 装置: FEI VITROBOT MARK III / 詳細: On grid decoration. |
| 詳細 | Rise 27.8 A, Twist -167.3 degrees |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 5908 / 平均露光時間: 15.0 sec. / 平均電子線量: 82.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














































 Z
Z Y
Y X
X