+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10837 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rix1-Rea1 pre-60S particle - Rea1, body 3 (rigid body refinement, composite structure of Rea1 ring and tail) | |||||||||
 マップデータ マップデータ | Local resolution filtered composite map of the Rea1 Ring and Tail of the nucleoplasmic Rix1-Rea1 pre-60S particle | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein-RNA complex remodeling /  regulation of ribosomal subunit export from nucleus / Antigen processing: Ubiquitination & Proteasome degradation / preribosome, large subunit precursor / regulation of ribosomal subunit export from nucleus / Antigen processing: Ubiquitination & Proteasome degradation / preribosome, large subunit precursor /  ribosomal large subunit export from nucleus / ribosomal large subunit export from nucleus /  ribosomal large subunit assembly / rRNA processing / ribosomal large subunit assembly / rRNA processing /  核小体 / 核小体 /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア ...protein-RNA complex remodeling / ミトコンドリア ...protein-RNA complex remodeling /  regulation of ribosomal subunit export from nucleus / Antigen processing: Ubiquitination & Proteasome degradation / preribosome, large subunit precursor / regulation of ribosomal subunit export from nucleus / Antigen processing: Ubiquitination & Proteasome degradation / preribosome, large subunit precursor /  ribosomal large subunit export from nucleus / ribosomal large subunit export from nucleus /  ribosomal large subunit assembly / rRNA processing / ribosomal large subunit assembly / rRNA processing /  核小体 / 核小体 /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  ATP binding / ATP binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) / Saccharomyces cerevisiae (パン酵母) /   Baker's yeast (パン酵母) Baker's yeast (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.2 Å クライオ電子顕微鏡法 / 解像度: 4.2 Å | |||||||||
 データ登録者 データ登録者 | Kater L / Beckmann R | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2020 ジャーナル: Mol Cell / 年: 2020タイトル: Construction of the Central Protuberance and L1 Stalk during 60S Subunit Biogenesis. 著者: Lukas Kater / Valentin Mitterer / Matthias Thoms / Jingdong Cheng / Otto Berninghausen / Roland Beckmann / Ed Hurt /  要旨: Ribosome assembly is driven by numerous assembly factors, including the Rix1 complex and the AAA ATPase Rea1. These two assembly factors catalyze 60S maturation at two distinct states, triggering ...Ribosome assembly is driven by numerous assembly factors, including the Rix1 complex and the AAA ATPase Rea1. These two assembly factors catalyze 60S maturation at two distinct states, triggering poorly understood large-scale structural transitions that we analyzed by cryo-electron microscopy. Two nuclear pre-60S intermediates were discovered that represent previously unknown states after Rea1-mediated removal of the Ytm1-Erb1 complex and reveal how the L1 stalk develops from a pre-mature nucleolar to a mature-like nucleoplasmic state. A later pre-60S intermediate shows how the central protuberance arises, assisted by the nearby Rix1-Rea1 machinery, which was solved in its pre-ribosomal context to molecular resolution. This revealed a Rix1-Ipi3 tetramer anchored to the pre-60S via Ipi1, strategically positioned to monitor this decisive remodeling. These results are consistent with a general underlying principle that temporarily stabilized immature RNA domains are successively remodeled by assembly factors, thereby ensuring failsafe assembly progression. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10837.map.gz emd_10837.map.gz | 4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10837-v30.xml emd-10837-v30.xml emd-10837.xml emd-10837.xml | 23.5 KB 23.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10837.png emd_10837.png | 66.2 KB | ||
| その他 |  emd_10837_additional_1.map.gz emd_10837_additional_1.map.gz emd_10837_additional_2.map.gz emd_10837_additional_2.map.gz | 37.9 MB 36.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10837 http://ftp.pdbj.org/pub/emdb/structures/EMD-10837 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10837 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10837 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10837.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10837.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution filtered composite map of the Rea1 Ring and Tail of the nucleoplasmic Rix1-Rea1 pre-60S particle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Local resolution filtered map of the Tail of...
| ファイル | emd_10837_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
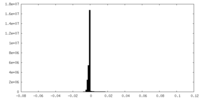
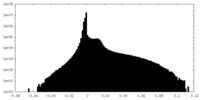
| 注釈 | Local resolution filtered map of the Tail of Rea1 of the nucleoplasmic Rix1-Rea1 pre-60S particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local resolution filtered map of the Ring of...
| ファイル | emd_10837_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
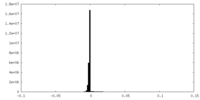
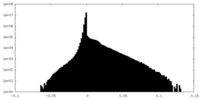
| 注釈 | Local resolution filtered map of the Ring of Rea1 of the nucleoplasmic Rix1-Rea1 pre-60S particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rix1-Rea1 pre-60S assembly particle - Rea1 with the Rsa4-UBL domain
| 全体 | 名称: Rix1-Rea1 pre-60S assembly particle - Rea1 with the Rsa4-UBL domain |
|---|---|
| 要素 |
|
-超分子 #1: Rix1-Rea1 pre-60S assembly particle - Rea1 with the Rsa4-UBL domain
| 超分子 | 名称: Rix1-Rea1 pre-60S assembly particle - Rea1 with the Rsa4-UBL domain タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Rix1-TAP Flag-Rea1 derived pre-60S assembly complex |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-分子 #1: Midasin
| 分子 | 名称: Midasin / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) Baker's yeast (パン酵母) |
| 分子量 | 理論値: 559.951438 KDa |
| 配列 | 文字列: MSQDRILLDL DVVNQRLILF NSAFPSDAIE APFHFSNKES TSENLDNLAG TILHSRSITG HVFLYKHIFL EIVARWIKDS KKKDYVLVI EKLASIITIF PVAMPLIEDY LDKENDHFIT ILQNPSTQKD SDMFKILLAY YRLLYHNKEV FARFIQPDIL Y QLVDLLTK ...文字列: MSQDRILLDL DVVNQRLILF NSAFPSDAIE APFHFSNKES TSENLDNLAG TILHSRSITG HVFLYKHIFL EIVARWIKDS KKKDYVLVI EKLASIITIF PVAMPLIEDY LDKENDHFIT ILQNPSTQKD SDMFKILLAY YRLLYHNKEV FARFIQPDIL Y QLVDLLTK EQENQVVIFL ALKVLSLYLD MGEKTLNDML DTYIKSRDSL LGHFEGDSGI DYSFLELNEA KRCANFSKLP SV PECFTIE KKSSYFIIEP QDLSTKVASI CGVIVPKVHT IHDKVFYPLT FVPTHKTVSS LRQLGRKIQN STPIMLIGKA GSG KTFLIN ELSKYMGCHD SIVKIHLGEQ TDAKLLIGTY TSGDKPGTFE WRAGVLATAV KEGRWVLIED IDKAPTDVLS ILLS LLEKR ELTIPSRGET VKAANGFQLI STVRINEDHQ KDSSNKIYNL NMIGMRIWNV IELEEPSEED LTHILAQKFP ILTNL IPKL IDSYKNVKSI YMNTKFISLN KGAHTRVVSV RDLIKLCERL DILFKNNGIN KPDQLIQSSV YDSIFSEAAD CFAGAI GEF KALEPIIQAI GESLDIASSR ISLFLTQHVP TLENLDDSIK IGRAVLLKEK LNIQKKSMNS TLFAFTNHSL RLMEQIS VC IQMTEPVLLV GETGTGKTTV VQQLAKMLAK KLTVINVSQQ TETGDLLGGY KPVNSKTVAV PIQENFETLF NATFSLKK N EKFHKMLHRC FNKNQWKNVV KLWNEAYKMA QSILKITNTE NENENAKKKK RRLNTHEKKL LLDKWADFND SVKKFEAQS SSIENSFVFN FVEGSLVKTI RAGEWLLLDE VNLATADTLE SISDLLTEPD SRSILLSEKG DAEPIKAHPD FRIFACMNPA TDVGKRDLP MGIRSRFTEI YVHSPERDIT DLLSIIDKYI GKYSVSDEWV GNDIAELYLE AKKLSDNNTI VDGSNQKPHF S IRTLTRTL LYVTDIIHIY GLRRSLYDGF CMSFLTLLDQ KSEAILKPVI EKFTLGRLKN VKSIMSQTPP SPGPDYVQFK HY WMKKGPN TIQEQAHYII TPFVEKNMMN LVRATSGKRF PVLIQGPTSS GKTSMIKYLA DITGHKFVRI NNHEHTDLQE YLG TYVTDD TGKLSFKEGV LVEALRKGYW IVLDELNLAP TDVLEALNRL LDDNRELFIP ETQEVVHPHP DFLLFATQNP PGIY GGRKI LSRAFRNRFL ELHFDDIPQD ELEIILRERC QIAPSYAKKI VEVYRQLSIE RSASRLFEQK NSFATLRDLF RWALR DAVG YEQLAASGYM LLAERCRTPQ EKVTVKKTLE KVMKVKLDMD QYYASLEDKS LEAIGSVTWT KGMRRLSVLV SSCLKN KEP VLLVGETGCG KTTICQLLAQ FMGRELITLN AHQNTETGDI LGAQRPVRNR SEIQYKLIKS LKTALNIAND QDVDLKE LL QLYSKSDNKN IAEDVQLEIQ KLRDSLNVLF EWSDGPLIQA MRTGNFFLLD EISLADDSVL ERLNSVLEPE RSLLLAEQ G SSDSLVTASE NFQFFATMNP GGDYGKKELS PALRNRFTEI WVPSMEDFND VNMIVSSRLL EDLKDLANPI VKFSEWFGK KLGGGNATSG VISLRDILAW VEFINKVFPK IQNKSTALIQ GASMVFIDAL GTNNTAYLAE NENDLKSLRT ECIIQLLKLC GDDLELQQI ETNEIIVTQD ELQVGMFKIP RFPDAQSSSF NLTAPTTASN LVRVVRAMQV HKPILLEGSP GVGKTSLITA L ANITGNKL TRINLSEQTD LVDLFGADAP GERSGEFLWH DAPFLRAMKK GEWVLLDEMN LASQSVLEGL NACLDHRGEA YI PELDISF SCHPNFLVFA AQNPQYQGGG RKGLPKSFVN RFSVVFIDML TSDDLLLIAK HLYPSIEPDI IAKMIKLMST LED QVCKRK LWGNSGSPWE FNLRDTLRWL KLLNQYSICE DVDVFDFVDI IVKQRFRTIS DKNKAQLLIE DIFGKFSTKE NFFK LTEDY VQINNEVALR NPHYRYPITQ NLFPLECNVA VYESVLKAIN NNWPLVLVGP SNSGKTETIR FLASILGPRV DVFSM NSDI DSMDILGGYE QVDLTRQISY ITEELTNIVR EIISMNMKLS PNATAIMEGL NLLKYLLNNI VTPEKFQDFR NRFNRF FSH LEGHPLLKTM SMNIEKMTEI ITKEASVKFE WFDGMLVKAV EKGHWLILDN ANLCSPSVLD RLNSLLEIDG SLLINEC SQ EDGQPRVLKP HPNFRLFLTM DPKYGELSRA MRNRGVEIYI DELHSRSTAF DRLTLGFELG ENIDFVSIDD GIKKIKLN E PDMSIPLKHY VPSYLSRPCI FAQVHDILLL SDEEPIEESL AAVIPISHLG EVGKWANNVL NCTEYSEKKI AERLYVFIT FLTDMGVLEK INNLYKPANL KFQKALGLHD KQLTEETVSL TLNEYVLPTV SKYSDKIKSP ESLYLLSSLR LLLNSLNALK LINEKSTHG KIDELTYIEL SAAAFNGRHL KNIPRIPIFC ILYNILTVMS ENLKTESLFC GSNQYQYYWD LLVIVIAALE T AVTKDEAR LRVYKELIDS WIASVKSKSD IEITPFLNIN LEFTDVLQLS RGHSITLLWD IFRKNYPTTS NSWLAFEKLI NL SEKFDKV RLLQFSESYN SIKDLMDVFR LLNDDVLNNK LSEFNLLLSK LEDGINELEL ISNKFLNKRK HYFADEFDNL IRY TFSVDT AELIKELAPA SSLATQKLTK LITNKYNYPP IFDVLWTEKN AKLTSFTSTI FSSQFLEDVV RKSNNLKSFS GNQI KQSIS DAELLLSSTI KCSPNLLKSQ MEYYKNMLLS WLRKVIDIHV GGDCLKLTLK ELCSLIEEKT ASETRVTFAE YIFPA LDLA ESSKSLEELG EAWITFGTGL LLLFVPDSPY DPAIHDYVLY DLFLKTKTFS QNLMKSWRNV RKVISGDEEI FTEKLI NTI SDDDAPQSPR VYRTGMSIDS LFDEWMAFLS STMSSRQIKE LVSSYKCNSD QSDRRLEMLQ QNSAHFLNRL ESGYSKF AD LNDILAGYIY SINFGFDLLK LQKSKDRASF QISPLWSMDP INISCAENVL SAYHELSRFF KKGDMEDTSI EKVLMYFL T LFKFHKRDTN LLEIFEAALY TLYSRWSVRR FRQEQEENEK SNMFKFNDNS DDYEADFRKL FPDYEDTALV TNEKDISSP ENLDDIYFKL ADTYISVFDK DHDANFSSEL KSGAIITTIL SEDLKNTRIE ELKSGSLSAV INTLDAETQS FKNTEVFGNI DFYHDFSIP EFQKAGDIIE TVLKSVLKLL KQWPEHATLK ELYRVSQEFL NYPIKTPLAR QLQKIEQIYT YLAEWEKYAS S EVSLNNTV KLITDLIVSW RKLELRTWKG LFNSEDAKTR KSIGKWWFYL YESIVISNFV SEKKETAPNA TLLVSSLNLF FS KSTLGEF NARLDLVKAF YKHIQLIGLR SSKIAGLLHN TIKFYYQFKP LIDERITNGK KSLEKEIDDI ILLASWKDVN VDA LKQSSR KSHNNLYKIV RKYRDLLNGD AKTIIEAGLL YSNENKLKLP TLKQHFYEDP NLEASKNLVK EISTWSMRAA PLRN IDTVA SNMDSYLEKI SSQEFPNFAD LASDFYAEAE RLRKETPNVY TKENKKRLAY LKTQKSKLLG DALKELRRIG LKVNF REDI QKVQSSTTTI LANIAPFNNE YLNSSDAFFF KILDLLPKLR SAASNPSDDI PVAAIERGMA LAQSLMFSLI TVRHPL SEF TNDYCKINGM MLDLEHFTCL KGDIVHSSLK ANVDNVRLFE KWLPSLLDYA AQTLSVISKY SATSEQQKIL LDAKSTL SS FFVHFNSSRI FDSSFIESYS RFELFINELL KKLENAKETG NAFVFDIIIE WIKANKGGPI KKEQKRGPSV EDVEQAFR R TFTSIILSFQ KVIGDGIESI SETDDNWLSA SFKKVMVNVK LLRSSVVSKN IETALSLLKD FDFTTTESIY VKSVISFTL PVITRYYNAM TVVLERSRIY YTNTSRGMYI LSTILHSLAK NGFCSPQPPS EEVDDKNLQE GTGLGDGEGA QNNNKDVEQD EDLTEDAQN ENKEQQDKDE RDDENEDDAV EMEGDMAGEL EDLSNGEEND DEDTDSEEEE LDEEIDDLNE DDPNAIDDKM W DDKASDNS KEKDTDQNLD GKNQEEDVQA AENDEQQRDN KEGGDEDPNA PEDGDEEIEN DENAEEENDV GEQEDEVKDE EG EDLEANV PEIETLDLPE DMNLDSEHEE SDEDVDMSDG MPDDLNKEEV GNEDEEVKQE SGIESDNEND EPGPEEDAGE TET ALDEEE GAEEDVDMTN DEGKEDEENG PEEQAMSDEE ELKQDAAMEE NKEKGGEQNT EGLDGVEEKA DTEDIDQEAA VQQD SGSKG AGADATDTQE QDDVGGSGTT QNTYEEDQED VTKNNEESRE EATAALKQLG DSMKEYHRRR QDIKEAQTNG EEDEN LEKN NERPDEFEHV EGANTETDTQ ALGSATQDQL QTIDEDMAID DDREEQEVDQ KELVEDADDE KMDIDEEEML SDIDAH DAN NDVDSKKSGF IGKRKSEEDF ENELSNEHFS ADQEDDSEIQ SLIENIEDNP PDASASLTPE RSLEESRELW HKSEIST AD LVSRLGEQLR LILEPTLATK LKGDYKTGKR LNMKRIIPYI ASQFRKDKIW LRRTKPSKRQ YQIMIALDDS KSMSESKC V KLAFDSLCLV SKTLTQLEAG GLSIVKFGEN IKEVHSFDQQ FSNESGARAF QWFGFQETKT DVKKLVAEST KIFERARAM VHNDQWQLEI VISDGICEDH ETIQKLVRRA RENKIMLVFV IIDGITSNES ILDMSQVNYI PDQYGNPQLK ITKYLDTFPF EFYVVVHDI SELPEMLSLI LRQYFTDLAS S |
-分子 #2: Ribosome assembly protein 4
| 分子 | 名称: Ribosome assembly protein 4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Baker's yeast (パン酵母) Baker's yeast (パン酵母) |
| 分子量 | 理論値: 57.106781 KDa |
| 配列 | 文字列: MSTLIPPPSK KQKKEAQLPR EVAIIPKDLP NVSIKFQALD TGDNVGGALR VPGAISEKQL EELLNQLNGT SDDPVPYTFS CTIQGKKAS DPVKTIDITD NLYSSLIKPG YNSTEDQITL LYTPRAVFKV KPVTRSSSAI AGHGSTILCS AFAPHTSSRM V TGAGDNTA ...文字列: MSTLIPPPSK KQKKEAQLPR EVAIIPKDLP NVSIKFQALD TGDNVGGALR VPGAISEKQL EELLNQLNGT SDDPVPYTFS CTIQGKKAS DPVKTIDITD NLYSSLIKPG YNSTEDQITL LYTPRAVFKV KPVTRSSSAI AGHGSTILCS AFAPHTSSRM V TGAGDNTA RIWDCDTQTP MHTLKGHYNW VLCVSWSPDG EVIATGSMDN TIRLWDPKSG QCLGDALRGH SKWITSLSWE PI HLVKPGS KPRLASSSKD GTIKIWDTVS RVCQYTMSGH TNSVSCVKWG GQGLLYSGSH DRTVRVWDIN SQGRCINILK SHA HWVNHL SLSTDYALRI GAFDHTGKKP STPEEAQKKA LENYEKICKK NGNSEEMMVT ASDDYTMFLW NPLKSTKPIA RMTG HQKLV NHVAFSPDGR YIVSASFDNS IKLWDGRDGK FISTFRGHVA SVYQVAWSSD CRLLVSCSKD TTLKVWDVRT RKLSV DLPG HKDEVYTVDW SVDGKRVCSG GKDKMVRLWT H |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R3/3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 0.3 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-48 / 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 75.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 273799 |
|---|---|
| CTF補正 | ソフトウェア - 名称: Gctf (ver. 1.06) |
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.0.8) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 3.0.8) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.0.8) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 4.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0.8) / 使用した粒子像数: 55397 |
 ムービー
ムービー コントローラー
コントローラー






























 Z
Z Y
Y X
X