+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10828 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of C. jejuni MotAB | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Campylobacter jejuni subsp. jejuni 81-176 (カンピロバクター) / Campylobacter jejuni subsp. jejuni 81-176 (カンピロバクター) /   Campylobacter jejuni subsp. jejuni serotype O:23/36 (strain 81-176) (カンピロバクター) Campylobacter jejuni subsp. jejuni serotype O:23/36 (strain 81-176) (カンピロバクター) | |||||||||
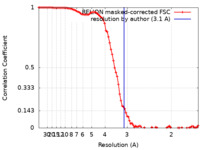
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Santiveri M / Roa-Eguiara A / Taylor NMI | |||||||||
| 資金援助 |  デンマーク, 2件 デンマーク, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: Structure and Function of Stator Units of the Bacterial Flagellar Motor. 著者: Mònica Santiveri / Aritz Roa-Eguiara / Caroline Kühne / Navish Wadhwa / Haidai Hu / Howard C Berg / Marc Erhardt / Nicholas M I Taylor /    要旨: Many bacteria use the flagellum for locomotion and chemotaxis. Its bidirectional rotation is driven by a membrane-embedded motor, which uses energy from the transmembrane ion gradient to generate ...Many bacteria use the flagellum for locomotion and chemotaxis. Its bidirectional rotation is driven by a membrane-embedded motor, which uses energy from the transmembrane ion gradient to generate torque at the interface between stator units and rotor. The structural organization of the stator unit (MotAB), its conformational changes upon ion transport, and how these changes power rotation of the flagellum remain unknown. Here, we present ~3 Å-resolution cryoelectron microscopy reconstructions of the stator unit in different functional states. We show that the stator unit consists of a dimer of MotB surrounded by a pentamer of MotA. Combining structural data with mutagenesis and functional studies, we identify key residues involved in torque generation and present a detailed mechanistic model for motor function and switching of rotational direction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10828.map.gz emd_10828.map.gz | 6.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10828-v30.xml emd-10828-v30.xml emd-10828.xml emd-10828.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10828_fsc.xml emd_10828_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10828.png emd_10828.png | 119.3 KB | ||
| マスクデータ |  emd_10828_msk_1.map emd_10828_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10828_half_map_1.map.gz emd_10828_half_map_1.map.gz emd_10828_half_map_2.map.gz emd_10828_half_map_2.map.gz | 49.4 MB 49.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10828 http://ftp.pdbj.org/pub/emdb/structures/EMD-10828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10828 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10828.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10828.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10828_msk_1.map emd_10828_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10828_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10828_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Stator unit MotAB
| 全体 | 名称: Stator unit MotAB |
|---|---|
| 要素 |
|
-超分子 #1: Stator unit MotAB
| 超分子 | 名称: Stator unit MotAB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: The stator unit consists of a dimer of MotB surrounded by a pentamer of MotA |
|---|---|
| 由来(天然) | 生物種:   Campylobacter jejuni subsp. jejuni 81-176 (カンピロバクター) Campylobacter jejuni subsp. jejuni 81-176 (カンピロバクター) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: C43(DE3) Escherichia coli (大腸菌) / 組換株: C43(DE3) |
-分子 #1: Chemotaxis protein MotA, putative
| 分子 | 名称: Chemotaxis protein MotA, putative / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Campylobacter jejuni subsp. jejuni serotype O:23/36 (strain 81-176) (カンピロバクター) Campylobacter jejuni subsp. jejuni serotype O:23/36 (strain 81-176) (カンピロバクター)株: 81-176 |
| 分子量 | 理論値: 28.195816 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MDLSTILGMV LAVTSISVGD ILEGGNPLHV IHLSSFLIVM PTAAFCAMTS THKKIVKAAY KELKVVFKGS GVNLPERIAQ LIEFAIIAR RDGLLALESR TNEIENEFLK NAMMMLVDGK SFEEIHESME IQTEQLEEHY KECAEYWIVF GETCPTMGLV G AVFGLILA ...文字列: MDLSTILGMV LAVTSISVGD ILEGGNPLHV IHLSSFLIVM PTAAFCAMTS THKKIVKAAY KELKVVFKGS GVNLPERIAQ LIEFAIIAR RDGLLALESR TNEIENEFLK NAMMMLVDGK SFEEIHESME IQTEQLEEHY KECAEYWIVF GETCPTMGLV G AVFGLILA LKLLDNPQAM AAGISGAFTA TVTGIFGAYA LFAPWGKKLK ANGMDLVKEQ IVITEAIKGI AEGANPRDLE AK LFNFLSH DDPRISQFDK G |
-分子 #2: Chemotaxis protein MotB, putative
| 分子 | 名称: Chemotaxis protein MotB, putative / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Campylobacter jejuni subsp. jejuni serotype O:23/36 (strain 81-176) (カンピロバクター) Campylobacter jejuni subsp. jejuni serotype O:23/36 (strain 81-176) (カンピロバクター)株: 81-176 |
| 分子量 | 理論値: 32.149244 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAKKHKCPEC PAGEKWAVPY ADFLSLLLAL FIALWAISKT NPAKVEALKT EFVKIFDYTS TQTVKEESKT QEKYKGAAKE ESDELKSLK QMTMTQQETI KRLQAALDQS DNQVALNLPS KVEFERGSAQ IVSADIQDYL KRMAELTTYL PPQAKIEIRG Y TDNSDSII ...文字列: MAKKHKCPEC PAGEKWAVPY ADFLSLLLAL FIALWAISKT NPAKVEALKT EFVKIFDYTS TQTVKEESKT QEKYKGAAKE ESDELKSLK QMTMTQQETI KRLQAALDQS DNQVALNLPS KVEFERGSAQ IVSADIQDYL KRMAELTTYL PPQAKIEIRG Y TDNSDSII RSYELAYQRA ENVLKYFIEG GANLKNISIK SYGLNNPING NPQALENNRV EIYFKVDTAD TSTQKSVLEL IN KIGTKAP GTLEVLFQGP GGSGSAWSHP QFEKGGGSGG GSGGSAWSHP QFEK |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 8 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.65 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R2/1 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.84 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X