+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0704 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of LbCas12a-crRNA complex bound to AcrVA4 (form A complex) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Lachnospiraceae bacterium (バクテリア) / Lachnospiraceae bacterium (バクテリア) /   Moraxella bovoculi (モラクセラ・ボーボクリ) / synthetic construct (人工物) Moraxella bovoculi (モラクセラ・ボーボクリ) / synthetic construct (人工物) | |||||||||
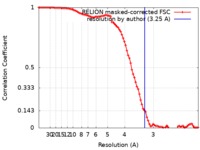
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.25 Å クライオ電子顕微鏡法 / 解像度: 3.25 Å | |||||||||
 データ登録者 データ登録者 | Peng R / Li Z / Xu Y / He S / Peng Q / Shi Y / Gao GF | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: Structural insight into multistage inhibition of CRISPR-Cas12a by AcrVA4. 著者: Ruchao Peng / Zhiteng Li / Ying Xu / Shaoshuai He / Qi Peng / Lian-Ao Wu / Ying Wu / Jianxun Qi / Peiyi Wang / Yi Shi / George F Gao /  要旨: Prokaryotes possess CRISPR-Cas systems to exclude parasitic predators, such as phages and mobile genetic elements (MGEs). These predators, in turn, encode anti-CRISPR (Acr) proteins to evade the ...Prokaryotes possess CRISPR-Cas systems to exclude parasitic predators, such as phages and mobile genetic elements (MGEs). These predators, in turn, encode anti-CRISPR (Acr) proteins to evade the CRISPR-Cas immunity. Recently, AcrVA4, an Acr protein inhibiting the CRISPR-Cas12a system, was shown to diminish Cas12a (LbCas12a)-mediated genome editing in human cells, but the underlying mechanisms remain elusive. Here we report the cryo-EM structures of AcrVA4 bound to CRISPR RNA (crRNA)-loaded LbCas12a and found AcrVA4 could inhibit LbCas12a at several stages of the CRISPR-Cas working pathway, different from other characterized type I/II Acr inhibitors which target only 1 stage. First, it locks the conformation of the LbCas12a-crRNA complex to prevent target DNA-crRNA hybridization. Second, it interacts with the LbCas12a-crRNA-dsDNA complex to release the bound DNA before cleavage. Third, AcrVA4 binds the postcleavage LbCas12a complex to possibly block enzyme recycling. These findings highlight the multifunctionality of AcrVA4 and provide clues for developing regulatory genome-editing tools. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0704.map.gz emd_0704.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0704-v30.xml emd-0704-v30.xml emd-0704.xml emd-0704.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0704_fsc.xml emd_0704_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0704.png emd_0704.png | 63.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0704 http://ftp.pdbj.org/pub/emdb/structures/EMD-0704 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0704 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0704 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0704.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0704.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : LbCas12a-crRNA complex bound to AcrVA4 (form A complex)
| 全体 | 名称: LbCas12a-crRNA complex bound to AcrVA4 (form A complex) |
|---|---|
| 要素 |
|
-超分子 #1: LbCas12a-crRNA complex bound to AcrVA4 (form A complex)
| 超分子 | 名称: LbCas12a-crRNA complex bound to AcrVA4 (form A complex) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium (バクテリア) Lachnospiraceae bacterium (バクテリア) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: BL21 Escherichia coli (大腸菌) / 組換株: BL21 |
| 分子量 | 実験値: 170 KDa |
-分子 #1: LbCas12a
| 分子 | 名称: LbCas12a / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lachnospiraceae bacterium (バクテリア) Lachnospiraceae bacterium (バクテリア) |
| 分子量 | 理論値: 143.888359 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MSKLEKFTNC YSLSKTLRFK AIPVGKTQEN IDNKRLLVED EKRAEDYKGV KKLLDRYYLS FINDVLHSIK LKNLNNYISL FRKKTRTEK ENKELENLEI NLRKEIAKAF KGNEGYKSLF KKDIIETILP EFLDDKDEIA LVNSFNGFTT AFTGFFDNRE N MFSEEAKS ...文字列: MSKLEKFTNC YSLSKTLRFK AIPVGKTQEN IDNKRLLVED EKRAEDYKGV KKLLDRYYLS FINDVLHSIK LKNLNNYISL FRKKTRTEK ENKELENLEI NLRKEIAKAF KGNEGYKSLF KKDIIETILP EFLDDKDEIA LVNSFNGFTT AFTGFFDNRE N MFSEEAKS TSIAFRCINE NLTRYISNMD IFEKVDAIFD KHEVQEIKEK ILNSDYDVED FFEGEFFNFV LTQEGIDVYN AI IGGFVTE SGEKIKGLNE YINLYNQKTK QKLPKFKPLY KQVLSDRESL SFYGEGYTSD EEVLEVFRNT LNKNSEIFSS IKK LEKLFK NFDEYSSAGI FVKNGPAIST ISKDIFGEWN VIRDKWNAEY DDIHLKKKAV VTEKYEDDRR KSFKKIGSFS LEQL QEYAD ADLSVVEKLK EIIIQKVDEI YKVYGSSEKL FDADFVLEKS LKKNDAVVAI MKDLLDSVKS FENYIKAFFG EGKET NRDE SFYGDFVLAY DILLKVDHIY DAIRNYVTQK PYSKDKFKLY FQNPQFMGGW DKDKETDYRA TILRYGSKYY LAIMDK KYA KCLQKIDKDD VNGNYEKINY KLLPGPNKML PKVFFSKKWM AYYNPSEDIQ KIYKNGTFKK GDMFNLNDCH KLIDFFK DS ISRYPKWSNA YDFNFSETEK YKDIAGFYRE VEEQGYKVSF ESASKKEVDK LVEEGKLYMF QIYNKDFSDK SHGTPNLH T MYFKLLFDEN NHGQIRLSGG AELFMRRASL KKEELVVHPA NSPIANKNPD NPKKTTTLSY DVYKDKRFSE DQYELHIPI AINKCPKNIF KINTEVRVLL KHDDNPYVIG IDRGERNLLY IVVVDGKGNI VEQYSLNEII NNFNGIRIKT DYHSLLDKKE KERFEARQN WTSIENIKEL KAGYISQVVH KICELVEKYD AVIALEDLNS GFKNSRVKVE KQVYQKFEKM LIDKLNYMVD K KSNPCATG GALKGYQITN KFESFKSMST QNGFIFYIPA WLTSKIDPST GFVNLLKTKY TSIADSKKFI SSFDRIMYVP EE DLFEFAL DYKNFSRTDA DYIKKWKLYS YGNRIRIFRN PKKNNVFDWE EVCLTSAYKE LFNKYGINYQ QGDIRALLCE QSD KAFYSS FMALMSLMLQ MRNSITGRTD VDFLISPVKN SDGIFYDSRN YEAQENAILP KNADANGAYN IARKVLWAIG QFKK AEDEK LDKVKIAISN KEWLEYAQTS VKH |
-分子 #2: AcrVA4
| 分子 | 名称: AcrVA4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Moraxella bovoculi (モラクセラ・ボーボクリ) Moraxella bovoculi (モラクセラ・ボーボクリ) |
| 分子量 | 理論値: 27.369162 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MYEIKLNDTL IHQTDDRVNA FVAYRYLLRR GDLPKCENIA RMYYDGKVIK TDVIDHDSVH SDEQAKVSNN DIIKMAISEL GVNNFKSLI KKQGYPFSNG HINSWFTDDP VKSKTMHNDE MYLVVQALIR ACIIKEIDLY TEQLYNIIKS LPYDKRPNVV Y SDQPLDPN ...文字列: MYEIKLNDTL IHQTDDRVNA FVAYRYLLRR GDLPKCENIA RMYYDGKVIK TDVIDHDSVH SDEQAKVSNN DIIKMAISEL GVNNFKSLI KKQGYPFSNG HINSWFTDDP VKSKTMHNDE MYLVVQALIR ACIIKEIDLY TEQLYNIIKS LPYDKRPNVV Y SDQPLDPN NLDLSEPELW AEQVGECMRY AHNDQPCFYI GSTKRELRVN YIVPVIGVRD EIERVMTLEE VRNLHK |
-分子 #3: RNA (42-MER)
| 分子 | 名称: RNA (42-MER) / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.48202 KDa |
| 配列 | 文字列: AAUUUCUACU AAGUGUAGAU CGGUCUCGCA AAGAAUGGAU AU |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 6 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー