+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0553 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
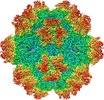
| タイトル | Cryo-EM structure of AAV-2 in complex with AAVR PKD domains 1 and 2 | |||||||||
 マップデータ マップデータ | EM sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報permeabilization of host organelle membrane involved in viral entry into host cell / symbiont entry into host cell via permeabilization of inner membrane / host cell nucleolus / T=1 icosahedral viral capsid /  neuron migration / cytoplasmic vesicle / clathrin-dependent endocytosis of virus by host cell / neuron migration / cytoplasmic vesicle / clathrin-dependent endocytosis of virus by host cell /  ゴルジ体 / structural molecule activity / virion attachment to host cell ...permeabilization of host organelle membrane involved in viral entry into host cell / symbiont entry into host cell via permeabilization of inner membrane / host cell nucleolus / T=1 icosahedral viral capsid / ゴルジ体 / structural molecule activity / virion attachment to host cell ...permeabilization of host organelle membrane involved in viral entry into host cell / symbiont entry into host cell via permeabilization of inner membrane / host cell nucleolus / T=1 icosahedral viral capsid /  neuron migration / cytoplasmic vesicle / clathrin-dependent endocytosis of virus by host cell / neuron migration / cytoplasmic vesicle / clathrin-dependent endocytosis of virus by host cell /  ゴルジ体 / structural molecule activity / virion attachment to host cell / ゴルジ体 / structural molecule activity / virion attachment to host cell /  核小体 / 核小体 /  ゴルジ体 / ゴルジ体 /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Adeno-associated virus - 2 (アデノ随伴ウイルス) Adeno-associated virus - 2 (アデノ随伴ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.4 Å クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Meyer NL / Xie Q / Davulcu O / Yoshioka C / Chapman MS | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: Structure of the gene therapy vector, adeno-associated virus with its cell receptor, AAVR. 著者: Nancy L Meyer / Guiqing Hu / Omar Davulcu / Qing Xie / Alex J Noble / Craig Yoshioka / Drew S Gingerich / Andrew Trzynka / Larry David / Scott M Stagg / Michael Stewart Chapman /  要旨: Adeno-associated virus (AAV) vectors are preeminent in emerging clinical gene therapies. Generalizing beyond the most tractable genetic diseases will require modulation of cell specificity and immune ...Adeno-associated virus (AAV) vectors are preeminent in emerging clinical gene therapies. Generalizing beyond the most tractable genetic diseases will require modulation of cell specificity and immune neutralization. Interactions of AAV with its cellular receptor, AAVR, are key to understanding cell-entry and trafficking with the rigor needed to engineer tissue-specific vectors. -electron tomography shows ordered binding of part of the flexible receptor to the viral surface, with distal domains in multiple conformations. Regions of the virus and receptor in close physical proximity can be identified by cross-linking/mass spectrometry. -electron microscopy with a two-domain receptor fragment reveals the interactions at 2.4 Å resolution. AAVR binds between AAV's spikes on a plateau that is conserved, except in one clade whose structure is AAVR-incompatible. AAVR's footprint overlaps the epitopes of several neutralizing antibodies, prompting a re-evaluation of neutralization mechanisms. The structure provides a roadmap for experimental probing and manipulation of viral-receptor interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0553.map.gz emd_0553.map.gz | 42.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0553-v30.xml emd-0553-v30.xml emd-0553.xml emd-0553.xml | 25.9 KB 25.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_0553.png emd_0553.png | 319.1 KB | ||
| マスクデータ |  emd_0553_msk_1.map emd_0553_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0553_half_map_1.map.gz emd_0553_half_map_1.map.gz emd_0553_half_map_2.map.gz emd_0553_half_map_2.map.gz | 170.7 MB 170.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0553 http://ftp.pdbj.org/pub/emdb/structures/EMD-0553 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0553 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0553 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0553.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0553.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.046 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
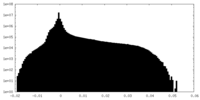
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0553_msk_1.map emd_0553_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
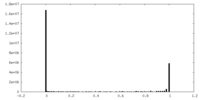
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_0553_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
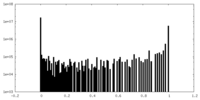
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_0553_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
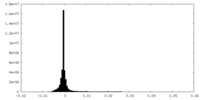
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus - 2
| 全体 | 名称:   Adeno-associated virus - 2 (アデノ随伴ウイルス) Adeno-associated virus - 2 (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Adeno-associated virus - 2
| 超分子 | 名称: Adeno-associated virus - 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: AAV-2 virus-like particles expressed in Sf9 cells using Invitrogen's Bac-to-Bac expression system NCBI-ID: 10804 / 生物種: Adeno-associated virus - 2 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| Host system | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 分子量 | 理論値: 22 KDa |
| ウイルス殻 | Shell ID: 1 / 直径: 250.0 Å / T番号(三角分割数): 1 |
-超分子 #2: Binary complex of AAV-2 with a fragment of its cellular receptor,...
| 超分子 | 名称: Binary complex of AAV-2 with a fragment of its cellular receptor, AAVR or dyslexia-associated protein KIAA0319-like protein. タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: Expressed N-terminally His6-tagged fragment of AAVR containing the PKD1 and PKD2 domains. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Dyslexia-associated protein KIAA0319-like protein
| 分子 | 名称: Dyslexia-associated protein KIAA0319-like protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31.837385 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MASVSAGESV QITLPKNEVQ LNAYVLQEPP KGETYTYDWQ LITHPRDYSG EMEGKHSQIL KLSKLTPGLY EFKVIVEGQN AHGEGYVNV TVKPEPRKNR PPIAIVSPQF QEISLPTTST VIDGSQSTDD DKIVQYHWEE LKGPLREEKI SEDTAILKLS K LVPGNYTF ...文字列: MASVSAGESV QITLPKNEVQ LNAYVLQEPP KGETYTYDWQ LITHPRDYSG EMEGKHSQIL KLSKLTPGLY EFKVIVEGQN AHGEGYVNV TVKPEPRKNR PPIAIVSPQF QEISLPTTST VIDGSQSTDD DKIVQYHWEE LKGPLREEKI SEDTAILKLS K LVPGNYTF SLTVVDSDGA TNSTTANLTV NKAVDYPPVA NAGPNQVITL PQNSITLFGN QSTDDHGITS YEWSLSPSSK GK VVEMQGV RTPTLQLSAM QEGDYTYQLT VTDTIGQQAT AQVTVIVQPE NNK |
-分子 #2: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Adeno-associated virus - 2 (アデノ随伴ウイルス) Adeno-associated virus - 2 (アデノ随伴ウイルス) |
| 分子量 | 理論値: 82.077445 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAADGYLPDW LEDTLSEGIR QWWKLKPGPP PPKPAERHKD DSRGLVLPGY KYLGPFNGLD KGEPVNEADA AALEHDKAYD RQLDSGDNP YLKYNHADAE FQERLKEDTS FGGNLGRAVF QAKKRVLEPL GLVEEPVKTA PGKKRPVEHS PVEPDSSSGT G KAGQQPAR ...文字列: MAADGYLPDW LEDTLSEGIR QWWKLKPGPP PPKPAERHKD DSRGLVLPGY KYLGPFNGLD KGEPVNEADA AALEHDKAYD RQLDSGDNP YLKYNHADAE FQERLKEDTS FGGNLGRAVF QAKKRVLEPL GLVEEPVKTA PGKKRPVEHS PVEPDSSSGT G KAGQQPAR KRLNFGQTGD ADSVPDPQPL GQPPAAPSGL GTNTMATGSG APMADNNEGA DGVGNSSGNW HCDSTWMGDR VI TTSTRTW ALPTYNNHLY KQISSQSGAS NDNHYFGYST PWGYFDFNRF HCHFSPRDWQ RLINNNWGFR PKRLNFKLFN IQV KEVTQN DGTTTIANNL TSTVQVFTDS EYQLPYVLGS AHQGCLPPFP ADVFMVPQYG YLTLNNGSQA VCRSSFYCLE YFPS QMLRT GNNFTFSYTF EDVPFHSSYA HSQSLDRLMN PLIDQYLYYL SRTNTPSGTT TQSRLQFSQA GASDIRDQSR NWLPG PCYR QQRVSKTSAD NNNSEYSWTG ATKYHLNGRD SLVNPGPAMA SHKDDEEKFF PQSGVLIFGK QGSEKTNVDI EKVMIT DEE EIRTTNPVAT EQYGSVSTNL QRGNRQAATA DVNTQGVLPG MVWQDRDVYL QGPIWAKIPH TDGHFHPSPL MGGFGLK HP PPQILIKNTP VPANPSTTFS AAKFASFITQ YSTGQVSVEI EWELQKENSK RWNPEIQYTS NYNKSVNVDF TVDTNGVY S EPRPIGTRYL TRNL |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 143 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||
| グリッド | 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa 詳細: Glow discharge settings: Pelco easiGlow, 25s, 25mA, 0.39mBar, negative polarity Grid: Ted Pella Cat#01824, ultrathin carbon film (<3nm) on lacey carbon support film | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298.15 K / 装置: FEI VITROBOT MARK IV 詳細: 4ul of AAV-2 (1.7uM) was added to the grid, followed by 4ul of AAVR-PKD1-2 (16.7uM), with manual blotting after each addition with Whatman paper (Cat. no. 1001-110.) 4ul of buffer containing ...詳細: 4ul of AAV-2 (1.7uM) was added to the grid, followed by 4ul of AAVR-PKD1-2 (16.7uM), with manual blotting after each addition with Whatman paper (Cat. no. 1001-110.) 4ul of buffer containing 25mM HEPES, 150mM NaCl, pH 7.4 was added to the grid before final blotting and plunge-freezing in the Vitrobot (blot time 1.5sec, blot force -1).. | ||||||||
| 詳細 | Virus-like particles at 0.10 mg/mL were adhered to thin continuous carbon-coated grids, then, after initial blotting, AAVR-PKD1-2 was added at 0.74 mg/mL (a 10-fold molar excess over AAV2 subunits) before blotting again. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.0 µm / 最小 デフォーカス(公称値): -0.8 µm / 倍率(公称値): 75000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.0 µm / 最小 デフォーカス(公称値): -0.8 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / 撮影したグリッド数: 1 / 実像数: 2329 / 平均露光時間: 40.0 sec. / 平均電子線量: 25.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 34450 詳細: RELION Autopicker using 3D AAV-2 reference filtered to 20A with 15 degree sampling |
|---|---|
| CTF補正 | ソフトウェア - 名称: Gctf (ver. 1.06) / ソフトウェア - 詳細: resolution range: 30-3A |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.0b) |
| 最終 3次元分類 | クラス数: 2 / ソフトウェア - 名称: RELION (ver. 3.0b) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.0b) |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: I (正20面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0b) / 使用した粒子像数: 21373 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0b) / 使用した粒子像数: 21373 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 217-735 |
|---|---|
| 詳細 | An initial model for AAVR was constructed by homology modeling using Modeller 9.2. After remodeling AAVR and the crystal structure of AAV2, the structure was refined using RSRef embedded in CNS for stereochemically restrained torsion angle simulated annealing and gradient descent optimization. Stand-alone RSRef was used for refinement of EM envelope corrections, resolution estimates, and restrained atomic B-factors. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Map values at grid points within 2 Angstrom of atoms |
| 得られたモデル |  PDB-6nz0: |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X