+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0333 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Helical RNA-bound Hantaan virus nucleocapsid | |||||||||
 マップデータ マップデータ | Helical RNA-bound Hantaan virus nucleocapsid structure | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / helical viral capsid / : / host cell Golgi apparatus / viral nucleocapsid /  endonuclease activity / endonuclease activity /  加水分解酵素; エステル加水分解酵素 / host cell perinuclear region of cytoplasm / 加水分解酵素; エステル加水分解酵素 / host cell perinuclear region of cytoplasm /  ribonucleoprotein complex / ribonucleoprotein complex /  RNA binding RNA binding類似検索 - 分子機能 | |||||||||
| 生物種 |  Hantaan orthohantavirus (ウイルス) / Hantaan orthohantavirus (ウイルス) /   Spodoptera frugiperda (ツマジロクサヨトウ) / Spodoptera frugiperda (ツマジロクサヨトウ) /   Fall armyworm (ツマジロクサヨトウ) Fall armyworm (ツマジロクサヨトウ) | |||||||||
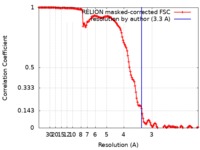
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Arragain B / Reguera J / Desfosses A / Gutsche I / Schoehn G / Malet H | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: High resolution cryo-EM structure of the helical RNA-bound Hantaan virus nucleocapsid reveals its assembly mechanisms. 著者: Benoît Arragain / Juan Reguera / Ambroise Desfosses / Irina Gutsche / Guy Schoehn / Hélène Malet /  要旨: Negative-strand RNA viruses condense their genome into helical nucleocapsids that constitute essential templates for viral replication and transcription. The intrinsic flexibility of nucleocapsids ...Negative-strand RNA viruses condense their genome into helical nucleocapsids that constitute essential templates for viral replication and transcription. The intrinsic flexibility of nucleocapsids usually prevents their full-length structural characterisation at high resolution. Here, we describe purification of full-length recombinant metastable helical nucleocapsid of Hantaan virus ( family, order) and determine its structure at 3.3 Å resolution by cryo-electron microscopy. The structure reveals the mechanisms of helical multimerisation via sub-domain exchanges between protomers and highlights nucleotide positions in a continuous positively charged groove compatible with viral genome binding. It uncovers key sites for future structure-based design of antivirals that are currently lacking to counteract life-threatening hantavirus infections. The structure also suggests a model of nucleoprotein-polymerase interaction that would enable replication and transcription solely upon local disruption of the nucleocapsid. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0333.map.gz emd_0333.map.gz | 14 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0333-v30.xml emd-0333-v30.xml emd-0333.xml emd-0333.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0333_fsc.xml emd_0333_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0333.png emd_0333.png | 149.2 KB | ||
| その他 |  emd_0333_half_map_1.map.gz emd_0333_half_map_1.map.gz emd_0333_half_map_2.map.gz emd_0333_half_map_2.map.gz | 228.7 MB 228.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0333 http://ftp.pdbj.org/pub/emdb/structures/EMD-0333 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0333 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0333 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0333.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0333.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Helical RNA-bound Hantaan virus nucleocapsid structure | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.067 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half map 2
| ファイル | emd_0333_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
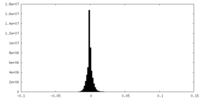
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_0333_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA-bound Hantaan virus nucleocapsid
| 全体 | 名称: RNA-bound Hantaan virus nucleocapsid |
|---|---|
| 要素 |
|
-超分子 #1: RNA-bound Hantaan virus nucleocapsid
| 超分子 | 名称: RNA-bound Hantaan virus nucleocapsid / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 48.2 KDa |
-超分子 #2: Hantaan virus nucleoprotein
| 超分子 | 名称: Hantaan virus nucleoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Hantaan orthohantavirus (ウイルス) Hantaan orthohantavirus (ウイルス) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)組換株: SF21 / 組換プラスミド: pFastBac |
-超分子 #3: RNA
| 超分子 | 名称: RNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
-分子 #1: RNA (5'-R(P*UP*UP*U)-3')
| 分子 | 名称: RNA (5'-R(P*UP*UP*U)-3') / タイプ: rna / ID: 1 詳細: A poly-U has been built but the RNA corresponds to heterogeneous insect cell RNA that bind to nucleoproteins during expression. コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Fall armyworm (ツマジロクサヨトウ) Fall armyworm (ツマジロクサヨトウ) |
| 分子量 | 理論値: 873.54 Da |
| 配列 | 文字列: UUU |
-分子 #2: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hantaan orthohantavirus (ウイルス) Hantaan orthohantavirus (ウイルス) |
| 分子量 | 理論値: 48.262266 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: GMATMEELQR EINAHEGQLV IARQKVRDAE KQYEKDPDEL NKRTLTDREG VAVSIQAKID ELKRQLADRI ATGKNLGKEQ DPTGVEPGD HLKERSMLSY GNVLDLNHLD IDEPTGQTAD WLSIIVYLTS FVVPILLKAL YMLTTRGRQT TKDNKGTRIR F KDDSSFED ...文字列: GMATMEELQR EINAHEGQLV IARQKVRDAE KQYEKDPDEL NKRTLTDREG VAVSIQAKID ELKRQLADRI ATGKNLGKEQ DPTGVEPGD HLKERSMLSY GNVLDLNHLD IDEPTGQTAD WLSIIVYLTS FVVPILLKAL YMLTTRGRQT TKDNKGTRIR F KDDSSFED VNGIRKPKHL YVSLPNAQSS MKAEEITPGR YRTAVCGLYP AQIKARQMIS PVMSVIGFLA LAKDWSDRIE QW LIEPCKL LPDTAAVSLL GGPATNRDYL RQRQVALGNM ETKESKAIRQ HAEAAGCSMI EDIESPSSIW VFAGAPDRCP PTC LFIAGI AELGAFFSIL QDMRNTIMAS KTVGTSEEKL RKKSSFYQSY LRRTQSMGIQ LGQRIIVLFM VAWGKEAVDN FHLG DDMDP ELRTLAQSLI DVKVKEISNQ EPLKL |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: 30 mA | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV 詳細: 3 ul of sample were applied on the resulting glow-discharged grids and excess solution was blotted during 2.5 sec force 7. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.5 µm / 最小 デフォーカス(補正後): 0.8 µm / 倍率(補正後): 46860 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3840 pixel / デジタル化 - サイズ - 縦: 3712 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 3-18 / 撮影したグリッド数: 1 / 実像数: 4328 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X