+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0325 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of the Ysh1-Mpe1 nuclease complex | ||||||||||||
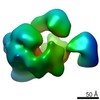
 マップデータ マップデータ | Cryo-EM map of the Ysh1-Mpe1 complex | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.0 Å クライオ電子顕微鏡法 / 解像度: 6.0 Å | ||||||||||||
 データ登録者 データ登録者 | Hill CH / Boreikaite V / Kumar A / Casanal A / Kubik P / Degliesposti G / Maslen S / Mariani A / von Loeffelholz O / Girbig M ...Hill CH / Boreikaite V / Kumar A / Casanal A / Kubik P / Degliesposti G / Maslen S / Mariani A / von Loeffelholz O / Girbig M / Skehel M / Passmore LA | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2019 ジャーナル: Mol Cell / 年: 2019タイトル: Activation of the Endonuclease that Defines mRNA 3' Ends Requires Incorporation into an 8-Subunit Core Cleavage and Polyadenylation Factor Complex. 著者: Chris H Hill / Vytautė Boreikaitė / Ananthanarayanan Kumar / Ana Casañal / Peter Kubík / Gianluca Degliesposti / Sarah Maslen / Angelica Mariani / Ottilie von Loeffelholz / Mathias Girbig ...著者: Chris H Hill / Vytautė Boreikaitė / Ananthanarayanan Kumar / Ana Casañal / Peter Kubík / Gianluca Degliesposti / Sarah Maslen / Angelica Mariani / Ottilie von Loeffelholz / Mathias Girbig / Mark Skehel / Lori A Passmore /   要旨: Cleavage and polyadenylation factor (CPF/CPSF) is a multi-protein complex essential for formation of eukaryotic mRNA 3' ends. CPF cleaves pre-mRNAs at a specific site and adds a poly(A) tail. The ...Cleavage and polyadenylation factor (CPF/CPSF) is a multi-protein complex essential for formation of eukaryotic mRNA 3' ends. CPF cleaves pre-mRNAs at a specific site and adds a poly(A) tail. The cleavage reaction defines the 3' end of the mature mRNA, and thus the activity of the endonuclease is highly regulated. Here, we show that reconstitution of specific pre-mRNA cleavage with recombinant yeast proteins requires incorporation of the Ysh1 endonuclease into an eight-subunit "CPF" complex. Cleavage also requires the accessory cleavage factors IA and IB, which bind substrate pre-mRNAs and CPF, likely facilitating assembly of an active complex. Using X-ray crystallography, electron microscopy, and mass spectrometry, we determine the structure of Ysh1 bound to Mpe1 and the arrangement of subunits within CPF. Together, our data suggest that the active mRNA 3' end processing machinery is a dynamic assembly that is licensed to cleave only when all protein factors come together at the polyadenylation site. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0325.map.gz emd_0325.map.gz | 1.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0325-v30.xml emd-0325-v30.xml emd-0325.xml emd-0325.xml | 21.6 KB 21.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0325_fsc.xml emd_0325_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0325.png emd_0325.png | 56.5 KB | ||
| マスクデータ |  emd_0325_msk_1.map emd_0325_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0325_half_map_1.map.gz emd_0325_half_map_1.map.gz emd_0325_half_map_2.map.gz emd_0325_half_map_2.map.gz | 6 MB 6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0325 http://ftp.pdbj.org/pub/emdb/structures/EMD-0325 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0325 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0325 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0325.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0325.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the Ysh1-Mpe1 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0325_msk_1.map emd_0325_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map 1
| ファイル | emd_0325_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map 2
| ファイル | emd_0325_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ysh1-Mpe1 complex Highly anisotropic due to severe preferred orie...
| 全体 | 名称: Ysh1-Mpe1 complex Highly anisotropic due to severe preferred orientation. |
|---|---|
| 要素 |
|
-超分子 #1: Ysh1-Mpe1 complex Highly anisotropic due to severe preferred orie...
| 超分子 | 名称: Ysh1-Mpe1 complex Highly anisotropic due to severe preferred orientation. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The trimeric Ysh1-Mpe1-Yjr141w complex was analysed, however several regions are flexible/disordered. This map corresponds to the Ysh1 N-terminal nuclease domain in complex with the Mpe1 N- ...詳細: The trimeric Ysh1-Mpe1-Yjr141w complex was analysed, however several regions are flexible/disordered. This map corresponds to the Ysh1 N-terminal nuclease domain in complex with the Mpe1 N-terminal ubiquitin-like domain. These are the only ordered, globular densities present in the above sample. |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) / 組換細胞: Sf9 unidentified baculovirus (ウイルス) / 組換細胞: Sf9 |
| 分子量 | 理論値: 57 KDa |
-分子 #1: Ysh1
| 分子 | 名称: Ysh1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MERTNTTTFK FFSLGGSNEV GRSCHILQYK GKTVMLDAGI HPAYQGLASL PFYDEFDLSK VDILLISHF HLDHAASLPY VMQRTNFQGR VFMTHPTKAI YRWLLRDFVR VTSIGSSSSS M GTKDEGLF SDEDLVDSFD KIETVDYHST VDVNGIKFTA FHAGHVLGAA ...文字列: MERTNTTTFK FFSLGGSNEV GRSCHILQYK GKTVMLDAGI HPAYQGLASL PFYDEFDLSK VDILLISHF HLDHAASLPY VMQRTNFQGR VFMTHPTKAI YRWLLRDFVR VTSIGSSSSS M GTKDEGLF SDEDLVDSFD KIETVDYHST VDVNGIKFTA FHAGHVLGAA MFQIEIAGLR VL FTGDYSR EVDRHLNSAE VPPLSSNVLI VESTFGTATH EPRLNRERKL TQLIHSTVMR GGR VLLPVF ALGRAQEIML ILDEYWSQHA DELGGGQVPI FYASNLAKKC MSVFQTYVNM MNDD IRKKF RDSQTNPFIF KNISYLRNLE DFQDFGPSVM LASPGMLQSG LSRDLLERWC PEDKN LVLI TGYSIEGTMA KFIMLEPDTI PSINNPEITI PRRCQVEEIS FAAHVDFQEN LEFIEK ISA PNIILVHGEA NPMGRLKSAL LSNFASLKGT DNEVHVFNPR NCVEVDLEFQ GVKVAKA VG NIVNEIYKEE NVEIKEEIAA KIEPIKEENE DNLDSQAEKG LVDEEEHKDI VVSGILVS D DKNFELDFLS LSDLREHHPD LSTTILRERQ SVRVNCKKEL IYWHILQMFG EAEVLQDDD RVTNQEPKVK EESKDNLTNT GKLILQIMGD IKLTIVNTLA VVEWTQDLMN DTVADSIIAI LMNVDSAPA SVKLSSHSCD DHDHNNVQSN AQGKIDEVER VKQISRLFKE QFGDCFTLFL N KDEYASNK EETITGVVTI GKSTAKIDFN NMKILECNSN PLKGRVESLL NIGGNLVTPL C |
-分子 #2: Mpe1
| 分子 | 名称: Mpe1 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MSSTIFYRFK SQRNTSRILF DGTGLTVFDL KREIIQENKL GDGTDFQLKI YNPDTEEEYD DDAFVIPRS TSVIVKRSPA IKSFSVHSRL KGNVGAAALG NATRYVTGRP RVLQKRQHTA T TTANVSGT TEEERIASMF ATQENQWEQT QEEMSAATPV FFKSQTNKNS ...文字列: MSSTIFYRFK SQRNTSRILF DGTGLTVFDL KREIIQENKL GDGTDFQLKI YNPDTEEEYD DDAFVIPRS TSVIVKRSPA IKSFSVHSRL KGNVGAAALG NATRYVTGRP RVLQKRQHTA T TTANVSGT TEEERIASMF ATQENQWEQT QEEMSAATPV FFKSQTNKNS AQENEGPPPP GY MCYRCGG RDHWIKNCPT NSDPNFEGKR IRRTTGIPKK FLKSIEIDPE TMTPEEMAQR KIM ITDEGK FVVQVEDKQS WEDYQRKREN RQIDGDETIW RKGHFKDLPD DLKCPLTGGL LRQP VKTSK CCNIDFSKEA LENALVESDF VCPNCETRDI LLDSLVPDQD KEKEVETFLK KQEEL HGSS KDGNQPETKK MKLMDPTGTA GLNNNTSLPT SVNNGGTPVP PVPLPFGIPP FPMFPM PFM PPTATITNPH QADASPKK |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil, UltrAuFoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blotted for 10 s. | ||||||
| 詳細 | 350 nM complex was used. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / 球面収差補正装置: hexapole Cs-corrector / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-42 / 撮影したグリッド数: 1 / 実像数: 994 / 平均露光時間: 9.0 sec. / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X