+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0324 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative-stain map of CPFcore | |||||||||
 マップデータ マップデータ | Negative-stain map of CPFcore | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
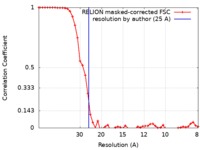
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 25.0 Å ネガティブ染色法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Hill CH / Boreikaite V / Kumar A / Casanal A / Kubik P / Degliesposti G / Maslen S / Mariani A / von Loeffelholz O / Girbig M ...Hill CH / Boreikaite V / Kumar A / Casanal A / Kubik P / Degliesposti G / Maslen S / Mariani A / von Loeffelholz O / Girbig M / Skehel M / Passmore LA | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2019 ジャーナル: Mol Cell / 年: 2019タイトル: Activation of the Endonuclease that Defines mRNA 3' Ends Requires Incorporation into an 8-Subunit Core Cleavage and Polyadenylation Factor Complex. 著者: Chris H Hill / Vytautė Boreikaitė / Ananthanarayanan Kumar / Ana Casañal / Peter Kubík / Gianluca Degliesposti / Sarah Maslen / Angelica Mariani / Ottilie von Loeffelholz / Mathias Girbig ...著者: Chris H Hill / Vytautė Boreikaitė / Ananthanarayanan Kumar / Ana Casañal / Peter Kubík / Gianluca Degliesposti / Sarah Maslen / Angelica Mariani / Ottilie von Loeffelholz / Mathias Girbig / Mark Skehel / Lori A Passmore /   要旨: Cleavage and polyadenylation factor (CPF/CPSF) is a multi-protein complex essential for formation of eukaryotic mRNA 3' ends. CPF cleaves pre-mRNAs at a specific site and adds a poly(A) tail. The ...Cleavage and polyadenylation factor (CPF/CPSF) is a multi-protein complex essential for formation of eukaryotic mRNA 3' ends. CPF cleaves pre-mRNAs at a specific site and adds a poly(A) tail. The cleavage reaction defines the 3' end of the mature mRNA, and thus the activity of the endonuclease is highly regulated. Here, we show that reconstitution of specific pre-mRNA cleavage with recombinant yeast proteins requires incorporation of the Ysh1 endonuclease into an eight-subunit "CPF" complex. Cleavage also requires the accessory cleavage factors IA and IB, which bind substrate pre-mRNAs and CPF, likely facilitating assembly of an active complex. Using X-ray crystallography, electron microscopy, and mass spectrometry, we determine the structure of Ysh1 bound to Mpe1 and the arrangement of subunits within CPF. Together, our data suggest that the active mRNA 3' end processing machinery is a dynamic assembly that is licensed to cleave only when all protein factors come together at the polyadenylation site. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0324.map.gz emd_0324.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0324-v30.xml emd-0324-v30.xml emd-0324.xml emd-0324.xml | 24.3 KB 24.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0324_fsc.xml emd_0324_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0324.png emd_0324.png | 47.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0324 http://ftp.pdbj.org/pub/emdb/structures/EMD-0324 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0324 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0324 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0324.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0324.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative-stain map of CPFcore | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.98 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CPFcore: a complex of Cft1-Pfs2-Yth1-Fip1-Pap1-Cft2-Ysh1-Mpe1
| 全体 | 名称: CPFcore: a complex of Cft1-Pfs2-Yth1-Fip1-Pap1-Cft2-Ysh1-Mpe1 |
|---|---|
| 要素 |
|
-超分子 #1: CPFcore: a complex of Cft1-Pfs2-Yth1-Fip1-Pap1-Cft2-Ysh1-Mpe1
| 超分子 | 名称: CPFcore: a complex of Cft1-Pfs2-Yth1-Fip1-Pap1-Cft2-Ysh1-Mpe1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
-分子 #1: Cft1
| 分子 | 名称: Cft1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MNVYDDVLDA TVVSHSLATH FTTSDYEELL VVRTNILSVY RPTRDGKLYL TDEFKFHGLI TDIGLIPQK DSPLSCLLLC TGVAKISILK FNTLTNSIDT LSLHYYEGKF KGKSLVELAK I STLRMDPG SSCALLFNND IIAFLPFHVN KNDDDEEEED EDENIDDSEL ...文字列: MNVYDDVLDA TVVSHSLATH FTTSDYEELL VVRTNILSVY RPTRDGKLYL TDEFKFHGLI TDIGLIPQK DSPLSCLLLC TGVAKISILK FNTLTNSIDT LSLHYYEGKF KGKSLVELAK I STLRMDPG SSCALLFNND IIAFLPFHVN KNDDDEEEED EDENIDDSEL IHSMNQKSQG TN TFNKRKR TKLGDKFTAP SVVLVASELY EGAKNIIDIQ FLKNFTKPTI ALLYQPKLVW AGN TTISKL PTQYVILTLN IQPAESATKI ESTTIAFVKE LPWDLHTIVP VSNGAIIVGT NELA FLDNT GVLQSTVLLN SFADKELQKT KIINNSSLEI MFREKNTTSI WIPSSKSKNG GSNND ETLL LMDLKSNIYY IQMEAEGRLL IKFDIFKLPI VNDLLKENSN PKCITRLNAT NSNKNM DLF IGFGSGNALV LRLNNLKSTI ETREAHNPSS GTNSLMDIND DDDEEMDDLY ADEAPEN GL TTNDSKGTVE TVQPFDIELL SSLRNVGPIT SLTVGKVSSI DDVVKGLPNP NKNEYSLV A TSGNGSGSHL TVIQTSVQPE IELALKFISI TQIWNLKIKG RDRYLITTDS TKSRSDIYE SDNNFKLHKG GRLRRDATTV YISMFGEEKR IIQVTTNHLY LYDTHFRRLT TIKFDYEVIH VSVMDPYIL VTVSRGDIKI FELEEKNKRK LLKVDLPEIL NEMVITSGLI LKSNMCNEFL I GLSKSQEE QLLFTFVTAD NQIIFFTKDH NDRIFQLNGV DQLNESLYIS TYQLGDEIVP DP SIKQVMI NKLGHDNKEE YLTILTFGGE IYQYRKLPQR RSRFYRNVTR NDLAITGAPD NAY AKGVSS IERIMHYFPD YNGYSVIFVT GSVPYILIKE DDSTPKIFKF GNIPLVSVTP WSER SVMCV DDIKNARVYT LTTDNMYYGN KLPLKQIKIS NVLDDYKTLQ KLVYHERAQL FLVSY CKRV PYEALGEDGE KVIGYDENVP HAEGFQSGIL LINPKSWKVI DKIDFPKNSV VNEMRS SMI QINSKTKRKR EYIIAGVANA TTEDTPPTGA FHIYDVIEVV PEPGKPDTNY KLKEIFQ EE VSGTVSTVCE VSGRFMISQS QKVLVRDIQE DNSVIPVAFL DIPVFVTDSK SFGNLLII G DAMQGFQFIG FDAEPYRMIS LGRSMSKFQT MSLEFLVNGG DMYFAATDAD RNVHVLKYA PDEPNSLSGQ RLVHCSSFTL HSTNSCMMLL PRNEEFGSPQ VPSFQNVGGQ VDGSVFKIVP LSEEKYRRL YVIQQQIIDR ELQLGGLNPR MERLANDFYQ MGHSMRPMLD FNVIRRFCGL A IDRRKSIA QKAGRHAHFE AWRDIINIEF SMRSLCQGK |
-分子 #2: Pfs2
| 分子 | 名称: Pfs2 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MDGHNQNQYQ NQNQIQQSQQ PPLKKYVTQR RSVDVSSPYI NLYYNRRHGL PNLVVEPETS YTIDIMPPN AYRGRDRVIN LPSKFTHLSS NKVKHVIPAI QWTPEGRRLV VATYSGEFSL W NASSFTFE TLMQAHDSAV TTMKYSHDSD WMISGDADGM IKIWQPNFSM ...文字列: MDGHNQNQYQ NQNQIQQSQQ PPLKKYVTQR RSVDVSSPYI NLYYNRRHGL PNLVVEPETS YTIDIMPPN AYRGRDRVIN LPSKFTHLSS NKVKHVIPAI QWTPEGRRLV VATYSGEFSL W NASSFTFE TLMQAHDSAV TTMKYSHDSD WMISGDADGM IKIWQPNFSM VKEIDAAHTE SI RDMAFSS NDSKFVTCSD DNILKIWNFS NGKQERVLSG HHWDVKSCDW HPEMGLIASA SKD NLVKLW DPRSGNCISS ILKFKHTVLK TRFQPTKGNL LMAISKDKSC RVFDIRYSMK ELMC VRDET DYMTLEWHPI NESMFTLACY DGSLKHFDLL QNLNEPILTI PYAHDKCITS LSYNP VGHI FATAAKDRTI RFWTRARPID PNAYDDPTYN NKKINGWFFG INNDINAVRE KSEFGA APP PPATLEPHAL PNMNGFINKK PRQEIPGIDS NIKSSTLPGL SI |
-分子 #3: Yth1
| 分子 | 名称: Yth1 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MSLIHPDTAK YPFKFEPFLR QEYSFSLDPD RPICEFYNSR EGPKSCPRGP LCPKKHVLPI FQNKIVCRH WLRGLCKKND QCEYLHEYNL RKMPECVFFS KNGYCTQSPD CQYLHIDPAS K IPKCENYE MGFCPLGSSC PRRHIKKVFC QRYMTGFCPL GKDECDMEHP ...文字列: MSLIHPDTAK YPFKFEPFLR QEYSFSLDPD RPICEFYNSR EGPKSCPRGP LCPKKHVLPI FQNKIVCRH WLRGLCKKND QCEYLHEYNL RKMPECVFFS KNGYCTQSPD CQYLHIDPAS K IPKCENYE MGFCPLGSSC PRRHIKKVFC QRYMTGFCPL GKDECDMEHP QFIIPDEGSK LR IKRDDEI NTRKMDEEKE RRLNAIINGE V |
-分子 #4: Fip1
| 分子 | 名称: Fip1 / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MSSSEDEDDK FLYGSDSELA LPSSKRSRDD EADAGASSNP DIVKRQKFDS PVEETPATAR DDRSDEDIY SDSSDDDSDS DLEVIISLGP DPTRLDAKLL DSYSTAATSS SKDVISVATD V SNTITKTS DERLITEGEA NQGVTATTVK ATESDGNVPK AMTGSIDLDK ...文字列: MSSSEDEDDK FLYGSDSELA LPSSKRSRDD EADAGASSNP DIVKRQKFDS PVEETPATAR DDRSDEDIY SDSSDDDSDS DLEVIISLGP DPTRLDAKLL DSYSTAATSS SKDVISVATD V SNTITKTS DERLITEGEA NQGVTATTVK ATESDGNVPK AMTGSIDLDK EGIFDSVGIT TI DPEVLKE KPWRQPGANL SDYFNYGFNE FTWMEYLHRQ EKLQQDYNPR RILMGLLSLQ QQG KLNSAN DTDSNLGNII DNNNNVNNAN MSNLNSNMGN SMSGTPNPPA PPMHPSFPPL PMFG SFPPF PMPGMMPPMN QQPNQNQNQN SK |
-分子 #5: Pap1
| 分子 | 名称: Pap1 / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MSSQKVFGIT GPVSTVGATA AENKLNDSLI QELKKEGSFE TEQETANRVQ VLKILQELAQ RFVYEVSKK KNMSDGMARD AGGKIFTYGS YRLGVHGPGS DIDTLVVVPK HVTREDFFTV F DSLLRERK ELDEIAPVPD AFVPIIKIKF SGISIDLICA RLDQPQVPLS ...文字列: MSSQKVFGIT GPVSTVGATA AENKLNDSLI QELKKEGSFE TEQETANRVQ VLKILQELAQ RFVYEVSKK KNMSDGMARD AGGKIFTYGS YRLGVHGPGS DIDTLVVVPK HVTREDFFTV F DSLLRERK ELDEIAPVPD AFVPIIKIKF SGISIDLICA RLDQPQVPLS LTLSDKNLLR NL DEKDLRA LNGTRVTDEI LELVPKPNVF RIALRAIKLW AQRRAVYANI FGFPGGVAWA MLV ARICQL YPNACSAVIL NRFFIILSEW NWPQPVILKP IEDGPLQVRV WNPKIYAQDR SHRM PVITP AYPSMCATHN ITESTKKVIL QEFVRGVQIT NDIFSNKKSW ANLFEKNDFF FRYKF YLEI TAYTRGSDEQ HLKWSGLVES KVRLLVMKLE VLAGIKIAHP FTKPFESSYC CPTEDD YEM IQDKYGSHKT ETALNALKLV TDENKEEESI KDAPKAYLST MYIGLDFNIE NKKEKVD IH IPCTEFVNLC RSFNEDYGDH KVFNLALRFV KGYDLPDEVF DENEKRPSKK SKRKNLDA R HETVKRSKSD AASGDNINGT TAAVDVN |
-分子 #6: Cft2
| 分子 | 名称: Cft2 / タイプ: protein_or_peptide / ID: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MTYKYNCCDD GSGTTVGSVV RFDNVTLLID PGWNPSKVSY EQCIKYWEKV IPEIDVIILS QPTIECLGA HSLLYYNFTS HFISRIQVYA TLPVINLGRV STIDSYASAG VIGPYDTNKL D LEDIEISF DHIVPLKYSQ LVDLRSRYDG LTLLAYNAGV CPGGSIWCIS ...文字列: MTYKYNCCDD GSGTTVGSVV RFDNVTLLID PGWNPSKVSY EQCIKYWEKV IPEIDVIILS QPTIECLGA HSLLYYNFTS HFISRIQVYA TLPVINLGRV STIDSYASAG VIGPYDTNKL D LEDIEISF DHIVPLKYSQ LVDLRSRYDG LTLLAYNAGV CPGGSIWCIS TYSEKLVYAK RW NHTRDNI LNAASILDAT GKPLSTLMRP SAIITTLDRF GSSQPFKKRS KIFKDTLKKG LSS DGSVII PVDMSGKFLD LFTQVHELLF ESTKINAHTQ VPVLILSYAR GRTLTYAKSM LEWL SPSLL KTWENRNNTS PFEIGSRIKI IAPNELSKYP GSKICFVSEV GALINEVIIK VGNSE KTTL ILTKPSFECA SSLDKILEIV EQDERNWKTF PEDGKSFLCD NYISIDTIKE EPLSKE ETE AFKVQLKEKK RDRNKKILLV KRESKKLANG NAIIDDTNGE RAMRNQDILV ENVNGVP PI DHIMGGDEDD DEEEENDNLL NLLKDNSEKS AAKKNTEVPV DIIIQPSAAS KHKMFPFN P AKIKKDDYGT VVDFTMFLPD DSDNVNQNSR KRPLKDGAKT TSPVNEEDNK NEEEDGYNM SDPISKRSKH RASRYSGFSG TGEAENFDNL DYLKIDKTLS KRTISTVNVQ LKCSVVILNL QSLVDQRSA SIIWPSLKSR KIVLSAPKQI QNEEITAKLI KKNIEVVNMP LNKIVEFSTT I KTLDISID SNLDNLLKWQ RISDSYTVAT VVGRLVKESL PQVNNHQKTA SRSKLVLKPL HG SSRSHKT GALSIGDVRL AQLKKLLTEK NYIAEFKGEG TLVINEKVAV RKINDAETII DGT PSELFD TVKKLVTDML AKI |
-分子 #7: Ysh1
| 分子 | 名称: Ysh1 / タイプ: protein_or_peptide / ID: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MERTNTTTFK FFSLGGSNEV GRSCHILQYK GKTVMLDAGI HPAYQGLASL PFYDEFDLSK VDILLISHF HLDHAASLPY VMQRTNFQGR VFMTHPTKAI YRWLLRDFVR VTSIGSSSSS M GTKDEGLF SDEDLVDSFD KIETVDYHST VDVNGIKFTA FHAGHVLGAA ...文字列: MERTNTTTFK FFSLGGSNEV GRSCHILQYK GKTVMLDAGI HPAYQGLASL PFYDEFDLSK VDILLISHF HLDHAASLPY VMQRTNFQGR VFMTHPTKAI YRWLLRDFVR VTSIGSSSSS M GTKDEGLF SDEDLVDSFD KIETVDYHST VDVNGIKFTA FHAGHVLGAA MFQIEIAGLR VL FTGDYSR EVDRHLNSAE VPPLSSNVLI VESTFGTATH EPRLNRERKL TQLIHSTVMR GGR VLLPVF ALGRAQEIML ILDEYWSQHA DELGGGQVPI FYASNLAKKC MSVFQTYVNM MNDD IRKKF RDSQTNPFIF KNISYLRNLE DFQDFGPSVM LASPGMLQSG LSRDLLERWC PEDKN LVLI TGYSIEGTMA KFIMLEPDTI PSINNPEITI PRRCQVEEIS FAAHVDFQEN LEFIEK ISA PNIILVHGEA NPMGRLKSAL LSNFASLKGT DNEVHVFNPR NCVEVDLEFQ GVKVAKA VG NIVNEIYKEE NVEIKEEIAA KIEPIKEENE DNLDSQAEKG LVDEEEHKDI VVSGILVS D DKNFELDFLS LSDLREHHPD LSTTILRERQ SVRVNCKKEL IYWHILQMFG EAEVLQDDD RVTNQEPKVK EESKDNLTNT GKLILQIMGD IKLTIVNTLA VVEWTQDLMN DTVADSIIAI LMNVDSAPA SVKLSSHSCD DHDHNNVQSN AQGKIDEVER VKQISRLFKE QFGDCFTLFL N KDEYASNK EETITGVVTI GKSTAKIDFN NMKILECNSN PLKGRVESLL NIGGNLVTPL C |
-分子 #8: Mpe1
| 分子 | 名称: Mpe1 / タイプ: protein_or_peptide / ID: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: MSSTIFYRFK SQRNTSRILF DGTGLTVFDL KREIIQENKL GDGTDFQLKI YNPDTEEEYD DDAFVIPRS TSVIVKRSPA IKSFSVHSRL KGNVGAAALG NATRYVTGRP RVLQKRQHTA T TTANVSGT TEEERIASMF ATQENQWEQT QEEMSAATPV FFKSQTNKNS ...文字列: MSSTIFYRFK SQRNTSRILF DGTGLTVFDL KREIIQENKL GDGTDFQLKI YNPDTEEEYD DDAFVIPRS TSVIVKRSPA IKSFSVHSRL KGNVGAAALG NATRYVTGRP RVLQKRQHTA T TTANVSGT TEEERIASMF ATQENQWEQT QEEMSAATPV FFKSQTNKNS AQENEGPPPP GY MCYRCGG RDHWIKNCPT NSDPNFEGKR IRRTTGIPKK FLKSIEIDPE TMTPEEMAQR KIM ITDEGK FVVQVEDKQS WEDYQRKREN RQIDGDETIW RKGHFKDLPD DLKCPLTGGL LRQP VKTSK CCNIDFSKEA LENALVESDF VCPNCETRDI LLDSLVPDQD KEKEVETFLK KQEEL HGSS KDGNQPETKK MKLMDPTGTA GLNNNTSLPT SVNNGGTPVP PVPLPFGIPP FPMFPM PFM PPTATITNPH QADASPKK |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 構成要素:
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate 詳細: Three microliters of sample was applied to the support and allowed to adsorb for 60 s before wicking away with filter paper. Grids were then applied sequentially to two 30 ul drops of 2% w/v ...詳細: Three microliters of sample was applied to the support and allowed to adsorb for 60 s before wicking away with filter paper. Grids were then applied sequentially to two 30 ul drops of 2% w/v uranyl acetate, first to wash (quick) and then to stain (30 s). Excess stain was then wicked away with filter paper until dry. | ||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 4.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR 詳細: Grids were CF400-CU-UL from Electron Microscopy Sciences. Thin layer continuous carbon over 400-mesh copper. Manually inspected before use (with a light microscope) | ||||||||||
| 詳細 | 250 nM complex was used |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): -0.6 µm / 最小 デフォーカス(公称値): -0.6 µm / 倍率(公称値): 26000 Bright-field microscopy / 最大 デフォーカス(公称値): -0.6 µm / 最小 デフォーカス(公称値): -0.6 µm / 倍率(公称値): 26000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) 平均露光時間: 2.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー