+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0308 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rea1 Wild type ADP state (tail part) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein-RNA complex remodeling /  regulation of ribosomal subunit export from nucleus / preribosome, large subunit precursor / regulation of ribosomal subunit export from nucleus / preribosome, large subunit precursor /  ribosomal large subunit export from nucleus / rRNA processing / ribosomal large subunit export from nucleus / rRNA processing /  ribosomal large subunit assembly / ribosomal large subunit assembly /  核小体 / 核小体 /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  核質 ...protein-RNA complex remodeling / 核質 ...protein-RNA complex remodeling /  regulation of ribosomal subunit export from nucleus / preribosome, large subunit precursor / regulation of ribosomal subunit export from nucleus / preribosome, large subunit precursor /  ribosomal large subunit export from nucleus / rRNA processing / ribosomal large subunit export from nucleus / rRNA processing /  ribosomal large subunit assembly / ribosomal large subunit assembly /  核小体 / 核小体 /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  ATP binding / ATP binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) / Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) /   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
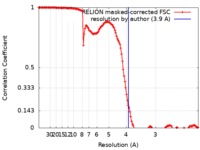
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Sosnowski P / Urnavicius L / Boland A / Fagiewicz R / Busselez J / Papai G / Schmidt H | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: The CryoEM structure of the ribosome maturation factor Rea1. 著者: Piotr Sosnowski / Linas Urnavicius / Andreas Boland / Robert Fagiewicz / Johan Busselez / Gabor Papai / Helgo Schmidt /   要旨: The biogenesis of 60S ribosomal subunits is initiated in the nucleus where rRNAs and proteins form pre-60S particles. These pre-60S particles mature by transiently interacting with various assembly ...The biogenesis of 60S ribosomal subunits is initiated in the nucleus where rRNAs and proteins form pre-60S particles. These pre-60S particles mature by transiently interacting with various assembly factors. The ~5000 amino-acid AAA+ ATPase Rea1 (or Midasin) generates force to mechanically remove assembly factors from pre-60S particles, which promotes their export to the cytosol. Here we present three Rea1 cryoEM structures. We visualise the Rea1 engine, a hexameric ring of AAA+ domains, and identify an α-helical bundle of AAA2 as a major ATPase activity regulator. The α-helical bundle interferes with nucleotide-induced conformational changes that create a docking site for the substrate binding MIDAS domain on the AAA +ring. Furthermore, we reveal the architecture of the Rea1 linker, which is involved in force generation and extends from the AAA+ ring. The data presented here provide insights into the mechanism of one of the most complex ribosome maturation factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0308.map.gz emd_0308.map.gz | 11.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0308-v30.xml emd-0308-v30.xml emd-0308.xml emd-0308.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0308_fsc.xml emd_0308_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0308.png emd_0308.png | 36.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0308 http://ftp.pdbj.org/pub/emdb/structures/EMD-0308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0308 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0308 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0308.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0308.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rea1 (MIDASIN) tail with ADP
| 全体 | 名称: Rea1 (MIDASIN) tail with ADP |
|---|---|
| 要素 |
|
-超分子 #1: Rea1 (MIDASIN) tail with ADP
| 超分子 | 名称: Rea1 (MIDASIN) tail with ADP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) Saccharomyces cerevisiae (strain ATCC 204508 / S288c) (パン酵母) |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-分子 #1: Midasin,Midasin,Midasin
| 分子 | 名称: Midasin,Midasin,Midasin / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c Saccharomyces cerevisiae (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 192.045828 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: PIEESLAAVI PISHLGEVGK WANNVLNCTE YSEKKIAERL YVFITFLTDM GVLEKINNLY KPANLKFQKA LGLHDKQLTE ETVSLTLNE YVLPTVSKYS DKIKSPESLY LLSSLRLLLN SLNALKLINE KSTHGKIDEL TYIELSAAAF NGRHLKNIPR I PIFCILYN ...文字列: PIEESLAAVI PISHLGEVGK WANNVLNCTE YSEKKIAERL YVFITFLTDM GVLEKINNLY KPANLKFQKA LGLHDKQLTE ETVSLTLNE YVLPTVSKYS DKIKSPESLY LLSSLRLLLN SLNALKLINE KSTHGKIDEL TYIELSAAAF NGRHLKNIPR I PIFCILYN ILTVMSENLK TESLFCGSNQ YQYYWDLLVI VIAALETAVT KDEARLRVYK ELIDSWIASV KSKSDIEITP FL NINLEFT DVLQLSRGHS ITLLWDIFRK NYPTTSNSWL AFEKLINLSE KFDKVRLLQF SESYNSIKDL MDVFRLLNDD VLN NKLSEF NLLLSKLEDG INELELISNK FLNKRKHYFA DEFDNLIRYT FSVDTAELIK ELAPASSLAT QKLTKLITNK YNYP PIFDV LWTEKNAKLT SFTSTIFSSQ FLEDVVRKSN NLKSFSGNQI KQSISDAELL LSSTIKCSPN LLKSQMEYYK NMLLS WLRK VIDIHVGGDC LKLTLKELCS LIEEKTASET RVTFAEYIFP ALDLAESSKS LEELGEAWIT FGTGLLLLFV PDSPYD PAI HDYVLYDLFL KTKTFSQNLM KSWRNVRKVI SGDEEIFTEK LINTISDDDA PQSPRVYRTG MSIDSLFDEW MAFLSST MS SRQIKELVSS YKCNSDQSDR RLEMLQQNSA HFLNRLESGY SKFADLNDIL AGYIYSINFG FDLLKLQKSK DRASFQIS P LWSMDPINIS CAENVLSAYH ELSRFFKKGD MEDTSIEKVL MYFLTLFKFH KRDTNLLEIF EAALYTLYSR WSVRRFRQE QEENEKSNMF KFNDNSDDYE ADFRKLFPDY EDTALVTNEK DISSPENLDD IYFKLADTYI SVFDKDHDAN FSSELKSGAI ITTILSEDL KNTRIEELKS GSLSAVINTL DAETQSFKNT EVFGNIDFYH DFSIPEFQKA GDIIETVLKS VLKLLKQWPE H ATLKELYR VSQEFLNYPI KTPLARQLQK IEQIYTYLAE WEKYASSEVS LNNTVKLITD LIVSWRKLEL RTWKGLFNSE DA KTRKSIG KWWFYLYESI VISNFVSEKK ETAPNATLLV SSLNLFFSKS TLGEFNARLD LVKAFYKHIQ LIGLRSSKIA GLL HNTIKF YYQFKPLIDE RITNGKKSLE KEIDDIILLA SWKDVNVDAL KQSSRKSHNN LYKIVRKYRD LLNGDAKTII EAGL LY(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) RNIDTVASNM DSYLEKISSQ EFPNFADLAS DFYAEAERLR KETPNVYTKE NKKRLAYLKT QKSKLLGDAL KELRRIGLKV NFREDIQKV QSSTTTILAN IAPFNNEYLN SSDAFFFKIL DLLPKLRSAA SNPSDDIPVA AIERGMALAQ SLMFSLITVR H PLSEFTND YCKINGMMLD LEHFTCLKGD IVHSSLKANV DNVRLFEKWL PSLLDYAAQT LSVISKYSAT SEQQKILLDA KS TLSSFFV HFNSSRIFDS SFIESYSRFE LFINELLKKL ENAKETGNAF VFDIIIEWIK ANKGGPIKKE QKRGPSVEDV EQA FRRTFT SIILSFQKVI GDGIESISET DDNWLSASFK KVMVNVKLLR SSVVSKNIET ALSLLKDFDF TTTESIYVKS VISF TLPVI TRYYNAMTVV LERSRIYYTN TSRGMYILST ILHSLAKN |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
詳細: ADP was added 5 minute before the plunging | ||||||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 105000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 0.01 mm / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 105000 |
| 特殊光学系 | 球面収差補正装置: Titan Krios Cs Corrector / エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3712 pixel / デジタル化 - サイズ - 縦: 3840 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 2-35 / 撮影したグリッド数: 14 / 実像数: 23230 / 平均露光時間: 0.2 sec. / 平均電子線量: 46.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー