+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0245 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the in vitro assembled bacteriophage phi6 P1P4 complex | |||||||||
 マップデータ マップデータ | Bacteriophage phi6 in vitro assembled P1P4 particle | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) | |||||||||
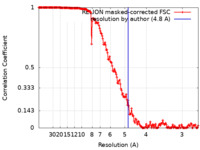
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.8 Å クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Huiskonen JT / Ilca SL | |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2018 ジャーナル: mBio / 年: 2018タイトル: Dual Role of a Viral Polymerase in Viral Genome Replication and Particle Self-Assembly. 著者: Xiaoyu Sun / Serban L Ilca / Juha T Huiskonen / Minna M Poranen /   要旨: Double-stranded RNA (dsRNA) viruses package several RNA-dependent RNA polymerases (RdRp) together with their dsRNA genome into an icosahedral protein capsid known as the polymerase complex. This ...Double-stranded RNA (dsRNA) viruses package several RNA-dependent RNA polymerases (RdRp) together with their dsRNA genome into an icosahedral protein capsid known as the polymerase complex. This structure is highly conserved among dsRNA viruses but is not found in any other virus group. RdRp subunits typically interact directly with the main capsid proteins, close to the 5-fold symmetric axes, and perform viral genome replication and transcription within the icosahedral protein shell. In this study, we utilized phage Φ6, a well-established virus self-assembly model, to probe the potential roles of the RdRp in dsRNA virus assembly. We demonstrated that Φ6 RdRp accelerates the polymerase complex self-assembly process and contributes to its conformational stability and integrity. We highlight the role of specific amino acid residues on the surface of the RdRp in its incorporation during the self-assembly reaction. Substitutions of these residues reduce RdRp incorporation into the polymerase complex during the self-assembly reaction. Furthermore, we determined that the overall transcription efficiency of the Φ6 polymerase complex increased when the number of RdRp subunits exceeded the number of genome segments. These results suggest a mechanism for RdRp recruitment in the polymerase complex and highlight its novel role in virion assembly, in addition to the canonical RNA transcription and replication functions. Double-stranded RNA viruses infect a wide spectrum of hosts, including animals, plants, fungi, and bacteria. Yet genome replication mechanisms of these viruses are conserved. During the infection cycle, a proteinaceous capsid, the polymerase complex, is formed. An essential component of this capsid is the viral RNA polymerase that replicates and transcribes the enclosed viral genome. The polymerase complex structure is well characterized for many double-stranded RNA viruses. However, much less is known about the hierarchical molecular interactions that take place in building up such complexes. Using the bacteriophage Φ6 self-assembly system, we obtained novel insights into the processes that mediate polymerase subunit incorporation into the polymerase complex for generation of functional structures. The results presented pave the way for the exploitation and engineering of viral self-assembly processes for biomedical and synthetic biology applications. An understanding of viral assembly processes at the molecular level may also facilitate the development of antivirals that target viral capsid assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0245.map.gz emd_0245.map.gz | 395.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0245-v30.xml emd-0245-v30.xml emd-0245.xml emd-0245.xml | 12.6 KB 12.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0245_fsc.xml emd_0245_fsc.xml | 16.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0245.png emd_0245.png | 221.6 KB | ||
| マスクデータ |  emd_0245_msk_1.map emd_0245_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0245 http://ftp.pdbj.org/pub/emdb/structures/EMD-0245 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0245 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0245 | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0245.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0245.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Bacteriophage phi6 in vitro assembled P1P4 particle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0245_msk_1.map emd_0245_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Pseudomonas phage phi6
| 全体 | 名称:   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Pseudomonas phage phi6
| 超分子 | 名称: Pseudomonas phage phi6 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10879 / 生物種: Pseudomonas phage phi6 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:   Pseudomonas syringae (シュードモナス・シリンガエ) Pseudomonas syringae (シュードモナス・シリンガエ) |
| Host system | 生物種:   Escherichia coli (大腸菌) / 組換株: JM109 Escherichia coli (大腸菌) / 組換株: JM109 |
-分子 #1: P1 protein from bacteriophage phi6
| 分子 | 名称: P1 protein from bacteriophage phi6 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MFNLKVKDL NGSARGLTQA FAIGELKNQL SVGALQLPLQ FTRTFSASMT S ELLWEVGK GNIDPVMYAR LFFQYAQAGG ALSVDELVNQ FTEYHQSTAC NP EIWRKLT AYITGSSNRA IKADAVGKVP PTAILEQLRT LAPSEHELFH HIT TDFVCH VLSPLGFILP ...文字列: MFNLKVKDL NGSARGLTQA FAIGELKNQL SVGALQLPLQ FTRTFSASMT S ELLWEVGK GNIDPVMYAR LFFQYAQAGG ALSVDELVNQ FTEYHQSTAC NP EIWRKLT AYITGSSNRA IKADAVGKVP PTAILEQLRT LAPSEHELFH HIT TDFVCH VLSPLGFILP DAAYVYRVGR TATYPNFYAL VDCVRASDLR RMLT ALSSV DSKMLQATFK AKGALAPALI SQHLANAATT AFERSRGNFD ANAVV SSVL TILGRLWSPS TPKELDPSAR LRNTNGIDQL RSNLALFIAY QDMVKQ RGR AEVIFSDEEL SSTIIPWFIE AMSEVSPFKL RPINETTSYI GQTSAID HM GQPSHVVVYE DWQFAKEITA FTPVKLANNS NQRFLDVEPG ISDRMSAT L APIGNTFAVS AFVKNRTAVY EAVSQRGTVN SNGAEMTLGF PSVVERDYA LDRDPMVAIA ALRTGIVDES LEARASNDLK RSMFNYYAAV MHYAVAHNPE VVVSEHQGV AAEQGSLYLV WNVRTELRIP VGYNAIEGGS IRTPEPLEAI A YNKPIQPS EVLQAKVLDL ANHTTSIHIW PWHEASTEFA YEDAYSVTIR NK RYTAEVK EFELLGLGQR RERVRILKPT VAHAIIQMWY SWFVEDDRTL AAA RRTSRD DAEKLAIDGR RMQNAVTLLR KIEMIGTTGI GASAVHLAQS RIVD QMAGR GLIDDSSDLH VGINRHRIRI WAGLAVLQMM GLLSRSEAEA LTKVL GDSN ALGMVVATTD IDPSL |
-分子 #2: P4 protein from bacteriophage phi6
| 分子 | 名称: P4 protein from bacteriophage phi6 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: DEXTRO |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MPIVVTQAH IDRVGIAADL LDASPVSLQV LGRPTAINTV VIKTYIAAVM E LASKQGGS LAGVDIRPSV LLKDTAIFTK PKAKSADVES DVDVLDTGIY SV PGLARKP VTHRWPSEGI YSGVTALMGA TGSGKSITLN EKLRPDVLIR WGE VAEAYD ELDTAVHIST ...文字列: MPIVVTQAH IDRVGIAADL LDASPVSLQV LGRPTAINTV VIKTYIAAVM E LASKQGGS LAGVDIRPSV LLKDTAIFTK PKAKSADVES DVDVLDTGIY SV PGLARKP VTHRWPSEGI YSGVTALMGA TGSGKSITLN EKLRPDVLIR WGE VAEAYD ELDTAVHIST LDEMLIVCIG LGALGFNVAV DSVRPLLFRL KGAA SAGGI VAVFYSLLTD ISNLFTQYDC SVVMVVNPMV DAEKIEYVFG QVMAS TVGA ILCADGNVSR TMFRTNKGRI FNGAAPLAAD THMPSMDRPT SMKALD HTS IASVAPLERG SVDTDDRNSA PRRGANFSL |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.0 mm Bright-field microscopy / Cs: 2.0 mm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 1.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X