+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0093 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human HCN4 hyperpolarization-activated cyclic nucleotide-gated ion channel | |||||||||
 マップデータ マップデータ | Cryosparc sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報voltage-gated potassium channel activity involved in SA node cell action potential depolarization / sinoatrial node development /  HCN channels / HCN channels /  HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential ...voltage-gated potassium channel activity involved in SA node cell action potential depolarization / sinoatrial node development / HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential ...voltage-gated potassium channel activity involved in SA node cell action potential depolarization / sinoatrial node development /  HCN channels / HCN channels /  HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential / sodium ion import across plasma membrane / HCN channel complex / regulation of cardiac muscle cell action potential involved in regulation of contraction / SA node cell action potential / membrane depolarization during SA node cell action potential / intracellularly cAMP-activated cation channel activity / cellular response to cGMP / membrane depolarization during cardiac muscle cell action potential / sodium ion import across plasma membrane /  voltage-gated sodium channel activity / voltage-gated sodium channel activity /  循環器 / 循環器 /  regulation of membrane depolarization / potassium ion import across plasma membrane / regulation of heart rate by cardiac conduction / regulation of membrane depolarization / potassium ion import across plasma membrane / regulation of heart rate by cardiac conduction /  voltage-gated potassium channel activity / monoatomic cation transport / sodium ion transmembrane transport / regulation of cardiac muscle contraction / voltage-gated potassium channel activity / monoatomic cation transport / sodium ion transmembrane transport / regulation of cardiac muscle contraction /  cAMP binding / cellular response to cAMP / potassium ion transmembrane transport / cAMP binding / cellular response to cAMP / potassium ion transmembrane transport /  regulation of membrane potential / regulation of membrane potential /  regulation of heart rate / regulation of heart rate /  muscle contraction / muscle contraction /  神経繊維 / 神経繊維 /  樹状突起 / perinuclear region of cytoplasm / identical protein binding / 樹状突起 / perinuclear region of cytoplasm / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
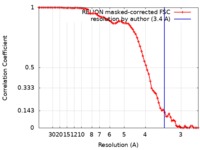
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Shintre CA / Pike ACW / Tessitore A / Young M / Bushell SR / Strain-Damerell C / Mukhopadhyay S / Burgess-Brown NA / Huiskonen JT / Arrowsmith CH ...Shintre CA / Pike ACW / Tessitore A / Young M / Bushell SR / Strain-Damerell C / Mukhopadhyay S / Burgess-Brown NA / Huiskonen JT / Arrowsmith CH / Edwards AM / Bountra C / Carpenter EP / Structural Genomics Consortium (SGC) | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of human HCN4 hyperpolarization-activated cyclic nucleotide-gated ion channel 著者: Shintre CA / Pike ACW / Tessitore A / Young M / Bushell SR / Strain-Damerell C / Mukhopadhyay S / Burgess-Brown NA / Huiskonen JT / Arrowsmith CH / Edwards AM / Bountra C / Carpenter EP | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0093.map.gz emd_0093.map.gz | 38 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0093-v30.xml emd-0093-v30.xml emd-0093.xml emd-0093.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0093_fsc.xml emd_0093_fsc.xml | 7.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0093.png emd_0093.png | 189.4 KB | ||
| マスクデータ |  emd_0093_msk_1.map emd_0093_msk_1.map | 40.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0093_additional.map.gz emd_0093_additional.map.gz emd_0093_half_map_1.map.gz emd_0093_half_map_1.map.gz emd_0093_half_map_2.map.gz emd_0093_half_map_2.map.gz | 20.3 MB 37.2 MB 37.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0093 http://ftp.pdbj.org/pub/emdb/structures/EMD-0093 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0093 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0093 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0093.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0093.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0093_msk_1.map emd_0093_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
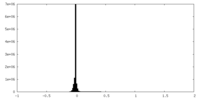
| 密度ヒストグラム |
-追加マップ: Cryosparc unsharpened map
| ファイル | emd_0093_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
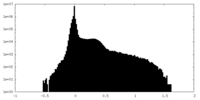
| 密度ヒストグラム |
-ハーフマップ: Half map1
| ファイル | emd_0093_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
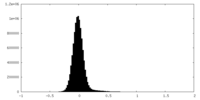
| 密度ヒストグラム |
-ハーフマップ: Half map2
| ファイル | emd_0093_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
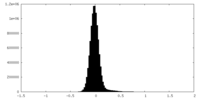
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Potassium/Sodium hyperpolarization-activated cyclic nucleotide-ga...
| 全体 | 名称: Potassium/Sodium hyperpolarization-activated cyclic nucleotide-gated ion channel 4 |
|---|---|
| 要素 |
|
-超分子 #1: Potassium/Sodium hyperpolarization-activated cyclic nucleotide-ga...
| 超分子 | 名称: Potassium/Sodium hyperpolarization-activated cyclic nucleotide-gated ion channel 4 タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
-分子 #1: Potassium/sodium hyperpolarization-activated cyclic nucleotide-ga...
| 分子 | 名称: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 4 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 60.836754 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: SMLPEAEVRL GQAGFMQRQF GAMLQPGVNK FSLRMFGSQK AVEREQERVK SAGFWIIHPY SDFRFYWDLT MLLLMVGNLI IIPVGITFF KDENTTPWIV FNVVSDTFFL IDLVLNFRTG IVVEDNTEII LDPQRIKMKY LKSWFMVDFI SSIPVDYIFL I VETRIDSE ...文字列: SMLPEAEVRL GQAGFMQRQF GAMLQPGVNK FSLRMFGSQK AVEREQERVK SAGFWIIHPY SDFRFYWDLT MLLLMVGNLI IIPVGITFF KDENTTPWIV FNVVSDTFFL IDLVLNFRTG IVVEDNTEII LDPQRIKMKY LKSWFMVDFI SSIPVDYIFL I VETRIDSE VYKTARALRI VRFTKILSLL RLLRLSRLIR YIHQWEEIFH MTYDLASAVV RIVNLIGMML LLCHWDGCLQ FL VPMLQDF PDDCWVSINN MVNNSWGKQY SYALFKAMSH MLCIGYGRQA PVGMSDVWLT MLSMIVGATC YAMFIGHATA LIQ SLDSSR RQYQEKYKQV EQYMSFHKLP PDTRQRIHDY YEHRYQGKMF DEESILGELS EPLREEIINF NCRKLVASMP LFAN ADPNF VTSMLTKLRF EVFQPGDYII REGTIGKKMY FIQHGVVSVL TKGNKETKLA DGSYFGEICL LTRGRRTASV RADTY CRLY SLSVDNFNEV LEEYPMMRRA FETVALDRLD RIGKKNS |
-分子 #2: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 2 / コピー数: 28 / 式: PC1 |
|---|---|
| 分子量 | 理論値: 790.145 Da |
| Chemical component information |  ChemComp-PC1: |
-分子 #3: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOETHANOLAMINE
| 分子 | 名称: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOETHANOLAMINE / タイプ: ligand / ID: 3 / コピー数: 8 / 式: 3PE |
|---|---|
| 分子量 | 理論値: 748.065 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV / 詳細: blotted for 5.5s before plunge. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 37313 Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.3 µm / 倍率(公称値): 37313 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | EBIC TITAN KRIOS M02 |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-48 / 撮影したグリッド数: 1 / 実像数: 1294 / 平均露光時間: 12.0 sec. / 平均電子線量: 48.7 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model refined against the cryosparc b-factor sharpened map using default restraints |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-6gyn: |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X