+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0056 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Feline Calicivirus Strain F9 bound to a soluble ectodomain fragment of feline junctional adhesion molecule A - leading to assembly of a portal structure at a unique three-fold axis. | |||||||||
 マップデータ マップデータ | C3 reconstruction of Feline Calicivirus decorated with a soluble ectodomain fragment of the cellular receptor feline junctional adhesion molecule A. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報establishment of endothelial intestinal barrier /  regulation of membrane permeability / T=3 icosahedral viral capsid / intestinal absorption / bicellular tight junction / host cell cytoplasm / membrane => GO:0016020 / regulation of membrane permeability / T=3 icosahedral viral capsid / intestinal absorption / bicellular tight junction / host cell cytoplasm / membrane => GO:0016020 /  細胞接着 / 細胞接着 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Felis catus (イエネコ) / Felis catus (イエネコ) /   Feline calicivirus strain F9 (ネコカリシウイルス) / Feline calicivirus strain F9 (ネコカリシウイルス) /   FCV (ネコカリシウイルス) FCV (ネコカリシウイルス) | |||||||||
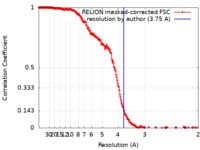
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.75 Å クライオ電子顕微鏡法 / 解像度: 3.75 Å | |||||||||
 データ登録者 データ登録者 | Conley MJ / Bhella D | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Calicivirus VP2 forms a portal-like assembly following receptor engagement. 著者: Michaela J Conley / Marion McElwee / Liyana Azmi / Mads Gabrielsen / Olwyn Byron / Ian G Goodfellow / David Bhella /  要旨: To initiate infection, many viruses enter their host cells by triggering endocytosis following receptor engagement. However, the mechanisms by which non-enveloped viruses escape the endosome are ...To initiate infection, many viruses enter their host cells by triggering endocytosis following receptor engagement. However, the mechanisms by which non-enveloped viruses escape the endosome are poorly understood. Here we present near-atomic-resolution cryo-electron microscopy structures for feline calicivirus both undecorated and labelled with a soluble fragment of its cellular receptor, feline junctional adhesion molecule A. We show that VP2, a minor capsid protein encoded by all caliciviruses, forms a large portal-like assembly at a unique three-fold axis of symmetry, following receptor engagement. This assembly-which was not detected in undecorated virions-is formed of twelve copies of VP2, arranged with their hydrophobic N termini pointing away from the virion surface. Local rearrangement at the portal site leads to the opening of a pore in the capsid shell. We hypothesize that the portal-like assembly functions as a channel for the delivery of the calicivirus genome, through the endosomal membrane, into the cytoplasm of a host cell, thereby initiating infection. VP2 was previously known to be critical for the production of infectious virus; our findings provide insights into its structure and function that advance our understanding of the Caliciviridae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0056.map.gz emd_0056.map.gz | 757.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0056-v30.xml emd-0056-v30.xml emd-0056.xml emd-0056.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0056_fsc.xml emd_0056_fsc.xml | 21 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0056.png emd_0056.png | 248.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0056 http://ftp.pdbj.org/pub/emdb/structures/EMD-0056 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0056 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0056 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6gsiMC  0054C  6gshC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10193 (タイトル: Calicivirus VP2 forms a portal to mediate endosome escape EMPIAR-10193 (タイトル: Calicivirus VP2 forms a portal to mediate endosome escapeData size: 867.0 Data #1: Motion corrected micrographs of feline calicivirus strain F9 decorated with feline junctional adhesion molecule A [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0056.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0056.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C3 reconstruction of Feline Calicivirus decorated with a soluble ectodomain fragment of the cellular receptor feline junctional adhesion molecule A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.065 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Feline calicivirus strain F9
| 全体 | 名称:   Feline calicivirus strain F9 (ネコカリシウイルス) Feline calicivirus strain F9 (ネコカリシウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Feline calicivirus strain F9
| 超分子 | 名称: Feline calicivirus strain F9 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Virus was propagated in Crandell Reese Feline Kidney cells ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Felis catus (イエネコ) Felis catus (イエネコ) |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 400.0 Å / T番号(三角分割数): 3 |
-超分子 #2: Feline calicivirus strain F9
| 超分子 | 名称: Feline calicivirus strain F9 / タイプ: virus / ID: 2 / 親要素: 1 / 含まれる分子: #1 / 詳細: Icosahedral Capsid / NCBI-ID: 11981 / 生物種: Feline calicivirus strain F9 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Felis catus (イエネコ) Felis catus (イエネコ) |
| ウイルス殻 | Shell ID: 2 / 名称: Capsid / 直径: 400.0 Å / T番号(三角分割数): 3 |
-超分子 #3: feline junctional adhesion molecule A
| 超分子 | 名称: feline junctional adhesion molecule A / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 詳細: Soluble ectodomain fragment comprising Ig-like domains D1 and D2 |
|---|---|
| 由来(天然) | 生物種:   Felis catus (イエネコ) Felis catus (イエネコ) |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ)組換細胞: CHO cells |
-超分子 #4: VP2 - portal.
| 超分子 | 名称: VP2 - portal. / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 詳細: Postulated to mediate endosome escape, this virion component is only present on the outer surface of the capsid following receptor engagement. |
|---|---|
| 由来(天然) | 生物種:   Feline calicivirus strain F9 (ネコカリシウイルス) Feline calicivirus strain F9 (ネコカリシウイルス) |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   FCV (ネコカリシウイルス) FCV (ネコカリシウイルス) |
| 分子量 | 理論値: 73.346664 KDa |
| 配列 | 文字列: MCSTCANVLK YYNWDPHFRL VIDPNKFLSV GFCDNPLMCC YPELLPEFGT VWDCDQSPLQ IYLESILGDD EWASTYEAID PAVPPMHWD AAGKIFQPHP GVLMHHLIGE VAKAWDPNLP MFRLEADGDG SITAPEQGTP VGGVIAEPSA QMSAAADMAT G KSVDSEWE ...文字列: MCSTCANVLK YYNWDPHFRL VIDPNKFLSV GFCDNPLMCC YPELLPEFGT VWDCDQSPLQ IYLESILGDD EWASTYEAID PAVPPMHWD AAGKIFQPHP GVLMHHLIGE VAKAWDPNLP MFRLEADGDG SITAPEQGTP VGGVIAEPSA QMSAAADMAT G KSVDSEWE AFFSFHTSVN WSTSETQGKI LFKQSLGPLL NPYLEHLAKL YVAWSGSIDV RFSISGSGVF GGKLAAIVVP PG VDPVQST SMLQYPHVLF DARQVEPVIF SIPDLRSTLY HLMSDTDTTS LVIMVYNDLI NPYANDSNSS GCIVTVETKP GAD FKFHLL KPPGSMLTHG SVPSDLIPKS SSLWIGNRHW TDITDFVIRP FVFQANRHFD FNQETAGWST PRYRPITITI SEKN GAKLG IGVATDYIVP GIPDGWPDTT IPEKLTPAGD YAITNKSGND ITTAAGYDGA DVIVNNTNFK GMYICGSLQR AWGDK KISN TAFITTATKV DNAIEPSNVI DMTKIAVYQD THVGKEVQTS DDTLSLLGYT GIGEQAIGSD RDRVVRISVL PETGAR GGN HPIFYKNSIK LGYVIRSIDV FNSQILHTSR QLSLNHYLLP PDSFAVYRII DSNGSWFDIG IDSDGFSFVG VSSIGKL EF PLTASYMGIQ LAKIRLASNI RSSMTKL |
-分子 #2: Junctional adhesion molecule A
| 分子 | 名称: Junctional adhesion molecule A / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Felis catus (イエネコ) Felis catus (イエネコ) |
| 分子量 | 理論値: 22.18065 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: AVYTSEPDVR VPEDKPAKLS CSYSGFSNPR VEWKFAHGDI TSLVCYKNKI TASYADRVTF SHSGITFHSV TRKDTGTYTC MVSDDGGNT YGEVSVQLTV LVPPSKPTVH IPSSATIGSR AVLTCSEKDG SPPSEYYWFK DGVRMPLEPK GNRAFSNSSY S LNEKTGEL ...文字列: AVYTSEPDVR VPEDKPAKLS CSYSGFSNPR VEWKFAHGDI TSLVCYKNKI TASYADRVTF SHSGITFHSV TRKDTGTYTC MVSDDGGNT YGEVSVQLTV LVPPSKPTVH IPSSATIGSR AVLTCSEKDG SPPSEYYWFK DGVRMPLEPK GNRAFSNSSY S LNEKTGEL VFDPVSAWDT GEYTCEAQNG YGMPMRSEAV RMEA |
-分子 #3: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   FCV (ネコカリシウイルス) FCV (ネコカリシウイルス) |
| 分子量 | 理論値: 12.209838 KDa |
| 配列 | 文字列: MNSILGLIDT VTNTIGKAQQ IELDKAALGQ QRELALKRMK LDHQALNNQV EQFNKILEQR VQGPIQSVRL ARAAGFRVDP YSYTDQNFY DDQLNAIRLS YRNLFKN |
-分子 #4: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 詳細: Phosphate buffered saline |
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Purified virions were incubated in the presence of purified ectodomain fragments. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 75000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 13865 / 平均電子線量: 63.0 e/Å2 詳細: Each micrograph was recorded as a movie of 50 individual fractions with a total dose of 63 e/angstrom squared |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー