登録情報 データベース : EMDB / ID : EMD-0026タイトル Neurturin-GFRa2-RET extracellular complex Consensus refinement with C2 symmetry applied. 複合体 : Neurturin-GFRa2-RET extracellular complex複合体 : Neurturinタンパク質・ペプチド : Neurturin複合体 : GDNF family receptor alpha-2タンパク質・ペプチド : GDNF family receptor alpha-2複合体 : Proto-oncogene tyrosine-protein kinase receptor Retタンパク質・ペプチド : Proto-oncogene tyrosine-protein kinase receptor Ret機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 6.3 Å Bigalke JM / Aibara S / Sandmark J / Amunts A 資金援助 Organization Grant number 国 Knut and Alice Wallenberg Foundation Swedish Research Council NT_2015-04107
ジャーナル : Sci Adv / 年 : 2019タイトル : Cryo-EM structure of the activated RET signaling complex reveals the importance of its cysteine-rich domain.著者 : Janna M Bigalke / Shintaro Aibara / Robert Roth / Göran Dahl / Euan Gordon / Sarah Dorbéus / A Amunts / Jenny Sandmark / 要旨 : Signaling through the receptor tyrosine kinase RET is essential during normal development. Both gain- and loss-of-function mutations are involved in a variety of diseases, yet the molecular details ... Signaling through the receptor tyrosine kinase RET is essential during normal development. Both gain- and loss-of-function mutations are involved in a variety of diseases, yet the molecular details of receptor activation have remained elusive. We have reconstituted the complete extracellular region of the RET signaling complex together with Neurturin (NRTN) and GFRα2 and determined its structure at 5.7-Å resolution by cryo-EM. The proteins form an assembly through RET-GFRα2 and RET-NRTN interfaces. Two key interaction points required for RET extracellular domain binding were observed: (i) the calcium-binding site in RET that contacts GFRα2 domain 3 and (ii) the RET cysteine-rich domain interaction with NRTN. The structure highlights the importance of the RET cysteine-rich domain and allows proposition of a model to explain how complex formation leads to RET receptor dimerization and its activation. This provides a framework for targeting RET activity and for further exploration of mechanisms underlying neurological diseases. 履歴 登録 2018年5月23日 - ヘッダ(付随情報) 公開 2018年8月22日 - マップ公開 2019年8月14日 - 更新 2019年12月18日 - 現状 2019年12月18日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 membrane protein proteolysis ...glial cell-derived neurotrophic factor receptor activity / glial cell-derived neurotrophic factor receptor binding / glial cell-derived neurotrophic factor receptor signaling pathway / Peyer's patch morphogenesis / positive regulation of metanephric glomerulus development / posterior midgut development / ureter maturation / embryonic epithelial tube formation / lymphocyte migration into lymphoid organs /
membrane protein proteolysis ...glial cell-derived neurotrophic factor receptor activity / glial cell-derived neurotrophic factor receptor binding / glial cell-derived neurotrophic factor receptor signaling pathway / Peyer's patch morphogenesis / positive regulation of metanephric glomerulus development / posterior midgut development / ureter maturation / embryonic epithelial tube formation / lymphocyte migration into lymphoid organs /  membrane protein proteolysis / positive regulation of peptidyl-serine phosphorylation of STAT protein / positive regulation of neuron maturation / neuron cell-cell adhesion /
membrane protein proteolysis / positive regulation of peptidyl-serine phosphorylation of STAT protein / positive regulation of neuron maturation / neuron cell-cell adhesion /  enteric nervous system development / nerve development /
enteric nervous system development / nerve development /  神経 /
神経 /  plasma membrane protein complex / neuron maturation / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / NCAM1 interactions / positive regulation of cell adhesion mediated by integrin /
plasma membrane protein complex / neuron maturation / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / NCAM1 interactions / positive regulation of cell adhesion mediated by integrin /  神経堤 / ureteric bud development / response to pain /
神経堤 / ureteric bud development / response to pain /  extrinsic component of membrane /
extrinsic component of membrane /  regulation of axonogenesis / homophilic cell adhesion via plasma membrane adhesion molecules / positive regulation of cell size / RET signaling /
regulation of axonogenesis / homophilic cell adhesion via plasma membrane adhesion molecules / positive regulation of cell size / RET signaling /  regulation of cell adhesion / cellular response to retinoic acid / NPAS4 regulates expression of target genes /
regulation of cell adhesion / cellular response to retinoic acid / NPAS4 regulates expression of target genes /  transmembrane receptor protein tyrosine kinase activity /
transmembrane receptor protein tyrosine kinase activity /  軸索誘導 /
軸索誘導 /  growth factor activity / positive regulation of neuron projection development /
growth factor activity / positive regulation of neuron projection development /  receptor tyrosine kinase binding /
receptor tyrosine kinase binding /  受容体型チロシンキナーゼ / neuron projection development /
受容体型チロシンキナーゼ / neuron projection development /  cell surface receptor protein tyrosine kinase signaling pathway / activation of cysteine-type endopeptidase activity involved in apoptotic process /
cell surface receptor protein tyrosine kinase signaling pathway / activation of cysteine-type endopeptidase activity involved in apoptotic process /  分裂促進因子活性化タンパク質キナーゼ / retina development in camera-type eye /
分裂促進因子活性化タンパク質キナーゼ / retina development in camera-type eye /  signaling receptor activity /
signaling receptor activity /  nervous system development / RAF/MAP kinase cascade /
nervous system development / RAF/MAP kinase cascade /  protein tyrosine kinase activity / positive regulation of MAPK cascade / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /
protein tyrosine kinase activity / positive regulation of MAPK cascade / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  receptor complex /
receptor complex /  エンドソーム / endosome membrane / positive regulation of cell migration / response to xenobiotic stimulus /
エンドソーム / endosome membrane / positive regulation of cell migration / response to xenobiotic stimulus /  神経繊維 / external side of plasma membrane /
神経繊維 / external side of plasma membrane /  signaling receptor binding /
signaling receptor binding /  protein phosphorylation /
protein phosphorylation /  樹状突起 / neuronal cell body /
樹状突起 / neuronal cell body /  calcium ion binding / positive regulation of gene expression / positive regulation of DNA-templated transcription /
calcium ion binding / positive regulation of gene expression / positive regulation of DNA-templated transcription /  シグナル伝達 / extracellular region /
シグナル伝達 / extracellular region /  ATP binding /
ATP binding /  細胞膜
細胞膜
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 単粒子再構成法 /
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.3 Å
クライオ電子顕微鏡法 / 解像度: 6.3 Å  データ登録者
データ登録者 スウェーデン, 2件
スウェーデン, 2件  引用
引用 ジャーナル: Sci Adv / 年: 2019
ジャーナル: Sci Adv / 年: 2019
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_0026.map.gz
emd_0026.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-0026-v30.xml
emd-0026-v30.xml emd-0026.xml
emd-0026.xml EMDBヘッダ
EMDBヘッダ emd_0026_fsc.xml
emd_0026_fsc.xml FSCデータファイル
FSCデータファイル emd_0026.png
emd_0026.png emd_0026_msk_1.map
emd_0026_msk_1.map マスクマップ
マスクマップ emd_0026_additional.map.gz
emd_0026_additional.map.gz emd_0026_half_map_1.map.gz
emd_0026_half_map_1.map.gz emd_0026_half_map_2.map.gz
emd_0026_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-0026
http://ftp.pdbj.org/pub/emdb/structures/EMD-0026 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0026
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0026 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_0026.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_0026.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_0026_msk_1.map
emd_0026_msk_1.map 試料の構成要素
試料の構成要素
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Cricetulus griseus (モンゴルキヌゲネズミ)
Cricetulus griseus (モンゴルキヌゲネズミ)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Cricetulus griseus (モンゴルキヌゲネズミ)
Cricetulus griseus (モンゴルキヌゲネズミ)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Cricetulus griseus (モンゴルキヌゲネズミ)
Cricetulus griseus (モンゴルキヌゲネズミ) 受容体型チロシンキナーゼ
受容体型チロシンキナーゼ
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Cricetulus griseus (モンゴルキヌゲネズミ)
Cricetulus griseus (モンゴルキヌゲネズミ) クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy
Bright-field microscopy
 画像解析
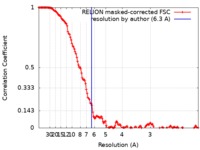
画像解析 ) / 解像度のタイプ: BY AUTHOR / 解像度: 6.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 186903
) / 解像度のタイプ: BY AUTHOR / 解像度: 6.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 186903 

 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X