+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4782 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the anti-feeding prophage (AFP) baseplate, 6-fold symmetrised | |||||||||
 マップデータ マップデータ | Cryo-EM structure of the anti-feeding prophage (AFP) baseplate, 6-fold symmetrised | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Serratia entomophila (バクテリア) Serratia entomophila (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Desfosses A | |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2019 ジャーナル: Nat Microbiol / 年: 2019タイトル: Atomic structures of an entire contractile injection system in both the extended and contracted states. 著者: Ambroise Desfosses / Hariprasad Venugopal / Tapan Joshi / Jan Felix / Matthew Jessop / Hyengseop Jeong / Jaekyung Hyun / J Bernard Heymann / Mark R H Hurst / Irina Gutsche / Alok K Mitra /       要旨: Contractile injection systems are sophisticated multiprotein nanomachines that puncture target cell membranes. Although the number of atomic-resolution insights into contractile bacteriophage tails, ...Contractile injection systems are sophisticated multiprotein nanomachines that puncture target cell membranes. Although the number of atomic-resolution insights into contractile bacteriophage tails, bacterial type six secretion systems and R-pyocins is rapidly increasing, structural information on the contraction of bacterial phage-like protein-translocation structures directed towards eukaryotic hosts is scarce. Here, we characterize the antifeeding prophage AFP from Serratia entomophila by cryo-electron microscopy. We present the high-resolution structure of the entire AFP particle in the extended state, trace 11 protein chains de novo from the apical cap to the needle tip, describe localization variants and perform specific structural comparisons with related systems. We analyse inter-subunit interactions and highlight their universal conservation within contractile injection systems while revealing the specificities of AFP. Furthermore, we provide the structure of the AFP sheath-baseplate complex in a contracted state. This study reveals atomic details of interaction networks that accompany and define the contraction mechanism of toxin-delivery tailocins, offering a comprehensive framework for understanding their mode of action and for their possible adaptation as biocontrol agents. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4782.map.gz emd_4782.map.gz | 20.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4782-v30.xml emd-4782-v30.xml emd-4782.xml emd-4782.xml | 24.3 KB 24.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4782_fsc.xml emd_4782_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4782.png emd_4782.png | 168.8 KB | ||
| その他 |  emd_4782_half_map_1.map.gz emd_4782_half_map_1.map.gz emd_4782_half_map_2.map.gz emd_4782_half_map_2.map.gz | 138.4 MB 138.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4782 http://ftp.pdbj.org/pub/emdb/structures/EMD-4782 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4782 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4782 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6raoMC  4783C  4784C  4800C  4801C  4802C  4803C  4859C  4871C  4876C  6rapC  6rbkC  6rbnC  6rc8C  6rglC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4782.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4782.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of the anti-feeding prophage (AFP) baseplate, 6-fold symmetrised | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: None
| ファイル | emd_4782_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
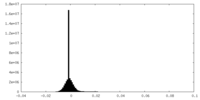
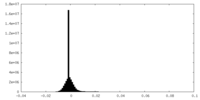
| 密度ヒストグラム |
-ハーフマップ: None
| ファイル | emd_4782_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
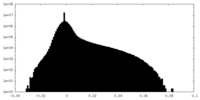
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Cryo-EM structure of the anti-feeding prophage (AFP) baseplate, 6...
+超分子 #1: Cryo-EM structure of the anti-feeding prophage (AFP) baseplate, 6...
+分子 #1: Afp1
+分子 #2: Afp2
+分子 #3: Afp3
+分子 #4: Afp5
+分子 #5: Afp9
+分子 #6: Afp4
+分子 #7: Afp7
+分子 #8: Afp11
+分子 #9: Afp12
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X