機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / /  データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード RNA-dependent RNA polymerase (RNA依存性RNAポリメラーゼ) / viral replication-transcription complex /
RNA-dependent RNA polymerase (RNA依存性RNAポリメラーゼ) / viral replication-transcription complex /  transcription (転写 (生物学)) /
transcription (転写 (生物学)) /  viral proteins (ウイルスタンパク質) / TRANSFERASE-HYDROLASE-RNA complex
viral proteins (ウイルスタンパク質) / TRANSFERASE-HYDROLASE-RNA complex 機能・相同性情報
機能・相同性情報 mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity /
mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity /  付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs ...protein guanylyltransferase activity / RNA endonuclease activity, producing 3'-phosphomonoesters /
付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs ...protein guanylyltransferase activity / RNA endonuclease activity, producing 3'-phosphomonoesters /  mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity /
mRNA guanylyltransferase activity / 5'-3' RNA helicase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity /  付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / Translation of Replicase and Assembly of the Replication Transcription Complex /
付加脱離酵素(リアーゼ); P-Oリアーゼ類; - / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / Translation of Replicase and Assembly of the Replication Transcription Complex /  snRNP Assembly / TRAF3-dependent IRF activation pathway / Replication of the SARS-CoV-2 genome /
snRNP Assembly / TRAF3-dependent IRF activation pathway / Replication of the SARS-CoV-2 genome /  加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ /
加水分解酵素; エステル加水分解酵素; 5'-リン酸モノエステル産生エキソリボヌクレアーゼ /  : / double membrane vesicle viral factory outer membrane /
: / double membrane vesicle viral factory outer membrane /  3C様プロテアーゼ / host cell endosome / 3'-5'-RNA exonuclease activity / host cell endoplasmic reticulum-Golgi intermediate compartment / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated degradation of host mRNA /
3C様プロテアーゼ / host cell endosome / 3'-5'-RNA exonuclease activity / host cell endoplasmic reticulum-Golgi intermediate compartment / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated degradation of host mRNA /  mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / SARS-CoV-2 modulates host translation machinery /
mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / SARS-CoV-2 modulates host translation machinery /  omega peptidase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / host cell Golgi apparatus / symbiont-mediated perturbation of host ubiquitin-like protein modification /
omega peptidase activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / mRNA (guanine-N7)-methyltransferase / methyltransferase cap1 / host cell Golgi apparatus / symbiont-mediated perturbation of host ubiquitin-like protein modification /  mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity /
mRNA (nucleoside-2'-O-)-methyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity /  ヘリカーゼ /
ヘリカーゼ /  ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity /
ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /  single-stranded RNA binding / host cell endoplasmic reticulum membrane / host cell perinuclear region of cytoplasm /
single-stranded RNA binding / host cell endoplasmic reticulum membrane / host cell perinuclear region of cytoplasm /  viral protein processing /
viral protein processing /  lyase activity /
lyase activity /  ヘリカーゼ / induction by virus of host autophagy /
ヘリカーゼ / induction by virus of host autophagy /  RNA依存性RNAポリメラーゼ / copper ion binding / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /
RNA依存性RNAポリメラーゼ / copper ion binding / symbiont-mediated suppression of host gene expression / viral RNA genome replication / cysteine-type endopeptidase activity /  RNA-dependent RNA polymerase activity / DNA-templated transcription /
RNA-dependent RNA polymerase activity / DNA-templated transcription /  lipid binding / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / SARS-CoV-2 activates/modulates innate and adaptive immune responses /
lipid binding / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / SARS-CoV-2 activates/modulates innate and adaptive immune responses /  ATP hydrolysis activity /
ATP hydrolysis activity /  タンパク質分解 /
タンパク質分解 /  RNA binding / zinc ion binding /
RNA binding / zinc ion binding /  ATP binding /
ATP binding /  生体膜
生体膜
 Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2)
Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) 単粒子再構成法 /
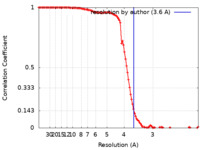
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å
クライオ電子顕微鏡法 / 解像度: 3.6 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021
ジャーナル: Proc Natl Acad Sci U S A / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_23008.map.gz
emd_23008.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-23008-v30.xml
emd-23008-v30.xml emd-23008.xml
emd-23008.xml EMDBヘッダ
EMDBヘッダ emd_23008_fsc.xml
emd_23008_fsc.xml FSCデータファイル
FSCデータファイル emd_23008.png
emd_23008.png emd-23008.cif.gz
emd-23008.cif.gz emd_23008_additional_1.map.gz
emd_23008_additional_1.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-23008
http://ftp.pdbj.org/pub/emdb/structures/EMD-23008 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23008
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23008 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_23008.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_23008.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy
Bright-field microscopy
 ムービー
ムービー コントローラー
コントローラー


























 Z
Z Y
Y X
X