+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20790 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human CALHM2 gap junction | |||||||||
 マップデータ マップデータ | Unsharpened final map refined with a soft solvent mask. Improved resolution for the well-defined parts of the protein, yet incomplete densities for the highly flexible regions such as S1. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Calcium homeostasis modulator family / Calcium homeostasis modulator / monoatomic cation channel activity / positive regulation of apoptotic process /  生体膜 / Calcium homeostasis modulator protein 2 生体膜 / Calcium homeostasis modulator protein 2 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Lu W / Du J / Choi W | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: The structures and gating mechanism of human calcium homeostasis modulator 2. 著者: Wooyoung Choi / Nicolina Clemente / Weinan Sun / Juan Du / Wei Lü /  要旨: Calcium homeostasis modulators (CALHMs) are voltage-gated, Ca-inhibited nonselective ion channels that act as major ATP release channels, and have important roles in gustatory signalling and neuronal ...Calcium homeostasis modulators (CALHMs) are voltage-gated, Ca-inhibited nonselective ion channels that act as major ATP release channels, and have important roles in gustatory signalling and neuronal toxicity. Dysfunction of CALHMs has previously been linked to neurological disorders. Here we present cryo-electron microscopy structures of the human CALHM2 channel in the Ca-free active or open state and in the ruthenium red (RUR)-bound inhibited state, at resolutions up to 2.7 Å. Our work shows that purified CALHM2 channels form both gap junctions and undecameric hemichannels. The protomer shows a mirrored arrangement of the transmembrane domains (helices S1-S4) relative to other channels with a similar topology, such as connexins, innexins and volume-regulated anion channels. Upon binding to RUR, we observed a contracted pore with notable conformational changes of the pore-lining helix S1, which swings nearly 60° towards the pore axis from a vertical to a lifted position. We propose a two-section gating mechanism in which the S1 helix coarsely adjusts, and the N-terminal helix fine-tunes, the pore size. We identified a RUR-binding site near helix S1 that may stabilize this helix in the lifted conformation, giving rise to channel inhibition. Our work elaborates on the principles of CALHM2 channel architecture and symmetry, and the mechanism that underlies channel inhibition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20790.map.gz emd_20790.map.gz | 115.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20790-v30.xml emd-20790-v30.xml emd-20790.xml emd-20790.xml | 14.2 KB 14.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20790.png emd_20790.png | 261.6 KB | ||
| その他 |  emd_20790_additional.map.gz emd_20790_additional.map.gz | 117.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20790 http://ftp.pdbj.org/pub/emdb/structures/EMD-20790 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20790 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20790 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20790.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20790.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened final map refined with a soft solvent mask. Improved resolution for the well-defined parts of the protein, yet incomplete densities for the highly flexible regions such as S1. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.026 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map refined without a soft solvent mask....
| ファイル | emd_20790_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map refined without a soft solvent mask. Better visualization of the highly flexible regions such as S1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
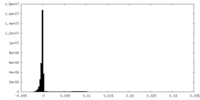
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human CALHM2
| 全体 | 名称: human CALHM2 |
|---|---|
| 要素 |
|
-超分子 #1: human CALHM2
| 超分子 | 名称: human CALHM2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Calcium homeostasis modulator protein 2
| 分子 | 名称: Calcium homeostasis modulator protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 22 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.198676 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAALIAENFR FLSLFFKSKD VMIFNGLVAL GTVGSQELFS VVAFHCPCSP ARNYLYGLAA IGVPALVLFI IGIILNNHTW NLVAECQHR RTKNCSAAPT FLLLSSILGR AAVAPVTWSV ISLLRGEAYV CALSEFVDPS SLTAREEHFP SAHATEILAR F PCKENPDN ...文字列: MAALIAENFR FLSLFFKSKD VMIFNGLVAL GTVGSQELFS VVAFHCPCSP ARNYLYGLAA IGVPALVLFI IGIILNNHTW NLVAECQHR RTKNCSAAPT FLLLSSILGR AAVAPVTWSV ISLLRGEAYV CALSEFVDPS SLTAREEHFP SAHATEILAR F PCKENPDN LSDFREEVSR RLRYESQLFG WLLIGVVAIL VFLTKCLKHY CSPLSYRQEA YWAQYRANED QLFQRTAEVH SR VLAANNV RRFFGFVALN KDDEELIANF PVEGTQPRPQ WNAITGVYLY RENQGLPLYS RLHKWAQGLA GNGAAPDNVE MAL LPSFES RLVPR |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 8.0 sec. / 平均電子線量: 54.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 576174 |
|---|---|
| CTF補正 | ソフトウェア - 名称: Gctf |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.0) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 3.0) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 300) |
| 最終 再構成 | 想定した対称性 - 点群: C11 (11回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0) / 使用した粒子像数: 71934 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0) / 使用した粒子像数: 71934 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6uix: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X