+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2289 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Second 3D model of wild type MjHSP16.5 at room temperature by CryoEM. | |||||||||
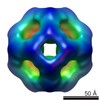
 マップデータ マップデータ | Second model for reconstruction of wild type MjHSP16.5 at room temperature | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Small heat shock protein / HSP16.5 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to stress / protein folding chaperone / response to salt stress / response to hydrogen peroxide / : / unfolded protein binding /  フォールディング / protein complex oligomerization / response to heat / protein stabilization ...response to stress / protein folding chaperone / response to salt stress / response to hydrogen peroxide / : / unfolded protein binding / フォールディング / protein complex oligomerization / response to heat / protein stabilization ...response to stress / protein folding chaperone / response to salt stress / response to hydrogen peroxide / : / unfolded protein binding /  フォールディング / protein complex oligomerization / response to heat / protein stabilization / protein-containing complex / identical protein binding / フォールディング / protein complex oligomerization / response to heat / protein stabilization / protein-containing complex / identical protein binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |    Methanocaldococcus jannaschii (メタン生成菌) Methanocaldococcus jannaschii (メタン生成菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 12.0 Å クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Roy A Q / Yan Z / Andrew L / Ian W / Ehmke P / Fei S | |||||||||
 引用 引用 |  ジャーナル: Philos Trans R Soc Lond B Biol Sci / 年: 2013 ジャーナル: Philos Trans R Soc Lond B Biol Sci / 年: 2013タイトル: Changes in the quaternary structure and function of MjHSP16.5 attributable to deletion of the IXI motif and introduction of the substitution, R107G, in the α-crystallin domain. 著者: Roy A Quinlan / Yan Zhang / Andrew Lansbury / Ian Williamson / Ehmke Pohl / Fei Sun /  要旨: The archael small heat-shock protein (sHSP), MjHSP16.5, forms a 24-subunit oligomer with octahedral symmetry. Here, we demonstrate that the IXI motif present in the C-terminal domain is necessary for ...The archael small heat-shock protein (sHSP), MjHSP16.5, forms a 24-subunit oligomer with octahedral symmetry. Here, we demonstrate that the IXI motif present in the C-terminal domain is necessary for the oligomerization of MjHSP16.5. Removal increased the in vitro chaperone activity with citrate synthase as the client protein. Less predictable were the effects of the R107G substitution in MjHSP16.5 because of the differences in the oligomerization of metazoan and non-metazoan sHSPs. We present the crystal structure for MjHSP16.5 R107G and compare this with an improved (2.5 Å) crystal structure for wild-type (WT) MjHSP16.5. Although no significant structural differences were found in the crystal, using cryo-electron microscopy, we identified two 24mer species with octahedral symmetry for the WT MjHSP16.5 both at room temperature and at 60°C, all showing two major species with the same diameter of 12.4 nm. Similarly, at room temperature, there are also two kinds of 12.4 nm oligomers for R107G MjHSP16.5, but in the 60°C sample, a larger 24mer species with a diameter of 13.6 nm was observed with significant changes in the fourfold symmetry axis and dimer-dimer interface. This highly conserved arginine, therefore, contributes to the quaternary organization of non-metazoan sHSP oligomers. Potentially, the R107G substitution has functional consequences as R107G MjHSP16.5 was far superior to the WT protein in protecting βL-crystallin against heat-induced aggregation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2289.map.gz emd_2289.map.gz | 56.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2289-v30.xml emd-2289-v30.xml emd-2289.xml emd-2289.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  2289_EMDB-2289.tif 2289_EMDB-2289.tif | 118.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2289 http://ftp.pdbj.org/pub/emdb/structures/EMD-2289 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2289 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2289 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2289.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2289.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second model for reconstruction of wild type MjHSP16.5 at room temperature | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.933 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : wild type small heat shock protein (sHSP) HSP16.5 from Methanocal...
| 全体 | 名称: wild type small heat shock protein (sHSP) HSP16.5 from Methanocaldococcus jannaschii (MjHSP16.5) |
|---|---|
| 要素 |
|
-超分子 #1000: wild type small heat shock protein (sHSP) HSP16.5 from Methanocal...
| 超分子 | 名称: wild type small heat shock protein (sHSP) HSP16.5 from Methanocaldococcus jannaschii (MjHSP16.5) タイプ: sample / ID: 1000 / 集合状態: 24mer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 396 KDa / 理論値: 396 KDa |
-分子 #1: MjHSP16.5
| 分子 | 名称: MjHSP16.5 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: small heat shock protein (sHSP) from HSP16.5 / コピー数: 24 / 集合状態: 24mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:    Methanocaldococcus jannaschii (メタン生成菌) Methanocaldococcus jannaschii (メタン生成菌)細胞中の位置: cytoplasmic |
| 分子量 | 実験値: 16.5 KDa / 理論値: 16.5 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換プラスミド: pET23b Escherichia coli (大腸菌) / 組換プラスミド: pET23b |
| 配列 | UniProtKB: Small heat shock protein HSP16.5 / GO:  細胞質, response to stress 細胞質, response to stressInterPro: Alpha crystallin/Hsp20 domain, HSP20-like chaperone |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 10mM HEPES, 100mM NaCl. |
| グリッド | 詳細: GIG holey grids (LifeTrust, China) were treated with a glow discharge machine (Master Plasmer) |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 93 K / 装置: FEI VITROBOT MARK IV 手法: The samples were blotted for 2 s with blot force 2 at 100% humidity. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 96000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダー: liquid nitrogen cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 温度 | 平均: 85 K |
| 日付 | 2012年9月28日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 1160 / 平均電子線量: 20 e/Å2 詳細: Electron micrograph exposures were made with the automatic collection package Leginon. ビット/ピクセル: 32 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each image |
|---|---|
| 最終 2次元分類 | クラス数: 193 |
| 最終 再構成 | 想定した対称性 - 点群: O (正8面体型対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN1 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 12.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN1詳細: The final reconstructed density map was further sharpened by application of an amplitude correction algorithm in the program BFACTOR. 使用した粒子像数: 4356 |
| 詳細 | The particles were selected using an automatic selection program Gautomatch developed in Fei Sun lab (to be published). Octahedron symmetry were imposed during 3D reconstructing. |
 ムービー
ムービー コントローラー
コントローラー