+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20236 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of S. cerevisiae protein O-mannosyltransferase Pmt1-Pmt2 complex bound to the sugar donor and a peptide acceptor | |||||||||
 マップデータ マップデータ | eukaryotic protein O-mannosyltransferase Pmt1/Pmt2 in complex with sugar donor and acceptor 真核生物 真核生物 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt3p dimer complex / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt5p-Pmt2p dimer complex /  dolichyl-phosphate-mannose-protein mannosyltransferase / dolichyl-phosphate-mannose-protein mannosyltransferase /  dolichyl-phosphate-mannose-protein mannosyltransferase activity / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt2p dimer complex / regulation of endoplasmic reticulum unfolded protein response / fungal-type cell wall biogenesis / protein O-linked mannosylation / protein exit from endoplasmic reticulum / protein O-linked glycosylation ...dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt3p dimer complex / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt5p-Pmt2p dimer complex / dolichyl-phosphate-mannose-protein mannosyltransferase activity / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt2p dimer complex / regulation of endoplasmic reticulum unfolded protein response / fungal-type cell wall biogenesis / protein O-linked mannosylation / protein exit from endoplasmic reticulum / protein O-linked glycosylation ...dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt3p dimer complex / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt5p-Pmt2p dimer complex /  dolichyl-phosphate-mannose-protein mannosyltransferase / dolichyl-phosphate-mannose-protein mannosyltransferase /  dolichyl-phosphate-mannose-protein mannosyltransferase activity / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt2p dimer complex / regulation of endoplasmic reticulum unfolded protein response / fungal-type cell wall biogenesis / protein O-linked mannosylation / protein exit from endoplasmic reticulum / protein O-linked glycosylation / : / endoplasmic reticulum membrane / dolichyl-phosphate-mannose-protein mannosyltransferase activity / dolichyl-phosphate-mannose-protein mannosyltransferase Pmt1p-Pmt2p dimer complex / regulation of endoplasmic reticulum unfolded protein response / fungal-type cell wall biogenesis / protein O-linked mannosylation / protein exit from endoplasmic reticulum / protein O-linked glycosylation / : / endoplasmic reticulum membrane /  小胞体 小胞体類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae W303 (パン酵母) Saccharomyces cerevisiae W303 (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Bai L / Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Structure of the eukaryotic protein O-mannosyltransferase Pmt1-Pmt2 complex. 著者: Lin Bai / Amanda Kovach / Qinglong You / Alanna Kenny / Huilin Li /  要旨: In eukaryotes, a nascent peptide entering the endoplasmic reticulum (ER) is scanned by two Sec61 translocon-associated large membrane machines for protein N-glycosylation and protein O-mannosylation, ...In eukaryotes, a nascent peptide entering the endoplasmic reticulum (ER) is scanned by two Sec61 translocon-associated large membrane machines for protein N-glycosylation and protein O-mannosylation, respectively. While the structure of the eight-protein oligosaccharyltransferase complex has been determined recently, the structures of mannosyltransferases of the PMT family, which are an integral part of ER protein homeostasis, are still unknown. Here we report cryo-EM structures of the Saccharomyces cerevisiae Pmt1-Pmt2 complex bound to a donor and an acceptor peptide at 3.2-Å resolution, showing that each subunit contains 11 transmembrane helices and a lumenal β-trefoil fold termed the MIR domain. The structures reveal the substrate recognition model and confirm an inverting mannosyl-transferring reaction mechanism by the enzyme complex. Furthermore, we found that the transmembrane domains of Pmt1 and Pmt2 share a structural fold with the catalytic subunits of oligosaccharyltransferases, confirming a previously proposed evolutionary relationship between protein O-mannosylation and protein N-glycosylation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20236.map.gz emd_20236.map.gz | 6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20236-v30.xml emd-20236-v30.xml emd-20236.xml emd-20236.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20236.png emd_20236.png | 179.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20236 http://ftp.pdbj.org/pub/emdb/structures/EMD-20236 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20236 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20236 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20236.map.gz / 形式: CCP4 / 大きさ: 37.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20236.map.gz / 形式: CCP4 / 大きさ: 37.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | eukaryotic protein O-mannosyltransferase Pmt1/Pmt2 in complex with sugar donor and acceptor | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.029 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Protein O-mannosyl transferase complex
| 全体 | 名称: Protein O-mannosyl transferase complex |
|---|---|
| 要素 |
|
-超分子 #1: Protein O-mannosyl transferase complex
| 超分子 | 名称: Protein O-mannosyl transferase complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) Saccharomyces cerevisiae W303 (パン酵母) |
| 組換発現 | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) Saccharomyces cerevisiae W303 (パン酵母) |
-分子 #1: Dolichyl-phosphate-mannose--protein mannosyltransferase 1
| 分子 | 名称: Dolichyl-phosphate-mannose--protein mannosyltransferase 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号:  dolichyl-phosphate-mannose-protein mannosyltransferase dolichyl-phosphate-mannose-protein mannosyltransferase |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) / 株: W303 Saccharomyces cerevisiae W303 (パン酵母) / 株: W303 |
| 分子量 | 理論値: 92.771125 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) Saccharomyces cerevisiae W303 (パン酵母) |
| 配列 | 文字列: MSEEKTYKRV EQDDPVPELD IKQGPVRPFI VTDPSAELAS LRTMVTLKEK LLVACLAVFT AVIRLHGLAW PDSVVFDEVH FGGFASQYI RGTYFMDVHP PLAKMLYAGV ASLGGFQGDF DFENIGDSFP STTPYVLMRF FSASLGALTV ILMYMTLRYS G VRMWVALM ...文字列: MSEEKTYKRV EQDDPVPELD IKQGPVRPFI VTDPSAELAS LRTMVTLKEK LLVACLAVFT AVIRLHGLAW PDSVVFDEVH FGGFASQYI RGTYFMDVHP PLAKMLYAGV ASLGGFQGDF DFENIGDSFP STTPYVLMRF FSASLGALTV ILMYMTLRYS G VRMWVALM SAICFAVENS YVTISRYILL DAPLMFFIAA AVYSFKKYEM YPANSLNAYK SLLATGIALG MASSSKWVGL FT VTWVGLL CIWRLWFMIG DLTKSSKSIF KVAFAKLAFL LGVPFALYLV FFYIHFQSLT LDGDGASFFS PEFRSTLKNN KIP QNVVAD VGIGSIISLR HLSTMGGYLH SHSHNYPAGS EQQQSTLYPH MDANNDWLLE LYNAPGESLT TFQNLTDGTK VRLF HTVTR CRLHSHDHKP PVSESSDWQK EVSCYGYSGF DGDANDDWVV EIDKKNSAPG VAQERVIALD TKFRLRHAMT GCYLF SHEV KLPAWGFEQQ EVTCASSGRH DLTLWYVENN SNPLLPEDTK RISYKPASFI SKFIESHKKM WHINKNLVEP HVYESQ PTS WPFLLRGISY WGENNRNVYL LGNAIVWWAV TAFIGIFGLI VITELFSWQL GKPILKDSKV VNFHVQVIHY LLGFAVH YA PSFLMQRQMF LHHYLPAYYF GILALGHALD IIVSYVFRSK RQMGYAVVIT FLAASVYFFK SFSPIIYGTP WTQELCQK S QWLSGWDYNC NTYFSSLEEY KNQTLTKRES QPAATSTVEE ITIEGDGPSY EDLMNEDGKK IFKDTEGNEL DPEVVKKML EEEGANILKV EKRAVLE |
-分子 #2: Dolichyl-phosphate-mannose--protein mannosyltransferase 2
| 分子 | 名称: Dolichyl-phosphate-mannose--protein mannosyltransferase 2 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号:  dolichyl-phosphate-mannose-protein mannosyltransferase dolichyl-phosphate-mannose-protein mannosyltransferase |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) / 株: W303 Saccharomyces cerevisiae W303 (パン酵母) / 株: W303 |
| 分子量 | 理論値: 86.957422 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) Saccharomyces cerevisiae W303 (パン酵母) |
| 配列 | 文字列: MSSSSSTGYS KNNAAHIKQE NTLRQRESSS ISVSEELSSA DERDAEDFSK EKPAAQSSLL RLESVVMPVI FTALALFTRM YKIGINNHV VWDEAHFGKF GSYYLRHEFY HDVHPPLGKM LVGLSGYLAG YNGSWDFPSG EIYPDYLDYV KMRLFNASFS A LCVPLAYF ...文字列: MSSSSSTGYS KNNAAHIKQE NTLRQRESSS ISVSEELSSA DERDAEDFSK EKPAAQSSLL RLESVVMPVI FTALALFTRM YKIGINNHV VWDEAHFGKF GSYYLRHEFY HDVHPPLGKM LVGLSGYLAG YNGSWDFPSG EIYPDYLDYV KMRLFNASFS A LCVPLAYF TAKAIGFSLP TVWLMTVLVL FENSYSTLGR FILLDSMLLF FTVASFFSFV MFHNQRSKPF SRKWWKWLLI TG ISLGCTI SVKMVGLFII TMVGIYTVID LWTFLADKSM SWKTYINHWL ARIFGLIIVP FCIFLLCFKI HFDLLSHSGT GDA NMPSLF QARLVGSDVG QGPRDIALGS SVVSIKNQAL GGSLLHSHIQ TYPDGSNQQQ VTCYGYKDAN NEWFFNRERG LPSW SENET DIEYLKPGTS YRLVHKSTGR NLHTHPVAAP VSKTQWEVSG YGDNVVGDNK DNWVIEIMDQ RGDEDPEKLH TLTTS FRIK NLEMGCYLAQ TGNSLPEWGF RQQEVVCMKN PFKRDKRTWW NIETHENERL PPRPEDFQYP KTNFLKDFIH LNLAMM ATN NALVPDPDKF DYLASSAWQW PTLNVGLRLC GWGDDNPKYF LLGTPASTWA SSVAVLAFMA TVVILLIRWQ RQYVDLR NP SNWNVFLMGG FYPLLAWGLH YMPFVIMSRV TYVHHYLPAL YFALIILAYC FDAGLQKWSR SKCGRIMRFV LYAGFMAL V IGCFWYFSPI SFGMEGPSSN FRYLNWFSTW DIADKQEA |
-分子 #3: acceptor peptide
| 分子 | 名称: acceptor peptide / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae W303 (パン酵母) Saccharomyces cerevisiae W303 (パン酵母) |
| 分子量 | 理論値: 478.538 Da |
| 配列 | 文字列: PYTV |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #5: 1-PALMITOYL-2-LINOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1-PALMITOYL-2-LINOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: CPL |
|---|---|
| 分子量 | 理論値: 758.06 Da |
| Chemical component information |  ChemComp-CPL: |
-分子 #6: (3R)-3,31-dimethyl-7,11,15,19,23,27-hexamethylidenedotriacont-31-...
| 分子 | 名称: (3R)-3,31-dimethyl-7,11,15,19,23,27-hexamethylidenedotriacont-31-en-1-yl dihydrogen phosphate タイプ: ligand / ID: 6 / コピー数: 1 / 式: NNM |
|---|---|
| 分子量 | 理論値: 644.947 Da |
| Chemical component information | 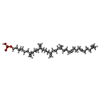 ChemComp-NNM: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 2.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 953292 |
|---|---|
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4.1) |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 3次元分類 | ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION (ver. 2.1) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア: (名称:  Coot, Coot,  UCSF Chimera) / 使用した粒子像数: 521848 UCSF Chimera) / 使用した粒子像数: 521848 |
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6p25: |
 ムービー
ムービー コントローラー
コントローラー















