+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9rsj | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of MATE transporter NorM-VC in complex with doxorubicin | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | TRANSPORT PROTEIN / NabFab / substrate / multidrug transporter | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報antiporter activity / xenobiotic transmembrane transporter activity / monoatomic ion transport / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  synthetic construct (人工物)  | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Romane, K. / Hsieh, P.Y. / Kowal, J. / Locher, K.P. / van Veen, H.W. | |||||||||
| 資金援助 |  スイス, スイス,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2025 ジャーナル: J Mol Biol / 年: 2025タイトル: Doxorubicin recognition and transport by the MATE multidrug transporter NorM from Vibrio cholerae. 著者: Pei-Yu Hsieh / Ksenija Romane / Julia Kowal / Kaspar P Locher / Hendrik W van Veen /   要旨: Multidrug and toxic compound extrusion (MATE) transport proteins contribute to multidrug resistance in human pathogens by extruding various cytotoxic compounds from the cellular interior. Despite ...Multidrug and toxic compound extrusion (MATE) transport proteins contribute to multidrug resistance in human pathogens by extruding various cytotoxic compounds from the cellular interior. Despite their importance across all domains of life, the specificities and mechanisms of substrate transport of these proteins remain poorly understood due to limited structural and functional information. Here, we determined the cryo-electron microscopy structure of NorM from Vibrio cholerae (NorM-VC) in complex with the anthracycline antibiotic doxorubicin, using the NabFab approach. The structure reveals that the doxorubicin-binding pocket is located halfway through the membrane, within the C-lobe of the protein. Functional studies targeting the doxorubicin-interacting residues validated the binding pocket and enabled detailed analysis of the doxorubicin transport reaction. Our findings indicate doxorubicin binding within a multisite binding chamber engaged in a general transport mechanism for a variety of substrates. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9rsj.cif.gz 9rsj.cif.gz | 230.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9rsj.ent.gz pdb9rsj.ent.gz | 179.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9rsj.json.gz 9rsj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  9rsj_validation.pdf.gz 9rsj_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  9rsj_full_validation.pdf.gz 9rsj_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  9rsj_validation.xml.gz 9rsj_validation.xml.gz | 52.7 KB | 表示 | |
| CIF形式データ |  9rsj_validation.cif.gz 9rsj_validation.cif.gz | 78.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rs/9rsj https://data.pdbj.org/pub/pdb/validation_reports/rs/9rsj ftp://data.pdbj.org/pub/pdb/validation_reports/rs/9rsj ftp://data.pdbj.org/pub/pdb/validation_reports/rs/9rsj | HTTPS FTP |
-関連構造データ
| 関連構造データ |  54220MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-抗体 , 4種, 4分子 HLNK
| #2: 抗体 | 分子量: 25684.463 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現) synthetic construct (人工物) / 発現宿主:  |
|---|---|
| #3: 抗体 | 分子量: 23258.783 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現) synthetic construct (人工物) / 発現宿主:  |
| #4: 抗体 | 分子量: 14356.979 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: Nanobody / 由来: (組換発現) synthetic construct (人工物) / 発現宿主:  |
| #5: 抗体 | 分子量: 13390.644 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: Nanobody / 由来: (組換発現)   |
-タンパク質 / 非ポリマー , 2種, 2分子 A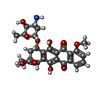

| #1: タンパク質 | 分子量: 49892.328 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   |
|---|---|
| #6: 化合物 | ChemComp-DM2 / |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: MATE transporter NorM from Vibrio cholerae in complex with NabFab, Nb17_4, anti-Fab nanobody and doxorubicin タイプ: COMPLEX / Entity ID: #1-#5 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.128 MDa / 実験値: NO |
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE-PROPANE / 湿度: 100 % |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2400 nm / 最小 デフォーカス(公称値): 600 nm |
| 試料ホルダ | 凍結剤: NITROGEN |
| 撮影 | 電子線照射量: 65 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 327809 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 精密化 | 最高解像度: 3.1 Å / 交差検証法: NONE 立体化学のターゲット値: REAL-SPACE (WEIGHTED MAP SUM AT ATOM CENTERS) | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj


