+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9o0u | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of CRAF/MEK1 complex with PLX4720 and CH5126766 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSFERASE / CRAF-MEK1 complex / MAPK pathway | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報death-inducing signaling complex assembly / epithelial cell proliferation involved in lung morphogenesis / positive regulation of endodermal cell differentiation / negative regulation of homotypic cell-cell adhesion / negative regulation of hypoxia-induced intrinsic apoptotic signaling pathway / regulation of vascular associated smooth muscle contraction / intermediate filament cytoskeleton organization / mitogen-activated protein kinase kinase / Golgi inheritance / placenta blood vessel development ...death-inducing signaling complex assembly / epithelial cell proliferation involved in lung morphogenesis / positive regulation of endodermal cell differentiation / negative regulation of homotypic cell-cell adhesion / negative regulation of hypoxia-induced intrinsic apoptotic signaling pathway / regulation of vascular associated smooth muscle contraction / intermediate filament cytoskeleton organization / mitogen-activated protein kinase kinase / Golgi inheritance / placenta blood vessel development / MAP-kinase scaffold activity / positive regulation of muscle contraction / regulation of axon regeneration / cerebellar cortex formation / labyrinthine layer development / regulation of Rho protein signal transduction / melanosome transport / type B pancreatic cell proliferation / Signaling by MAP2K mutants / SHOC2 M1731 mutant abolishes MRAS complex function / Gain-of-function MRAS complexes activate RAF signaling / Rap1 signalling / vesicle transport along microtubule / positive regulation of axonogenesis / positive regulation of Ras protein signal transduction / insulin secretion involved in cellular response to glucose stimulus / regulation of Golgi inheritance / mitogen-activated protein kinase kinase kinase binding / central nervous system neuron differentiation / triglyceride homeostasis / trachea formation / Negative feedback regulation of MAPK pathway / regulation of early endosome to late endosome transport / IFNG signaling activates MAPKs / regulation of stress-activated MAPK cascade / Frs2-mediated activation / GP1b-IX-V activation signalling / MAPK3 (ERK1) activation / ERBB2-ERBB3 signaling pathway / neurotrophin TRK receptor signaling pathway / regulation of neurotransmitter receptor localization to postsynaptic specialization membrane / pseudopodium / face development / endodermal cell differentiation / MAP kinase kinase activity / Bergmann glial cell differentiation / positive regulation of ATP biosynthetic process / regulation of cell differentiation / thyroid gland development / Uptake and function of anthrax toxins / positive regulation of protein serine/threonine kinase activity / extrinsic apoptotic signaling pathway via death domain receptors / somatic stem cell population maintenance / protein kinase activator activity / positive regulation of peptidyl-serine phosphorylation / MAP kinase kinase kinase activity / type II interferon-mediated signaling pathway / negative regulation of protein-containing complex assembly / Schwann cell development / response to axon injury / negative regulation of extrinsic apoptotic signaling pathway via death domain receptors / keratinocyte differentiation / neuron projection morphogenesis / response to muscle stretch / ERK1 and ERK2 cascade / myelination / positive regulation of autophagy / protein serine/threonine/tyrosine kinase activity / CD209 (DC-SIGN) signaling / dendrite cytoplasm / insulin-like growth factor receptor signaling pathway / response to glucocorticoid / MAP3K8 (TPL2)-dependent MAPK1/3 activation / thymus development / adenylate cyclase activator activity / protein serine/threonine kinase activator activity / Signal transduction by L1 / cell motility / positive regulation of transcription elongation by RNA polymerase II / wound healing / RAF activation / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / small GTPase binding / Stimuli-sensing channels / neuron differentiation / chemotaxis / Negative regulation of MAPK pathway / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / Signaling by BRAF and RAF1 fusions / cellular senescence / insulin receptor signaling pathway / late endosome / MAPK cascade / heart development / response to oxidative stress / protein tyrosine kinase activity 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.91 Å 分子置換 / 解像度: 2.91 Å | ||||||
 データ登録者 データ登録者 | Jang, D.M. / Eck, M.J. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2025 ジャーナル: J.Biol.Chem. / 年: 2025タイトル: Characterization and inhibitor sensitivity of ARAF, BRAF, and CRAF kinases. 著者: Tkacik, E. / Jang, D.M. / Boxer, K. / Ha, B.H. / Eck, M.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9o0u.cif.gz 9o0u.cif.gz | 512.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9o0u.ent.gz pdb9o0u.ent.gz | 385.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9o0u.json.gz 9o0u.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  9o0u_validation.pdf.gz 9o0u_validation.pdf.gz | 2.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  9o0u_full_validation.pdf.gz 9o0u_full_validation.pdf.gz | 2.9 MB | 表示 | |
| XML形式データ |  9o0u_validation.xml.gz 9o0u_validation.xml.gz | 93.8 KB | 表示 | |
| CIF形式データ |  9o0u_validation.cif.gz 9o0u_validation.cif.gz | 120.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/o0/9o0u https://data.pdbj.org/pub/pdb/validation_reports/o0/9o0u ftp://data.pdbj.org/pub/pdb/validation_reports/o0/9o0u ftp://data.pdbj.org/pub/pdb/validation_reports/o0/9o0u | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9o0vC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| 4 | 
| ||||||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 8分子 ACEGBDFH
| #1: タンパク質 | 分子量: 32005.803 Da / 分子数: 4 / 変異: Y340D, Y341D / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: RAF1, RAF Homo sapiens (ヒト) / 遺伝子: RAF1, RAF発現宿主:  参照: UniProt: P04049, non-specific serine/threonine protein kinase #2: タンパク質 | 分子量: 43576.039 Da / 分子数: 4 / 変異: S218A, S222A / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MAP2K1, MEK1, PRKMK1 Homo sapiens (ヒト) / 遺伝子: MAP2K1, MEK1, PRKMK1発現宿主:  参照: UniProt: Q02750, mitogen-activated protein kinase kinase |
|---|
-非ポリマー , 4種, 53分子 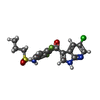






| #3: 化合物 | ChemComp-324 / #4: 化合物 | ChemComp-CHU / #5: 化合物 | ChemComp-ANP / | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.87 Å3/Da / 溶媒含有率: 57.14 % |
|---|---|
| 結晶化 | 温度: 295 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 18% PEG 3350, 0.1 M Sodium citrate pH 5.6, 4% Tacsimate pH 5 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS-II NSLS-II  / ビームライン: 17-ID-1 / 波長: 0.9202 Å / ビームライン: 17-ID-1 / 波長: 0.9202 Å |
| 検出器 | タイプ: DECTRIS EIGER X 9M / 検出器: PIXEL / 日付: 2024年11月8日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9202 Å / 相対比: 1 |
| 反射 | 解像度: 2.91→50 Å / Num. obs: 147338 / % possible obs: 99.7 % / 冗長度: 4.79 % / Biso Wilson estimate: 67.3 Å2 / CC1/2: 0.995 / Rmerge(I) obs: 0.132 / Net I/σ(I): 9.63 |
| 反射 シェル | 解像度: 2.91→3.08 Å / 冗長度: 4.69 % / Rmerge(I) obs: 1.044 / Num. unique obs: 23539 / CC1/2: 0.995 / % possible all: 98.8 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 2.91→46.1 Å / SU ML: 0.3576 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 25.7681 分子置換 / 解像度: 2.91→46.1 Å / SU ML: 0.3576 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 25.7681 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.1 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 80.51 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.91→46.1 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj






















