+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7tea | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of S. aureus GlnR-DNA complex | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / GlnR / winged-HTH / S. aureus / femC / glutamine synthetase / transcription repressor / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.35 Å 分子置換 / 解像度: 2.35 Å | ||||||
 データ登録者 データ登録者 | Schumacher, M.A. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Molecular dissection of the glutamine synthetase-GlnR nitrogen regulatory circuitry in Gram-positive bacteria. 著者: Brady A Travis / Jared V Peck / Raul Salinas / Brandon Dopkins / Nicholas Lent / Viet D Nguyen / Mario J Borgnia / Richard G Brennan / Maria A Schumacher /  要旨: How bacteria sense and respond to nitrogen levels are central questions in microbial physiology. In Gram-positive bacteria, nitrogen homeostasis is controlled by an operon encoding glutamine ...How bacteria sense and respond to nitrogen levels are central questions in microbial physiology. In Gram-positive bacteria, nitrogen homeostasis is controlled by an operon encoding glutamine synthetase (GS), a dodecameric machine that assimilates ammonium into glutamine, and the GlnR repressor. GlnR detects nitrogen excess indirectly by binding glutamine-feedback-inhibited-GS (FBI-GS), which activates its transcription-repression function. The molecular mechanisms behind this regulatory circuitry, however, are unknown. Here we describe biochemical and structural analyses of GS and FBI-GS-GlnR complexes from pathogenic and non-pathogenic Gram-positive bacteria. The structures show FBI-GS binds the GlnR C-terminal domain within its active-site cavity, juxtaposing two GlnR monomers to form a DNA-binding-competent GlnR dimer. The FBI-GS-GlnR interaction stabilizes the inactive GS conformation. Strikingly, this interaction also favors a remarkable dodecamer to tetradecamer transition in some GS, breaking the paradigm that all bacterial GS are dodecamers. These data thus unveil unique structural mechanisms of transcription and enzymatic regulation. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7tea.cif.gz 7tea.cif.gz | 238.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7tea.ent.gz pdb7tea.ent.gz | 186.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7tea.json.gz 7tea.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7tea_validation.pdf.gz 7tea_validation.pdf.gz | 1.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7tea_full_validation.pdf.gz 7tea_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  7tea_validation.xml.gz 7tea_validation.xml.gz | 16.7 KB | 表示 | |
| CIF形式データ |  7tea_validation.cif.gz 7tea_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/te/7tea https://data.pdbj.org/pub/pdb/validation_reports/te/7tea ftp://data.pdbj.org/pub/pdb/validation_reports/te/7tea ftp://data.pdbj.org/pub/pdb/validation_reports/te/7tea | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7tdpC  7tdvC 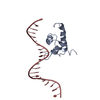 7tecC  7tenC  7tf6C  7tf7C  7tf9C  7tfaC  7tfbC  7tfcC  7tfdC  7tfeC  4r4eS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||
| 2 | 
| |||||||||
| 単位格子 |
| |||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: タンパク質 | 分子量: 10141.709 Da / 分子数: 4 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: glnR, femC, BN1321_240156, BSG37_06895, BTN44_06150, C7P97_11270, CFN13_06110, CSC87_13065, CV021_11625, DD547_01329, DQU54_07135, E3A28_02075, E3K14_06475, E4U00_04150, E5491_06810, ELP52_ ...遺伝子: glnR, femC, BN1321_240156, BSG37_06895, BTN44_06150, C7P97_11270, CFN13_06110, CSC87_13065, CV021_11625, DD547_01329, DQU54_07135, E3A28_02075, E3K14_06475, E4U00_04150, E5491_06810, ELP52_06590, FA040_05040, FAF32_003415, FPP26_06465, FVP29_03765, G6X35_05925, G6X37_15500, G6Y24_07775, GO782_03725, GO788_03065, GO793_18140, GO810_03920, GO814_05910, GO941_02620, GO942_07700, GQX37_02050, GZ128_03000, GZ145_02540, HMPREF3211_01353, NCTC10654_01351, NCTC10702_02054, NCTC5664_02143, NCTC6133_01601, RK64_06965, SA0759_02803, SA950122_02714, SAGV51_01473, SAGV69_01244, SAHC1335_02241, SAJPND4_01239, SAMEA1029536_01443, SAMEA103891420_00579, SAMEA103891454_00903, SAMEA2076468_01609, SAMEA2076503_01121, SAMEA2077334_01678, SAMEA2078260_01440, SAMEA2078588_01695, SAMEA2079501_01495, SAMEA2080344_01609, SAMEA2081063_01288, SAMEA4008569_01468, SAMEA70146418_02626, SAMEA70245418_01181 発現宿主:  #2: DNA鎖 | 分子量: 6447.185 Da / 分子数: 2 / 由来タイプ: 合成 / 詳細: synthetic GlnR operator DNA 由来: (合成)  #3: DNA鎖 | 分子量: 6438.171 Da / 分子数: 2 / 由来タイプ: 合成 由来: (合成)  #4: 化合物 | #5: 水 | ChemComp-HOH / | 研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.97 Å3/Da / 溶媒含有率: 58.61 % |
|---|---|
| 結晶化 | 温度: 275 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 50 mM sodium cacodylate pH 6.5, 13% PEG 8000, 0.15 M calcium acetate, 20% glycerol |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 5.0.2 / 波長: 1.09 Å / ビームライン: 5.0.2 / 波長: 1.09 Å | |||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2021年3月1日 | |||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||
| 放射波長 | 波長: 1.09 Å / 相対比: 1 | |||||||||||||||||||||
| 反射 | 解像度: 2.35→49.63 Å / Num. obs: 31531 / % possible obs: 98.6 % / 冗長度: 6.2 % / CC1/2: 1 / Rpim(I) all: 0.027 / Rsym value: 0.042 / Net I/σ(I): 21.4 | |||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 4R4E 解像度: 2.35→49.63 Å / SU ML: 0.47 / 交差検証法: THROUGHOUT / σ(F): 0 / 位相誤差: 38.36 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 165.69 Å2 / Biso mean: 73.7533 Å2 / Biso min: 30 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.35→49.63 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 14
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: 41.7984 Å / Origin y: 61.1108 Å / Origin z: 91.5599 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj











































