| 登録情報 | データベース: PDB / ID: 7szl
|
|---|
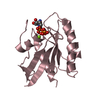
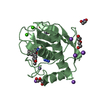
| タイトル | Crystal structure of the Toll/interleukin-1 receptor domain of human IL-1R10 (IL-1RAPL2) |
|---|
 要素 要素 | X-linked interleukin-1 receptor accessory protein-like 2 |
|---|
 キーワード キーワード | MEMBRANE PROTEIN / TIR domain / Interleukin-1 receptor / IMMUNE SYSTEM |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
interleukin-1, type II, blocking receptor activity / interleukin-1 receptor activity / Receptor-type tyrosine-protein phosphatases / NADP+ nucleosidase activity / ADP-ribosyl cyclase/cyclic ADP-ribose hydrolase / NAD+ nucleotidase, cyclic ADP-ribose generating / regulation of presynapse assembly / central nervous system development / membrane => GO:0016020 / glutamatergic synapse / plasma membrane類似検索 - 分子機能 Interleukin-1 receptor type II / IL-1Ra-like, immunoglobulin domain / Immunoglobulin domain / Interleukin-1 receptor family / TIR domain / Toll - interleukin 1 - resistance / Immunoglobulin domain / TIR domain profile. / Toll/interleukin-1 receptor homology (TIR) domain / Toll/interleukin-1 receptor homology (TIR) domain superfamily ...Interleukin-1 receptor type II / IL-1Ra-like, immunoglobulin domain / Immunoglobulin domain / Interleukin-1 receptor family / TIR domain / Toll - interleukin 1 - resistance / Immunoglobulin domain / TIR domain profile. / Toll/interleukin-1 receptor homology (TIR) domain / Toll/interleukin-1 receptor homology (TIR) domain superfamily / Immunoglobulin / Immunoglobulin domain / Immunoglobulin subtype / Immunoglobulin / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily / Immunoglobulin-like fold類似検索 - ドメイン・相同性 X-linked interleukin-1 receptor accessory protein-like 2類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.3 Å 分子置換 / 解像度: 2.3 Å |
|---|
 データ登録者 データ登録者 | Nimma, S. / Gu, W. / Manik, M.K. / Ve, T. / Nanson, J.D. / Kobe, B. |
|---|
| 資金援助 |  オーストラリア, 2件 オーストラリア, 2件 | 組織 | 認可番号 | 国 |
|---|
| Australian Research Council (ARC) | FL180100109, FT200100572 |  オーストラリア オーストラリア | | National Health and Medical Research Council (NHMRC, Australia) | 1107804, 1160570, 1071659 |  オーストラリア オーストラリア |
|
|---|
 引用 引用 |  ジャーナル: Febs Lett. / 年: 2022 ジャーナル: Febs Lett. / 年: 2022
タイトル: Crystal structure of the Toll/interleukin-1 receptor (TIR) domain of IL-1R10 provides structural insights into TIR domain signalling.
著者: Nimma, S. / Gu, W. / Manik, M.K. / Ve, T. / Nanson, J.D. / Kobe, B. |
|---|
| 履歴 | | 登録 | 2021年11月28日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2022年2月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2022年4月20日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2023年10月18日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.3 Å
分子置換 / 解像度: 2.3 Å  データ登録者
データ登録者 オーストラリア, 2件
オーストラリア, 2件  引用
引用 ジャーナル: Febs Lett. / 年: 2022
ジャーナル: Febs Lett. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7szl.cif.gz
7szl.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7szl.ent.gz
pdb7szl.ent.gz PDB形式
PDB形式 7szl.json.gz
7szl.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/sz/7szl
https://data.pdbj.org/pub/pdb/validation_reports/sz/7szl ftp://data.pdbj.org/pub/pdb/validation_reports/sz/7szl
ftp://data.pdbj.org/pub/pdb/validation_reports/sz/7szl
 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: IL1RAPL2, IL1R9 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: IL1RAPL2, IL1R9 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX2 / 波長: 0.95372 Å
/ ビームライン: MX2 / 波長: 0.95372 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj






