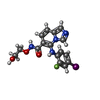
登録情報 データベース : EMDB / ID : EMD-48401タイトル Cryo-EM structure of CRAF/MEK1/14-3-3 complex (open monomer conformation, CRAF Y340D/Y341D mutant) 複合体 : CRAF/MEK1/14-3-3 complex複合体 : CRAFタンパク質・ペプチド : RAF proto-oncogene serine/threonine-protein kinase複合体 : MEK1タンパク質・ペプチド : Dual specificity mitogen-activated protein kinase kinase 1複合体 : 14-3-3 dimerタンパク質・ペプチド : 14-3-3 protein zetaリガンド : PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTERリガンド : MAGNESIUM IONリガンド : 5-[(2-fluoro-4-iodophenyl)amino]-N-(2-hydroxyethoxy)imidazo[1,5-a]pyridine-6-carboxamide / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / Spodoptera exigua (シロイチモジヨトウ)手法 / / 解像度 : 3.9 Å Jang DM / Jeon H / Eck MJ 資金援助 Organization Grant number 国 National Institutes of Health/National Cancer Institute (NIH/NCI) R35CA242461-06
ジャーナル : Nat Commun / 年 : 2025タイトル : Cryo-EM structures of CRAF/MEK1/14-3-3 complexes in autoinhibited and open-monomer states reveal features of RAF regulation.著者 : Dong Man Jang / Kayla Boxer / Byung Hak Ha / Emre Tkacik / Talya Levitz / Shaun Rawson / Rebecca J Metivier / Anna Schmoker / Hyesung Jeon / Michael J Eck / 要旨 : CRAF (RAF1) is one of three RAF-family kinases that initiate MAP kinase signaling in response to activated RAS and is essential for oncogenic signaling from mutant KRAS. Like BRAF, CRAF is regulated ... CRAF (RAF1) is one of three RAF-family kinases that initiate MAP kinase signaling in response to activated RAS and is essential for oncogenic signaling from mutant KRAS. Like BRAF, CRAF is regulated by 14-3-3 engagement and by intramolecular autoinhibitory interactions of its N-terminal regulatory region. Unlike BRAF, it is thought to require tyrosine phosphorylation in its N-terminal acidic (NtA) motif for full catalytic activation. Here we describe cryo-EM reconstructions of full-length CRAF in complex with MEK1 and a 14-3-3 dimer. These structures reveal a fully autoinhibited conformation analogous to that observed for BRAF and two "open monomer" states in which the inhibitory interactions of the CRD and 14-3-3 dimer are released or rearranged, but the kinase domain remains inactive. Structure-function studies of the NtA motif indicate that phosphorylation or acidic mutations in this segment increase catalytic activity by destabilizing the inactive conformation of the kinase domain. Collectively, these studies provide a structural foundation for understanding the shared and unique regulatory features of CRAF and will inform efforts to selectively block CRAF signaling in cancer. 履歴 登録 2024年12月20日 - ヘッダ(付随情報) 公開 2025年9月10日 - マップ公開 2025年9月10日 - 更新 2025年10月8日 - 現状 2025年10月8日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) /  Spodoptera exigua (シロイチモジヨトウ)
Spodoptera exigua (シロイチモジヨトウ) データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2025
ジャーナル: Nat Commun / 年: 2025
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_48401.map.gz
emd_48401.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-48401-v30.xml
emd-48401-v30.xml emd-48401.xml
emd-48401.xml EMDBヘッダ
EMDBヘッダ emd_48401_fsc.xml
emd_48401_fsc.xml FSCデータファイル
FSCデータファイル emd_48401.png
emd_48401.png emd_48401_msk_1.map
emd_48401_msk_1.map マスクマップ
マスクマップ emd-48401.cif.gz
emd-48401.cif.gz emd_48401_half_map_1.map.gz
emd_48401_half_map_1.map.gz emd_48401_half_map_2.map.gz
emd_48401_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-48401
http://ftp.pdbj.org/pub/emdb/structures/EMD-48401 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-48401
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-48401 emd_48401_validation.pdf.gz
emd_48401_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_48401_full_validation.pdf.gz
emd_48401_full_validation.pdf.gz emd_48401_validation.xml.gz
emd_48401_validation.xml.gz emd_48401_validation.cif.gz
emd_48401_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48401
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48401 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48401
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48401 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_48401.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_48401.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_48401_msk_1.map
emd_48401_msk_1.map 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー





















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)