[日本語] English
 万見
万見- EMDB-25903: Cryo-electron microscopy of Adeno-associated virus serotype 4 at 2.2 A -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron microscopy of Adeno-associated virus serotype 4 at 2.2 A | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAV4 / adeno-associated virus / serotype 4 / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | Phospholipase A2-like domain / Phospholipase A2-like domain / Parvovirus coat protein VP2 / Parvovirus coat protein VP1/VP2 / Parvovirus coat protein VP1/VP2 / Capsid/spike protein, ssDNA virus / T=1 icosahedral viral capsid / structural molecule activity / Capsid 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Adeno-associated virus - 4 (アデノ随伴ウイルス) / Adeno-associated virus - 4 (アデノ随伴ウイルス) /   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.21 Å | |||||||||
 データ登録者 データ登録者 | Zane GM / Silveria MA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2023 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2023タイトル: Cryo-EM structure of adeno-associated virus 4 at 2.2 Å resolution. 著者: Grant Zane / Mark Silveria / Nancy Meyer / Tommi White / Rui Duan / Xiaoqin Zou / Michael Chapman /  要旨: Adeno-associated virus (AAV) is the vector of choice for several approved gene-therapy treatments and is the basis for many ongoing clinical trials. Various strains of AAV exist (referred to as ...Adeno-associated virus (AAV) is the vector of choice for several approved gene-therapy treatments and is the basis for many ongoing clinical trials. Various strains of AAV exist (referred to as serotypes), each with their own transfection characteristics. Here, a high-resolution cryo-electron microscopy structure (2.2 Å) of AAV serotype 4 (AAV4) is presented. The receptor responsible for transduction of the AAV4 clade of AAV viruses (including AAV11, AAV12 and AAVrh32.33) is unknown. Other AAVs interact with the same cell receptor, adeno-associated virus receptor (AAVR), in one of two different ways. AAV5-like viruses interact exclusively with the polycystic kidney disease-like 1 (PKD1) domain of AAVR, while most other AAVs interact primarily with the PKD2 domain. A comparison of the present AAV4 structure with prior corresponding structures of AAV5, AAV2 and AAV1 in complex with AAVR provides a foundation for understanding why the AAV4-like clade is unable to interact with either PKD1 or PKD2 of AAVR. The conformation of the AAV4 capsid in variable regions I, III, IV and V on the viral surface appears to be sufficiently different from AAV2 to ablate binding with PKD2. Differences between AAV4 and AAV5 in variable region VII appear to be sufficient to exclude binding with PKD1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25903.map.gz emd_25903.map.gz | 240.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25903-v30.xml emd-25903-v30.xml emd-25903.xml emd-25903.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25903_fsc.xml emd_25903_fsc.xml | 16 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25903.png emd_25903.png | 250.3 KB | ||
| Filedesc metadata |  emd-25903.cif.gz emd-25903.cif.gz | 6.8 KB | ||
| その他 |  emd_25903_half_map_1.map.gz emd_25903_half_map_1.map.gz emd_25903_half_map_2.map.gz emd_25903_half_map_2.map.gz | 673.8 MB 673.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25903 http://ftp.pdbj.org/pub/emdb/structures/EMD-25903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25903 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25903 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25903_validation.pdf.gz emd_25903_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25903_full_validation.pdf.gz emd_25903_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_25903_validation.xml.gz emd_25903_validation.xml.gz | 28.3 KB | 表示 | |
| CIF形式データ |  emd_25903_validation.cif.gz emd_25903_validation.cif.gz | 36.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25903 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25903 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25903 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25903 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7thrMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25903.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25903.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.50799 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_25903_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
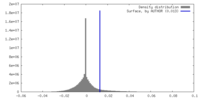
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_25903_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
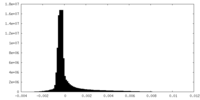
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus
| 全体 | 名称:   Adeno-associated virus (アデノ随伴ウイルス) Adeno-associated virus (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Adeno-associated virus
| 超分子 | 名称: Adeno-associated virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 / NCBI-ID: 272636 / 生物種: Adeno-associated virus / Sci species strain: Serotype 4 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 3.746 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 250.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid
| 分子 | 名称: Capsid / タイプ: protein_or_peptide / ID: 1 / 詳細: VP1 sequence of AAV4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Adeno-associated virus - 4 (アデノ随伴ウイルス) Adeno-associated virus - 4 (アデノ随伴ウイルス) |
| 分子量 | 理論値: 80.688023 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TTDGYLPDWL EDNLSEGVRE WWALQPGAPK PKANQQHQDN ARGLVLPGYK YLGPGNGLDK GEPVNAADAA ALEHDKAYDQ QLKAGDNPY LKYNHADAEF QQRLQGDTSF GGNLGRAVFQ AKKRVLEPLG LVEQAGETAP GKKRPLIESP QQPDSSTGIG K KGKQPAKK ...文字列: TTDGYLPDWL EDNLSEGVRE WWALQPGAPK PKANQQHQDN ARGLVLPGYK YLGPGNGLDK GEPVNAADAA ALEHDKAYDQ QLKAGDNPY LKYNHADAEF QQRLQGDTSF GGNLGRAVFQ AKKRVLEPLG LVEQAGETAP GKKRPLIESP QQPDSSTGIG K KGKQPAKK KLVFEDETGA GDGPPEGSTS GAMSDDSEMR AAAGGAAVEG GQGADGVGNA SGDWHCDSTW SEGHVTTTST RT WVLPTYN NHLYKRLGES LQSNTYNGFS TPWGYFDFNR FHCHFSPRDW QRLINNNWGM RPKAMRVKIF NIQVKEVTTS NGE TTVANN LTSTVQIFAD SSYELPYVMD AGQEGSLPPF PNDVFMVPQY GYCGLVTGNT SQQQTDRNAF YCLEYFPSQM LRTG NNFEI TYSFEKVPFH SMYAHSQSLD RLMNPLIDQY LWGLQSTTTG TTLNAGTATT NFTKLRPTNF SNFKKNWLPG PSIKQ QGFS KTANQNYKIP ATGSDSLIKY ETHSTLDGRW SALTPGPPMA TAGPADSKFS NSQLIFAGPK QNGNTATVPG TLIFTS EEE LAATNATDTD MWGNLPGGDQ SNSNLPTVDR LTALGAVPGM VWQNRDIYYQ GPIWAKIPHT DGHFHPSPLI GGFGLKH PP PQIFIKNTPV PANPATTFSS TPVNSFITQY STGQVSVQID WEIQKERSKR WNPEVQFTSN YGQQNSLLWA PDAAGKYT E PRAIGTRYLT HHL UniProtKB: Capsid |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 219 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.33 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: blot force: 4 time: 2 s. | |||||||||
| 詳細 | The sample was isolated by multiple rounds of cesium chloride density centrifugation and dialyzed into a HEPES-buffered, sodium chloride solution (see Meyer et al., 2019 Bioprotocols paper). |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4809 / 平均露光時間: 1.4 sec. / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 16500 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 211-734 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Link to CNS_RSRef software: https://chapman.missouri.edu/software/ |
| 得られたモデル |  PDB-7thr: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)