+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22281 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of human presequence protease in partial closed state 1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Partial open state / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Mitochondrial protein import / amyloid-beta complex / growth cone lamellipodium / cellular response to norepinephrine stimulus / growth cone filopodium / microglia development / collateral sprouting in absence of injury / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; その他のペプチターゼ / Formyl peptide receptors bind formyl peptides and many other ligands / axo-dendritic transport ...Mitochondrial protein import / amyloid-beta complex / growth cone lamellipodium / cellular response to norepinephrine stimulus / growth cone filopodium / microglia development / collateral sprouting in absence of injury / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; その他のペプチターゼ / Formyl peptide receptors bind formyl peptides and many other ligands / axo-dendritic transport / regulation of Wnt signaling pathway / regulation of synapse structure or activity / axon midline choice point recognition / astrocyte activation involved in immune response / NMDA selective glutamate receptor signaling pathway / regulation of spontaneous synaptic transmission / mating behavior / growth factor receptor binding / peptidase activator activity / protein targeting to mitochondrion / Golgi-associated vesicle / PTB domain binding / positive regulation of amyloid fibril formation / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / astrocyte projection / Lysosome Vesicle Biogenesis / neuron remodeling / Deregulated CDK5 triggers multiple neurodegenerative pathways in Alzheimer's disease models / nuclear envelope lumen / dendrite development / positive regulation of protein metabolic process / TRAF6 mediated NF-kB activation / signaling receptor activator activity / Advanced glycosylation endproduct receptor signaling / negative regulation of long-term synaptic potentiation / modulation of excitatory postsynaptic potential / The NLRP3 inflammasome / main axon / transition metal ion binding / regulation of multicellular organism growth / intracellular copper ion homeostasis / ECM proteoglycans / regulation of presynapse assembly / positive regulation of T cell migration / neuronal dense core vesicle / Purinergic signaling in leishmaniasis infection / positive regulation of chemokine production / cellular response to manganese ion / Notch signaling pathway / clathrin-coated pit / extracellular matrix organization / neuron projection maintenance / astrocyte activation / ionotropic glutamate receptor signaling pathway / Mitochondrial protein degradation / positive regulation of calcium-mediated signaling / positive regulation of mitotic cell cycle / axonogenesis / protein serine/threonine kinase binding / response to interleukin-1 / platelet alpha granule lumen / cellular response to copper ion / cellular response to cAMP / positive regulation of glycolytic process / central nervous system development / dendritic shaft / trans-Golgi network membrane / endosome lumen / positive regulation of interleukin-1 beta production / positive regulation of long-term synaptic potentiation / adult locomotory behavior / learning / positive regulation of JNK cascade / Post-translational protein phosphorylation / locomotory behavior / enzyme activator activity / serine-type endopeptidase inhibitor activity / microglial cell activation / positive regulation of non-canonical NF-kappaB signal transduction / TAK1-dependent IKK and NF-kappa-B activation / cellular response to nerve growth factor stimulus / regulation of long-term neuronal synaptic plasticity / recycling endosome / synapse organization / visual learning / protein processing / metalloendopeptidase activity / positive regulation of interleukin-6 production / response to lead ion / Golgi lumen / cognition / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / endocytosis / cellular response to amyloid-beta / neuron projection development / metallopeptidase activity / positive regulation of inflammatory response / positive regulation of tumor necrosis factor production / Platelet degranulation / heparin binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
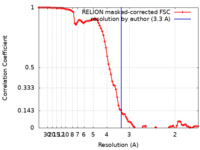
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Liang WG / Zhao M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for the mechanisms of human presequence protease conformational switch and substrate recognition. 著者: Wenguang G Liang / Juwina Wijaya / Hui Wei / Alex J Noble / Jordan M Mancl / Swansea Mo / David Lee / John V Lin King / Man Pan / Chang Liu / Carla M Koehler / Minglei Zhao / Clinton S Potter ...著者: Wenguang G Liang / Juwina Wijaya / Hui Wei / Alex J Noble / Jordan M Mancl / Swansea Mo / David Lee / John V Lin King / Man Pan / Chang Liu / Carla M Koehler / Minglei Zhao / Clinton S Potter / Bridget Carragher / Sheng Li / Wei-Jen Tang /  要旨: Presequence protease (PreP), a 117 kDa mitochondrial M16C metalloprotease vital for mitochondrial proteostasis, degrades presequence peptides cleaved off from nuclear-encoded proteins and other ...Presequence protease (PreP), a 117 kDa mitochondrial M16C metalloprotease vital for mitochondrial proteostasis, degrades presequence peptides cleaved off from nuclear-encoded proteins and other aggregation-prone peptides, such as amyloid β (Aβ). PreP structures have only been determined in a closed conformation; thus, the mechanisms of substrate binding and selectivity remain elusive. Here, we leverage advanced vitrification techniques to overcome the preferential denaturation of one of two ~55 kDa homologous domains of PreP caused by air-water interface adsorption. Thereby, we elucidate cryoEM structures of three apo-PreP open states along with Aβ- and citrate synthase presequence-bound PreP at 3.3-4.6 Å resolution. Together with integrative biophysical and pharmacological approaches, these structures reveal the key stages of the PreP catalytic cycle and how the binding of substrates or PreP inhibitor drives a rigid body motion of the protein for substrate binding and catalysis. Together, our studies provide key mechanistic insights into M16C metalloproteases for future therapeutic innovations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22281.map.gz emd_22281.map.gz | 49.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22281-v30.xml emd-22281-v30.xml emd-22281.xml emd-22281.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22281_fsc.xml emd_22281_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22281.png emd_22281.png | 164.8 KB | ||
| マスクデータ |  emd_22281_msk_1.map emd_22281_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22281.cif.gz emd-22281.cif.gz | 7.1 KB | ||
| その他 |  emd_22281_additional_1.map.gz emd_22281_additional_1.map.gz emd_22281_half_map_1.map.gz emd_22281_half_map_1.map.gz emd_22281_half_map_2.map.gz emd_22281_half_map_2.map.gz | 59.9 MB 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22281 http://ftp.pdbj.org/pub/emdb/structures/EMD-22281 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22281 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22281 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22281_validation.pdf.gz emd_22281_validation.pdf.gz | 958.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22281_full_validation.pdf.gz emd_22281_full_validation.pdf.gz | 957.9 KB | 表示 | |
| XML形式データ |  emd_22281_validation.xml.gz emd_22281_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  emd_22281_validation.cif.gz emd_22281_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22281 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22281 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22281 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22281 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6xovMC  6xosC  6xotC  6xouC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10937 (タイトル: CryoEM of PreP prepared via Chameleon / Data size: 2.2 TB / Data #1: Apo-PreP micrographs [micrographs - multiframe] / Data #2: PreP half particles [micrographs - single frame] EMPIAR-10937 (タイトル: CryoEM of PreP prepared via Chameleon / Data size: 2.2 TB / Data #1: Apo-PreP micrographs [micrographs - multiframe] / Data #2: PreP half particles [micrographs - single frame]Data #3: Citrate synthase presequence bound PreP [micrographs - multiframe] Data #4: Amyloid beta bound PreP [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22281.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22281.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22281_msk_1.map emd_22281_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_22281_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_22281_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_22281_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CryoEM map of Human Presequence Protease in partial close state 1
| 全体 | 名称: CryoEM map of Human Presequence Protease in partial close state 1 |
|---|---|
| 要素 |
|
-超分子 #1: CryoEM map of Human Presequence Protease in partial close state 1
| 超分子 | 名称: CryoEM map of Human Presequence Protease in partial close state 1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Presequence protease, mitochondrial
| 分子 | 名称: Presequence protease, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; その他のペプチターゼ |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 114.901461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHAAA CERALQYKLG DKIHGFTVNQ VTSVPELFLT AVKLTHDDTG ARYLHLARED TNNLFSVQFR TTPMDSTGVP HILEHTVLC GSQKYPCRDP FFKMLNRSLS TFMNAFTASD YTLYPFSTQN PKDFQNLLSV YLDATFFPCL RELDFWQEGW R LEHENPSD ...文字列: MHHHHHHAAA CERALQYKLG DKIHGFTVNQ VTSVPELFLT AVKLTHDDTG ARYLHLARED TNNLFSVQFR TTPMDSTGVP HILEHTVLC GSQKYPCRDP FFKMLNRSLS TFMNAFTASD YTLYPFSTQN PKDFQNLLSV YLDATFFPCL RELDFWQEGW R LEHENPSD PQTPLVFKGV VFNEMKGAFT DNERIFSQHL QNRLLPDHTY SVVSGGDPLC IPELTWEQLK QFHATHYHPS NA RFFTYGN FPLEQHLKQI HEEALSKFQK IEPSTVVPAQ TPWDKPREFQ ITCGPDSFAT DPSKQTTVSV SFLLPDITDT FEA FTLSLL SSLLTSGPNS PFYKALIESG LGTDFSPDVG YNGYTREAYF SVGLQGIVEK DIETVRSLID RTIDEVVEKG FEDD RIEAL LHKIEIQMKH QSTSFGLMLT SYIASCWNHD GDPVELLKLG NQLAKFRQCL QENPKFLQEK VKQYFKNNQH KLTLS MRPD DKYHEKQAQV EATKLKQKVE ALSPGDRQQI YEKGLELRSQ QSKPQDASCL PALKVSDIEP TIPVTELDVV LTAGDI PVQ YCAQPTNGMV YFRAFSSLNT LPEELRPYVP LFCSVLTKLG CGLLDYREQA QQIELKTGGM SASPHVLPDD SHMDTYE QG VLFSSLCLDR NLPDMMQLWS EIFNNPCFEE EEHFKVLVKM TAQELANGIP DSGHLYASIR AGRTLTPAGD LQETFSGM D QVRLMKRIAE MTDIKPILRK LPRIKKHLLN GDNMRCSVNA TPQQMPQTEK AVEDFLRSIG RSKKERRPVR PHTVEKPVP SSSGGDAHVP HGSQVIRKLV MEPTFKPWQM KTHFLMPFPV NYVGECIRTV PYTDPDHASL KILARLMTAK FLHTEIREKG GAYGGGAKL SHNGIFTLYS YRDPNTIETL QSFGKAVDWA KSGKFTQQDI DEAKLSVFST VDAPVAPSDK GMDHFLYGLS D EMKQAHRE QLFAVSHDKL LAVSDRYLGT GKSTHGLAIL GPENPKIAKD PSWIIR UniProtKB: Presequence protease, mitochondrial |
-分子 #2: Amyloid-beta precursor protein
| 分子 | 名称: Amyloid-beta precursor protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 4.335852 KDa |
| 配列 | 文字列: DAEFRHDSGY EVHHQKLVFF AEDVGSNKGA IIGLMVGGVV UniProtKB: Amyloid-beta precursor protein |
-分子 #3: Amyloid-beta precursor protein
| 分子 | 名称: Amyloid-beta precursor protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 273.33 Da |
| 配列 | 文字列: (UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.7 詳細: 20 mM Tris, pH 7.7, 150 mM NaCl, 10mM KCl, 20 mM EDTA and 1 mM 2-mercaptoethanol |
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 308 K / 装置: SPOTITON |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均露光時間: 6.0 sec. / 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)