-検索条件
-検索結果
検索 (著者・登録者: deng & b)の結果458件中、1から50件目までを表示しています

EMDB-40650: 
Phosphoinositide phosphate 3 kinase gamma

EMDB-40651: 
Phosphoinositide phosphate 3 kinase gamma bound with ATP

EMDB-40652: 
Phosphoinositide phosphate 3 kinase gamma bound with ADP

EMDB-40653: 
Phosphoinositide phosphate 3 kinase gamma bound with ADP and Gbetagamma

EMDB-40654: 
Phosphoinositide phosphate 3 kinase gamma bound with ADP and two Gbetagamma subunits in State 1

EMDB-40655: 
Phosphoinositide phosphate 3 kinase gamma bound with ADP and two Gbetagamma subunits in State 2
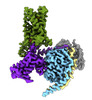
EMDB-43647: 
CryoEM structure of Gi-coupled TAS2R14 with cholesterol and an intracellular tastant
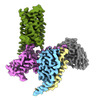
EMDB-43650: 
CryoEM structure of Ggust-coupled TAS2R14 with cholesterol and an intracellular tastant

EMDB-43656: 
CryoEM structure of Gi-coupled TAS2R14 with cholesterol and an intracellular tastant (Locally refined map)

EMDB-43657: 
CryoEM structure of Ggust-coupled TAS2R14 with cholesterol and an intracellular tastant (Locally refined map)

EMDB-43091: 
hGBP1 conformer on the bacterial outer membrane

EMDB-43153: 
hGBP1 conformer on the bacterial outer membrane

EMDB-35832: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

PDB-8iyx: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

EMDB-37516: 
BA.2(S375) Spike (S6P)/hACE2 complex

EMDB-37517: 
Local refinement of RBD-ACE2

PDB-8wgv: 
BA.2(S375) Spike (S6P)/hACE2 complex

PDB-8wgw: 
Local refinement of RBD-ACE2

EMDB-43542: 
ELIC5 with cysteamine in 2:1:1 POPC:POPE:POPG nanodisc in open conformation

PDB-8vuw: 
ELIC5 with cysteamine in 2:1:1 POPC:POPE:POPG nanodisc in open conformation

EMDB-36368: 
Cryo-EM structure of CCHFV envelope protein Gc trimer in complex with Gc13 Fab

EMDB-36406: 
CCHFV envelope protein Gc in complex with Gc8

EMDB-36407: 
CCHFV envelope protein Gc in complex with Gc13

PDB-8jkd: 
Cryo-EM structure of CCHFV envelope protein Gc trimer in complex with Gc13 Fab

PDB-8jlw: 
CCHFV envelope protein Gc in complex with Gc8

PDB-8jlx: 
CCHFV envelope protein Gc in complex with Gc13

EMDB-34552: 
Structure of dimeric mouse SCMC core complex

EMDB-34554: 
Structure of mouse SCMC bound with KH domain of FILIA

EMDB-34555: 
Structure of mouse SCMC bound with full-length FILIA

EMDB-34556: 
Structure of mouse SCMC core complex

PDB-8h93: 
Structure of dimeric mouse SCMC core complex

PDB-8h94: 
Structure of mouse SCMC bound with KH domain of FILIA

PDB-8h95: 
Structure of mouse SCMC bound with full-length FILIA

PDB-8h96: 
Structure of mouse SCMC core complex

EMDB-40270: 
Cryo-EM structure of GPR34-Gi complex

PDB-8sai: 
Cryo-EM structure of GPR34-Gi complex

EMDB-34954: 
Cryo-EM structure of SSX1 bound to the H2AK119Ub nucleosome at a resolution of 3.05 angstrom

EMDB-36747: 
Cryo-EM structure of 2 SSX1 bound to H2AK119Ub nucleosome at a resolution of 3.6 angstroms (2:1 complex)

PDB-8hqy: 
Cryo-EM structure of SSX1 bound to the H2AK119Ub nucleosome at a resolution of 3.05 angstrom

EMDB-36751: 
Outward_facing SLC15A4 monomer

EMDB-36752: 
Outward-facing SLC15A4 dimer

EMDB-36753: 
SLC15A4_TASL complex

PDB-8jzr: 
Outward_facing SLC15A4 monomer

PDB-8jzs: 
Outward-facing SLC15A4 dimer

PDB-8jzu: 
SLC15A4_TASL complex

EMDB-35347: 
Structure of R2 with 3'UTR and DNA in binding state

EMDB-35348: 
Structure of R2 with 3'UTR and DNA in unwinding state

EMDB-35349: 
Structure of R2 with 5'ORF

EMDB-35350: 
Structure of R2 with 5'ORF and 3'UTR

PDB-8ibw: 
Structure of R2 with 3'UTR and DNA in binding state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
