-検索条件
-検索結果
検索 (著者・登録者: zhang & dl)の結果182件中、1から50件目までを表示しています
EMDB-45078: 
E.coli GroEL + PBZ1587 inhibitor
EMDB-45079: 
E.coli GroEL apoenzyme
EMDB-45080: 
E.Faecium GroEL

EMDB-45098: 
E.coli GroEL + PBZ1587 inhibitor C1 reconstruction

PDB-9c0b: 
E.coli GroEL + PBZ1587 inhibitor

PDB-9c0c: 
E.coli GroEL apoenzyme

PDB-9c0d: 
E.Faecium GroEL
EMDB-40180: 
MsbA bound to cerastecin C

PDB-8gk7: 
MsbA bound to cerastecin C

EMDB-41140: 
APC/C-CDH1-UBE2C-Ubiquitin-CyclinB-NTD

EMDB-41142: 
APC/C-CDH1-UBE2C-UBE2S-Ubiquitin-CyclinB
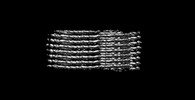
EMDB-40305: 
Cryo-EM structure of insulin amyloid-like fibril that is composed of two antiparallel protofilaments

EMDB-36241: 
Cryo-EM structure of mouse Piezo1-MDFIC complex (consensus map)

EMDB-36242: 
Cryo-EM structure of mouse Piezo1-MDFIC complex (Masked refinement of the cap domain)

EMDB-36243: 
Cryo-EM structure of mouse Piezo1-MDFIC complex (masked refinement of the transmembrane domain)

EMDB-36244: 
Cryo-EM structure of mouse Piezo1-MDFIC(C240A) complex

EMDB-35577: 
Cryo-EM structure of mouse Piezo1-MDFIC complex (composite map)
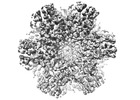
EMDB-16187: 
Wild tye immature Gag Structure

EMDB-16190: 
KAKA mutation immature Gag structure

EMDB-32952: 
Structure of the phosphorylation-site double mutant S431A/T432A of the KaiC circadian clock protein

EMDB-32953: 
Structure of the phosphorylation-site double mutant S431E/T432E of the KaiC circadian clock protein

EMDB-35522: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex(mask on receptor)

EMDB-35523: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex(mask on Giq-scFV16 complex)

EMDB-35524: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi1 complex(mask on receptor)

EMDB-35525: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi1 complex(mask on Gil-scFV16 complex)

EMDB-35529: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex (consensus map)

EMDB-35533: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi complex(consensus map)

EMDB-35356: 
Cryo-EM structure of the 9-hydroxystearic acid bound GPR120-Gi complex

EMDB-35357: 
Cryo-EM structure of the linoleic acid bound GPR120-Gi complex

EMDB-35358: 
Cryo-EM structure of the oleic acid bound GPR120-Gi complex

EMDB-35359: 
Cryo-EM structure of the TUG891 bound GPR120-Gi complex

EMDB-35360: 
Cryo-EM structure of the eicosapentaenoic acid bound GPR120-Gi complex

EMDB-29736: 
Cryo-EM structure of the TUG891 bound GPR120-Giq complex

EMDB-28881: 
Cryo-EM structure of a Zinc-loaded wild-type YiiP-Fab complex

EMDB-28882: 
Cryo-EM structure of a Zinc-loaded D51A mutant of the YiiP-Fab complex

EMDB-28883: 
Cryo-EM structure of a Zinc-loaded asymmetrical TMD D70A mutant of the YiiP-Fab complex

EMDB-28884: 
Cryo-EM structure of a Zinc-loaded symmetrical D70A mutant of the YiiP-Fab complex

EMDB-28885: 
Cryo-EM structure of a Zinc-loaded D287A mutant of the YiiP-Fab complex

EMDB-28886: 
Cryo-EM structure of a Zinc-loaded H263A/D287A mutant of the YiiP-Fab complex

EMDB-33196: 
Structure of human inner kinetochore CCAN-DNA complex

EMDB-33197: 
Structure of human inner kinetochore CCAN complex

EMDB-31394: 
Cytochrome c-type biogenesis protein CcmABCD from E. coli

EMDB-31395: 
Cytochrome c-type biogenesis protein CcmABCD from E. coli in complex with ANP
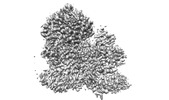
EMDB-31396: 
Cytochrome c-type biogenesis protein CcmABCD from E. coli in complex with Heme and ATP.

EMDB-31956: 
Cytochrome c-type biogenesis protein CcmABCD

EMDB-31957: 
Cytochrome c-type biogenesis protein CcmABCD from E. coli in complex with heme and single ATP

EMDB-25929: 
SAN27-14 bound to a antigenic site V on prefusion-stabilized hMPV F

EMDB-25982: 
Cryo-EM structure of hMPV preF bound by Fabs MPE8 and SAN32-2

PDB-7tjq: 
SAN27-14 bound to a antigenic site V on prefusion-stabilized hMPV F

PDB-7tl0: 
Cryo-EM structure of hMPV preF bound by Fabs MPE8 and SAN32-2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
